Python第三方库之MedPy
Posted Albert Darren
tags:
篇首语:本文由小常识网(cha138.com)小编为大家整理,主要介绍了Python第三方库之MedPy相关的知识,希望对你有一定的参考价值。
文章目录
- 1.MedPy简介
- 2.MedPy安装
- 3.MedPy常用函数
- 3.1 `medpy.io.load(image)`
- 3.2 `medpy.metric.binary.dc(result, reference)`
- 3.3 `medpy.metric.binary.jc(result, reference)`
- 3.4 `medpy.metric.binary.hd(result,reference,voxelspacing=None,connectivity=1,)`
- 3.5 `medpy.metric.binary.hd95(result,reference,voxelspacing=None,connectivity=1,)`
- 3.6 `medpy.metric.binary.recall(result, reference)`
- 3.7 `medpy.metric.binary.sensitivity(result, reference)`
- 3.8`medpy.metric.binary.true_positive_rate(result, reference)`
- 3.9`medpy.metric.binary.specificity(result, reference)`
- 3.10`medpy.metric.binary.true_negative_rate(result, reference)`
- 3.11`medpy.metric.binary.precision(result, reference)`
- 3.12 `medpy.metric.binary.positive_predictive_value(result, reference)`
- 3.13 `medpy.io.save(arr, filename, hdr=False, force=True, use_compression=False)`
- 4.参考文献
1.MedPy简介
MedPy 是一个图像处理库和针对医学(如高维)图像处理的脚本集合,此处主要讨论利用该库计算常见的医学图像分割任务评价指标,如Dice、Jaccard、Hausdorff Distance、Sensitivity、Specificity、Positive predictive value等。
论文表格的表头一般使用评价指标的英文全称首字母大写简写,如PPV代表阳性预测值,故此处也给出。
-
Dice相似系数(Dice Similarity Coefficient,DSC)或Dice系数(Dice Coefficient,DC)
也称为索伦森-骰子系数是用于衡量两个样本相似性的统计量。它是由植物学家Thorvald Sørensen和Lee Raymond Dice独立开发的,他们分别于1948年和1945年发表。是最常用的一项评价指标,用于有效衡量分割算法预测标签与真实标注标签的空间重叠程度,其对应值越大表示分割精度越高。该指标最初应用于离散数据,给定两个集合A 和B,则定义为两个集合共有的元素数除以每个集合中元素数之和的两倍
D S C = 2 ∣ A ∩ B ∣ ∣ A ∣ + ∣ B ∣ DSC=\\frac2|A\\cap B||A|+|B| DSC=∣A∣+∣B∣2∣A∩B∣
其中, ∣ A ∣ |A| ∣A∣和 ∣ B ∣ |B| ∣B∣是两个集合的基数(即每个集合中的元素数)当应用于布尔数据时,使用真阳性 (TP)、假阳性(FP) 和假阴性(FN) 的定义,它可以写为
D S C = 2 T P 2 T P + F P + F N D S C=\\frac2 T P2 T P+F P+F N DSC=2TP+FP+FN2TP -
Jaccard相似系数(Jaccard Similarity Coefficient,JSC)或Jaccard系数(Jaccard Coefficient,JC)
与 Dice 相似系数指标相似,也是一种衡量两幅图像相似程度的指标,其由实际分割结果与真实标签的交集同二者并集的比值得出,反映了分割方法的准确程度,其值越高,分割结果越准确。设TP表示正确预测为正例的数量,FP表示错误预测为正例的数量,FN表示错误预测为反例的数量,则定义为
Jaccard = T P T P + F P + F N \\text Jaccard=\\fracT PT P+F P+F N Jaccard=TP+FP+FNTP
-
豪斯多夫距离(Hausdorff distance,HD)
表示预测分割区域边界与真实肿瘤区域边界之间的最大距离,其值越小代表脑肿瘤边界分割误差越小、质量越好。设 X 和 Y 是度量空间 ( M , d ) (M,d) (M,d)的两个非空子集,则定义他们的Hausdorff distance d H ( X , Y ) d_H(X,Y) dH(X,Y)为
d H ( X , Y ) = max d X Y , d Y X = max max x ∈ X min y ∈ Y d ( x , y ) , max y ∈ Y min x ∈ X d ( x , y ) d_H(X, Y)=\\max \\left\\d_X Y, d_Y X\\right\\=\\max \\left\\\\max _x \\in X \\min_y \\in Yd(x, y), \\max _y \\in Y \\min _x \\in X d(x, y)\\right\\ dH(X,Y)=maxdXY,dYX=maxx∈Xmaxy∈Ymind(x,y),y∈Ymaxx∈Xmind(x,y)
计算绿色曲线 X 和蓝色曲线 Y 之间的豪斯多夫距离的分量过程如下图所示。首先,对点集X中的每一个点 x x x计算其到点集Y中的每一个点 y y y的距离,保留最短距离 inf x ∈ X d ( x , y ) \\inf_x \\in Xd(x,y) infx∈Xd(x,y),然后找出保留的最短距离中的最大距离 sup y ∈ Y inf x ∈ X d ( x , y ) \\sup_y \\in Y\\inf_x \\in Xd(x,y) supy∈Yinfx∈Xd(x,y)记为 d X Y d_XY dXY。然后,对点集Y中的每一个点 y y y计算其到点集X中的每一个点 x x x的距离,保留最短距离 inf y ∈ Y d ( x , y ) \\inf_y \\in Yd(x,y) infy∈Yd(x,y),然后找出保留的最短距离中的最大距离 sup x ∈ X inf y ∈ Y d ( x , y ) \\sup_x \\in X\\inf_y \\in Yd(x,y) supx∈Xinfy∈Yd(x,y)记为 d Y X d_YX dYX。最后,取 d X Y d_XY dXY和 d Y X d_YX dYX最大值作为点集X和Y之间的豪斯多夫距离。

-
95% 豪斯多夫距离(95% Hausdorff distance,HD95)
为了排除一些离群点造成的不合理距离,保持整体数值稳定性,一般选择从小到大排名前 95%的距离作为实际豪斯多夫距离,称之为 95% 豪斯多夫距离。
-
灵敏度(Sensitivity)
也称真阳性率(True Positive Rate)、命中率(hit rate)和召回率(Recall),表示预测为正的肿瘤标签占真实肿瘤标签的比例,其值越大,漏检率越低。设P表示数据中真阳样例的数量,TP表示正确预测为正例的数量,FN表示错误预测为反例的数量,则定义为
S e n s i t i v i t y = T P R = R e c a l l = T P P = T P T P + F N = 1 − F N R \\mathrmSensitivity=\\mathrmTPR=\\mathrmRecall=\\frac\\mathrmTP\\mathrmP=\\frac\\mathrmTP\\mathrmTP+\\mathrmFN=1-\\mathrmFNR Sensitivity=TPR=Recall=PTP=TP+FNTP=1−FNR -
特异度(Specificity)
也称真阴性率(True Negative Rate)和选择性(Selectivity),表示预测为正的背景标签占所有真实背景标签的比例,其值越高则误诊率越低。设N表示数据中真阴样例的数量,TN表示正确预测为负例的数量,FP表示错误预测为正例的数量,则定义为
S p e c i f i c i t y = S e l e c t i v i t y = T N R = T N N = T N T N + F P = 1 − F P R \\mathrmSpecificity=\\mathrmSelectivity=\\mathrmTNR=\\frac\\mathrmTN\\mathrmN=\\frac\\mathrmTN\\mathrmTN+\\mathrmFP=1-\\mathrmFPR Specificity=Selectivity=TNR=NTN=TN+FPTN=1−FPR -
阳性预测值(Positive Predictive Value,PPV)
也称精确度(Precision),指在所有样本的预测结果中,真阳性占所有阳性样本的比例,其值越大越好。设TP表示正确预测为正例的数量,FP表示错误预测为正例的数量,则定义为
P r e c i s i o n = P P V = T P T P + F P = 1 − F D R \\mathrmPrecision=\\mathrmPPV=\\frac\\mathrmTP\\mathrmTP+\\mathrmFP=1-\\mathrmFDR Precision=PPV=TP+FPTP=1−FDR
2.MedPy安装
medpy安装较为简单,此处以Ubuntu 18.04.5 LTS系统的root用户为例,其他情况详见参考文献安装即可。
为了启用graph-cut包,我们需要安装下列软件包
sudo apt-get install libboost-python-dev build-essential
然后使用阿里云镜像源高速安装medpy
sudo pip install medpy -i http://mirrors.aliyun.com/pypi/simple/ --trusted-host mirrors.aliyun.com
3.MedPy常用函数
3.1 medpy.io.load(image)
加载图像并返回包含图像像素内容以及头部对象的 ndarray
import os
from medpy.io import load,save
# 文件夹路径和文件名
dirname="/dataset/RSNA_ASNR_MICCAI_BraTS2021_TrainingData_16July2021/BraTS2021_00000/"
basename="BraTS2021_00000_seg.nii.gz"
full_path=os.path.join(dirname,basename)
# 加载脑肿瘤标记图像返回图像数据数组和头部信息
image_data, image_header = load(full_path)
print("图像数据类型为,图像数据形状为".format(type(image_data),image_data.shape))

# 获取一个切片数组
slice_0=image_data[:,:,77]
Python第三方库之openpyxl(3)
区域图
区域图类似于折线图,绘图线下面的区域会被填充,通过将分组设置为“standard”、“stacked”或“percentStacked”,可以获得不同的变体;“standard”是默认的。
2D区域图
from openpyxl import Workbook
from openpyxl.chart import (
AreaChart,
Reference,
Series,
)
wb = Workbook()
ws = wb.active
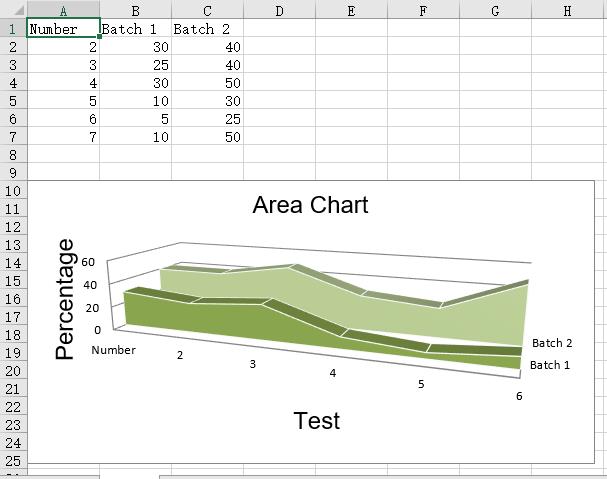
rows = [
[\'Number\', \'Batch 1\', \'Batch 2\'],
[2, 40, 30],
[3, 40, 25],
[4, 50, 30],
[5, 30, 10],
[6, 25, 5],
[7, 50, 10],
]
for row in rows:
ws.append(row)
chart = AreaChart()
chart.title = "Area Chart"
chart.style = 13
chart.x_axis.title = \'Test\'
chart.y_axis.title = \'Percentage\'
cats = Reference(ws, min_col=1, min_row=1, max_row=7)
data = Reference(ws, min_col=2, min_row=1, max_col=3, max_row=7)
chart.add_data(data, titles_from_data=True)
chart.set_categories(cats)
ws.add_chart(chart, "A10")
wb.save("area.xlsx")
运行结果:
3D区域图
from openpyxl import Workbook
from openpyxl.chart import (
AreaChart3D,
Reference,
Series,
)
wb = Workbook()
ws = wb.active
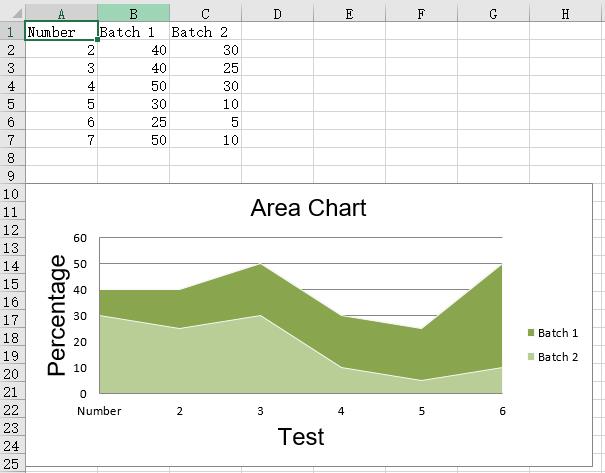
rows = [
[\'Number\', \'Batch 1\', \'Batch 2\'],
[2, 30, 40],
[3, 25, 40],
[4 ,30, 50],
[5 ,10, 30],
[6, 5, 25],
[7 ,10, 50],
]
for row in rows:
ws.append(row)
chart = AreaChart3D()
chart.title = "Area Chart"
chart.style = 13
chart.x_axis.title = \'Test\'
chart.y_axis.title = \'Percentage\'
chart.legend = None
cats = Reference(ws, min_col=1, min_row=1, max_row=7)
data = Reference(ws, min_col=2, min_row=1, max_col=3, max_row=7)
chart.add_data(data, titles_from_data=True)
chart.set_categories(cats)
ws.add_chart(chart, "A10")
wb.save("area3D.xlsx")
运行结果