r语言randomforest包下载不了
Posted
tags:
篇首语:本文由小常识网(cha138.com)小编为大家整理,主要介绍了r语言randomforest包下载不了相关的知识,希望对你有一定的参考价值。
原因:1.可能因为lib没有制定文件夹,导致R不知道下载哪里;2.可能因为源用不了(一个是没有在global option里面选择,一个是因为开了vpn)解决:
#.libPaths("C:/Users/yeziguniang/Documents/R/Rpakage")##创一个环境让这个包下载到这个路径下面,同时关闭vpn,选择好源
#install.packages("pheatmap")
#library("pheatmap")
成功! 参考技术A r语言randomforest包下载不了:你可以在R中装一个devtools,然后安装githubinstall包,以后下载包的时候遇到装不了的你就直接去github上面下载安装,这样再也不会出现有些包安装不了了. 参考技术B 原因:
1.可能因为lib没有制定文件夹,导致R不知道下载哪里;
2.可能因为源用不了(一个是没有在global option里面选择,一个是因为开了vpn)
解决:
#.libPaths("C:/Users/yeziguniang/Documents/R/Rpakage")##创一个环境让这个包下载到这个路径下面,同时关闭vpn,选择好源
#install.packages("pheatmap")
#library("pheatmap")
成功! 参考技术C 由于RStudio的源是Global,所以在使用install命令去安装包的时候可能会出现数据部分丢失的情况。
R语言 | randomForest包的随机森林回归模型以及对重要变量的选择

文献中使用随机森林回归的一个实例
首先来看一篇与植物根系微生物组有关研究,将文中涉及到随机森林回归的部分内容节选出来以帮助大家理解。
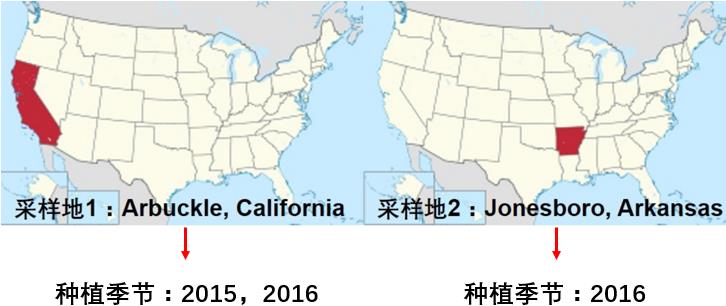
Edwards等(2018)分析了来自两个地区(加利福尼亚州、阿肯色州)以及三个连续种植季(2014、2015、2016)的水稻根系微生物组,意在阐述水稻根系微生物群落的时间动态特征,并解释驱动微生物群落组建的成因。文中部分内容使用到随机森林的回归模型建立微生物丰度和水稻生长时期的关系,用以识别重要的时间响应微生物类群。
作者首先分析了水稻根系微生物群落的组成结构,显示其受到地理区域(土壤环境)、植物生长时期以及植物根系不同部位的多重影响。随后分析各个因素的相对效应,发现水稻生长过程中能够逐渐对其根系菌群进行选择,植物生长时期的效应越来越明显,根系菌群朝向一组保守的物种组成趋势发展,同时地理环境效应的比重逐渐减弱。主要体现在:
(1)不同地理区域种植的水稻,随着水稻生长时期的推移,根际和根内菌群的相似度逐渐增加,并在后期趋于稳定,与种植季节无关:
(2)对于水稻根际和根内菌群,地区间独特OTU的相对丰度均随时间推移而不断下降,剩余的OTU中,两地区间共有OTU的比重上升。
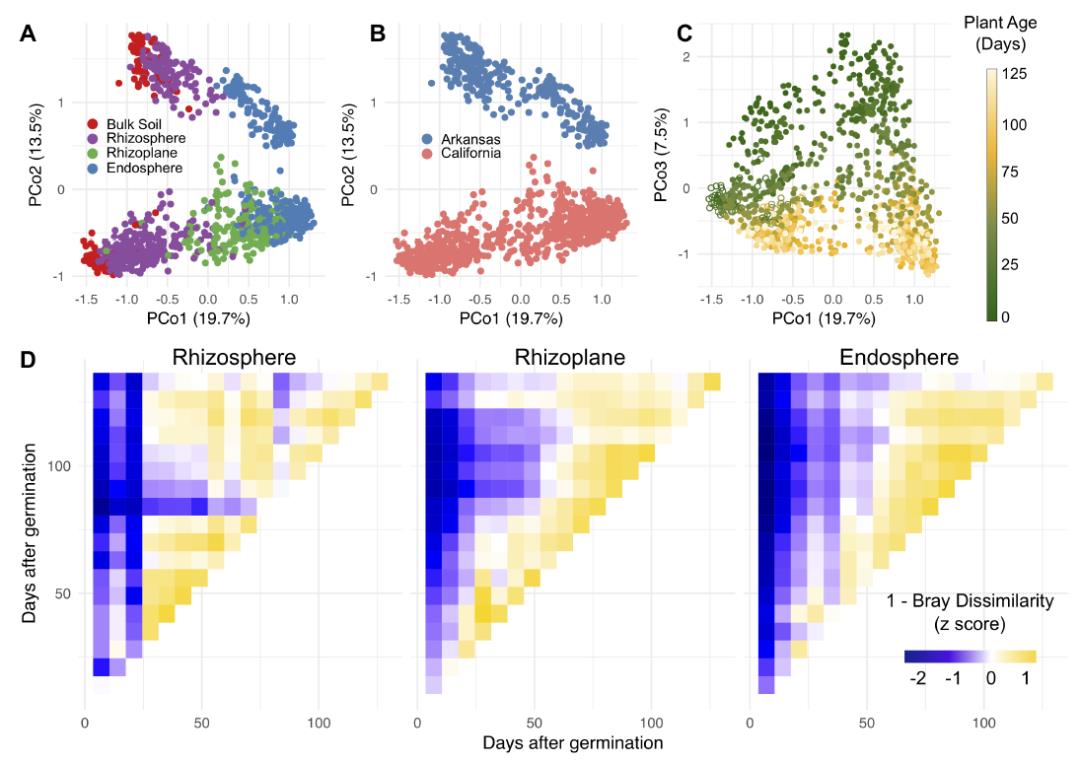
图A-C,水稻根系微生物群落的PCoA,分别突出了群落组成相似度与根系区域(A)、地理区域(B)或植物生长时间(C)的关系。
图D,根区、根际和根内样本中,微生物群落相似度随植物生长的关系。
总之存在一类具有显著时间特征的微生物类群,它们的丰度变化与水稻的生长时期密切相关,无论是增加(根系富集)或减少(根系排斥)。随后,作者期望寻找可用于区分水稻生长时期的微生物OTU组合,包括根际和根内菌群,就使用了随机森林。
作者选择了一部分的样本(加利福尼亚州2014年和阿肯色州2016年的部分数据)用作训练集,通过随机森林分别将根际和根内所有微生物OTU的相对丰度与水稻生长时期进行了回归,并根据各个OTU对模型预测准确性的相对贡献按重要性进行排名。十折交叉验证用于评估模型中OTU数量与模型误差的关系,并按重要性排名对OTU进行选择,删除了大部分不重要OTU后,最终保留的OTU用于构建最终的随机森林回归模型。
结果中,共确定了85个最重要的与水稻生长时期密切关联的OTU。训练集的数据显示,通过这85个OTU构建的随机森林回归模型分别解释了根际和根内菌群对植物年龄有关方差的91.5%和88.4%,表明它们的丰度组合能够准确预测水稻的生长时期。此外,测试集以及验证集数据(涉及了训练集样本外的其它采样时间、地理区域的样本)也一致地表明,通过这85个OTU构建的随机森林回归模型同样准确预测了水稻的生长时期,凸显了水稻根际和根内存在特定的微生物类群,在跨地理区域和季节的水稻植物的生命周期中表现出一致的模式。
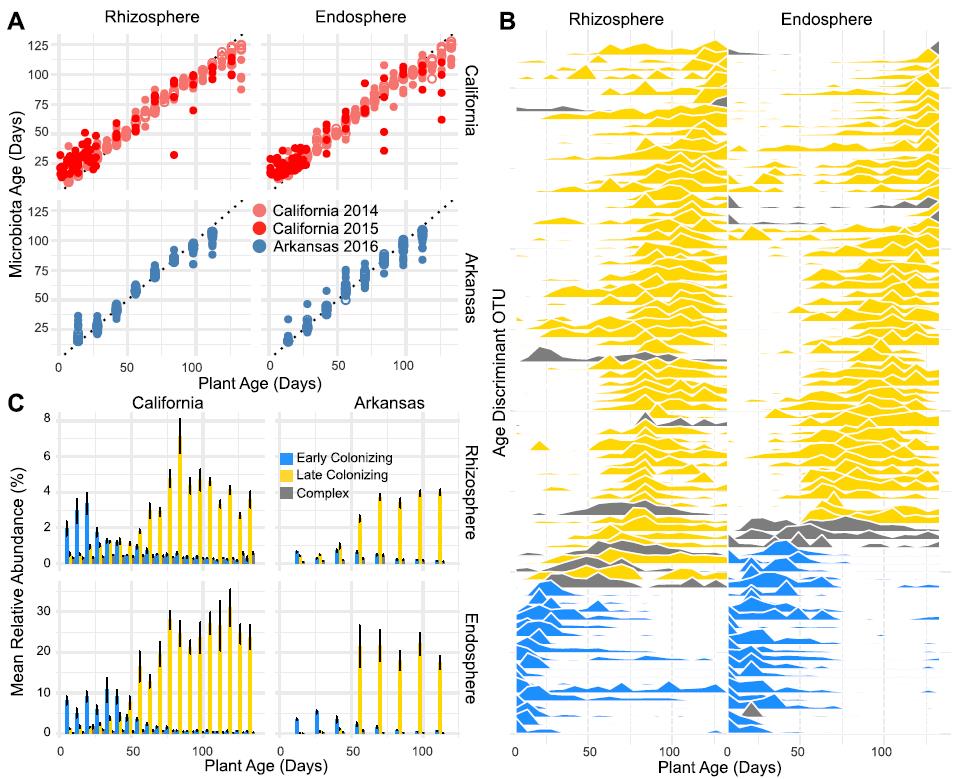
图A,横坐标是水稻的实际生长时期,纵坐标是随机森林模型通过根际或根内微生物丰度预测的水稻生长时期,模型具有很高的精度。
图B,重要的85个OTU在水稻根际或根内的丰度与时间的关系。
图C,85个OTU根据丰度的时间响应模式,可分为早期定植(丰度早期很高,但随时间降低)或晚期定植(丰度早期很低,但随时间增加)以及复合型(早期晚期定植区分不明显)类群,柱形图展示了3种类群中OTU平均丰度变化趋势。
文中后续还有内容涉及到随机森林模型的应用,这里就不再展示了。节选的部分章节想必也足以能够帮助大家了解这个方法的实际应用了。
此外,该文献的补充材料中提供了有关OTU丰度表、分析过程的R代码等非常全面的数据,大家若有兴趣参考原文即可。
接下来,展示随机森林回归及对重要变量选择在R语言中的实现方法。
通过R包randomForest的随机森林执行回归
对于随机森林应用在类别型响应变量中的分类功能,前文“”中,已经以R包randomForest中的方法为例展示了如何通过随机森林构建分类预测模型(分类模型的训练和测试),以及筛选对区分已知分类具有高度鉴别模式的变量组合(评估变量的相对重要性)。
在下文中,将响应变量更换为连续型变量,继续展示R包randomForest的随机森林回归方法以及实现对重要变量的选择。其实无论执行的分类还是回归,过程中使用的方法都是一样的,R函数还是那些R函数,区别只在于对部分函数的参数设置以及结果的解读方式上。
下文的测试数据,R代码等的百度盘链接(提取码,fljz):
https://pan.baidu.com/s/1Lfk21hGrWDehodWchBiIIg
若百度盘失效,也可在GitHub的备份中获取:
https://github.com/lyao222lll/sheng-xin-xiao-bai-yu
示例数据
植物根系菌群结构与植物生长密切相关,到目前为止,已经在许多研究中都有报道了,这已经是个共识。因此,下文的示例数据也同样来自某植物根际区域细菌群落组成的16S扩增子测序数据,类似地,仿照上文文献中的过程,通过随机森林建立微生物与植物生长时期的响应关系,根据微生物丰度推断植物生长时期,并寻找一组重要的时间特征类群。
示例数据“otu_table.txt”中,共记录了45个连续生长时间中植物根际土壤样本中细菌OTU的相对丰度信息。
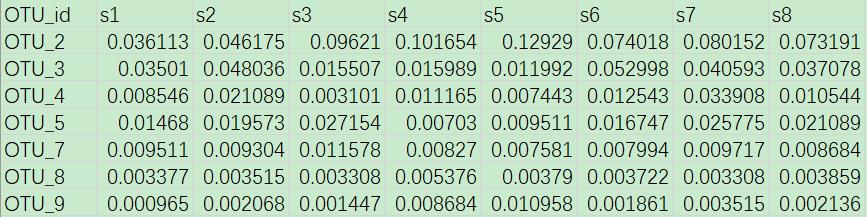
“plant_age.txt”中,记录了这45个根际土壤样本对应的植物生长时间,时间单位是天。
随机森林回归模型的初步构建
将OTU丰度数据读入R中,可以事先作一些预处理,例如剔除低丰度类群等。
随后,对应OTU丰度样本与植物生长时间的关系,加载randomForest包并运行随机森林。
##数据预处理
#读取 OTUs 丰度表
otu <- read.delim('otu_table.txt', row.names = 1)
#过滤低丰度 OTUs 类群,它们对分类贡献度低,且影响计算效率
#例如剔除总丰度低于 0.05% 的值
otu <- otu[which(rowSums(otu) >= 0.0005), ]
#合并有关于植物生长时间的信息
plant <- read.delim('plant_age.txt', row.names = 1)
otu <- data.frame(t(otu))
otu <- otu[rownames(plant), ]
otu <- cbind(otu, plant)
#为了方便后续评估随机森林模型的性能
#将总数据集分为训练集(占 70%)和测试集(占 30%)
set.seed(123)
train <- sample(nrow(otu), nrow(otu)*0.7)
otu_train <- otu[train, ]
otu_test <- otu[-train, ]
##randomForest 包的随机森林
library(randomForest)
#随机森林计算(默认生成 500 棵决策树),详情 ?randomForest
set.seed(123)
otu_train.forest <- randomForest(plant_age~., data = otu_train, importance = TRUE)
otu_train.forest
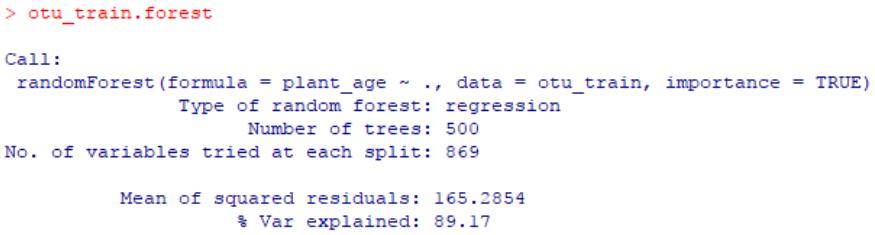
结果中,% Var explained体现了预测变量(用于回归的所有OTU)对响应变量(植物年龄)有关方差的整体解释率。在本示例中,剔除了低丰度的OTU后,剩余的OTU(约2600个左右)解释了约89.17%的总方差,可以理解为该回归的R2=0.8917,相当可观的一个数值,表明该植物根际细菌的群落结构随植物生长密切相关。
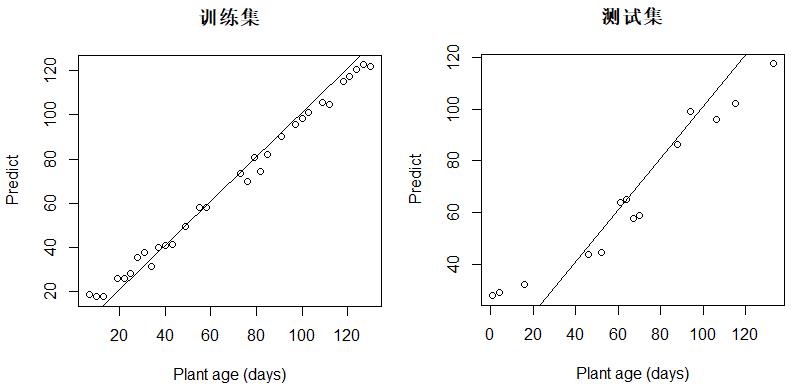
查看该模型的预测性能,可以看到具有较高的精度。
#使用训练集,查看预测精度
plant_predict <- predict(otu_train.forest, otu_train)
plot(otu_train$plant_age, plant_predict, main = '训练集',
xlab = 'Plant age (days)', ylab = 'Predict')
abline(1, 1)
#使用测试集,评估预测性能
plant_predict <- predict(otu_train.forest, otu_test)
plot(otu_test$plant_age, plant_predict, main = '测试集',
xlab = 'Plant age (days)', ylab = 'Predict')
abline(1, 1)
重要的预测变量选择
但是,并非这所有的2600余个OTU都对回归的精度具有可观的贡献。有些OTU丰度的时间特征并不明显,可能在回归中产生较大噪声,对模型精度带来较高的误差。因此,最好将低贡献的OTU去除。
基于已经构建好的随机森林回归模型,可以从中评估OTU的重要性。将OTU按重要程度高低进行排名后,选择排名靠前的一部分OTU,这些重要的OTU是明显的与植物生长时间密切关联的一些细菌类群。
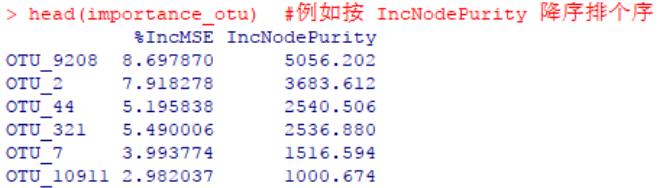
##OTU 的重要性评估
#查看表示每个预测变量(细菌 OTU)重要性的得分
#summary(otu_train.forest)
importance_otu <- otu_train.forest$importance
head(importance_otu)
#或者使用函数 importance()
importance_otu <- data.frame(importance(otu_train.forest), check.names = FALSE)
head(importance_otu)
#作图展示 top30 重要的 OTUs
varImpPlot(otu_train.forest, n.var = min(30, nrow(otu_train.forest$importance)),
main = 'Top 30 - variable importance')
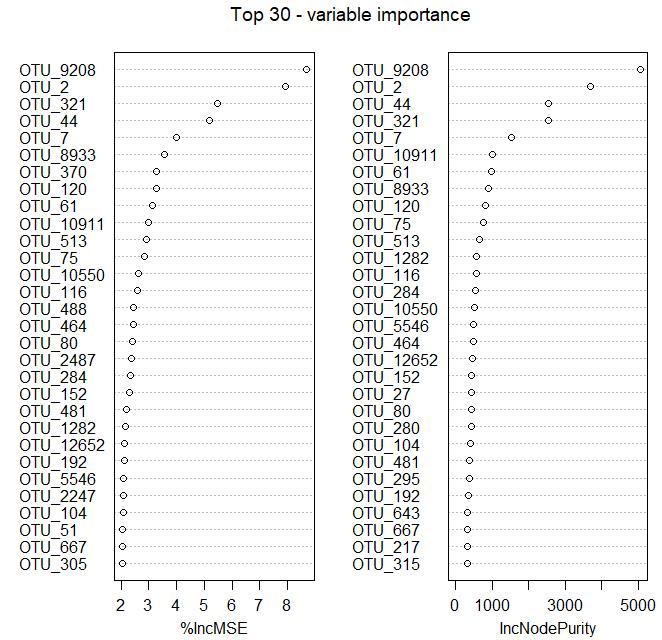
“%IncMSE”即increase in mean squared error,通过对每一个预测变量随机赋值,如果该预测变量更为重要,那么其值被随机替换后模型预测的误差会增大。因此,该值越大表示该变量的重要性越大;
“IncNodePurity”即increase in node purity,通过残差平方和来度量,代表了每个变量对分类树每个节点上观测值的异质性的影响,从而比较变量的重要性。该值越大表示该变量的重要性越大。
对于“%IncMSE”或“IncNodePurity”,二选一作为判断预测变量重要性的指标。需注意的是,二者的排名存在一定的差异。
#可以根据某种重要性的高低排个序,例如根据“IncNodePurity”指标
importance_otu <- importance_otu[order(importance_otu$IncNodePurity, decreasing = TRUE), ]
head(importance_otu)
#输出表格
#write.table(importance_otu, 'importance_otu.txt', sep = ' ', col.names = NA, quote = FALSE)
交叉验证
那么,最终选择多少重要的预测变量(本示例为OTUs)是更合适的呢?
可通过执行十折交叉验证,根据交叉验证曲线对OTU进行取舍。交叉验证法的作用就是尝试利用不同的训练集/验证集划分来对模型做多组不同的训练/验证,来应对单独测试结果过于片面以及训练数据不足的问题。此处使用训练集本身进行交叉验证。
##交叉验证辅助评估选择特定数量的 OTU
#5 次重复十折交叉验证
set.seed(123)
otu_train.cv <- replicate(5, rfcv(otu_train[-ncol(otu_train)], otu_train$plant_age, cv.fold = 10, step = 1.5), simplify = FALSE)
otu_train.cv
#提取验证结果绘图
otu_train.cv <- data.frame(sapply(otu_train.cv, '[[', 'error.cv'))
otu_train.cv$otus <- rownames(otu_train.cv)
otu_train.cv <- reshape2::melt(otu_train.cv, id = 'otus')
otu_train.cv$otus <- as.numeric(as.character(otu_train.cv$otus))
otu_train.cv.mean <- aggregate(otu_train.cv$value, by = list(otu_train.cv$otus), FUN = mean)
head(otu_train.cv.mean, 10)
#拟合线图
library(ggplot2)
ggplot(otu_train.cv.mean, aes(Group.1, x)) +
geom_line() +
theme(panel.grid = element_blank(), panel.background = element_rect(color = 'black', fill = 'transparent')) +
labs(title = '',x = 'Number of OTUs', y = 'Cross-validation error')
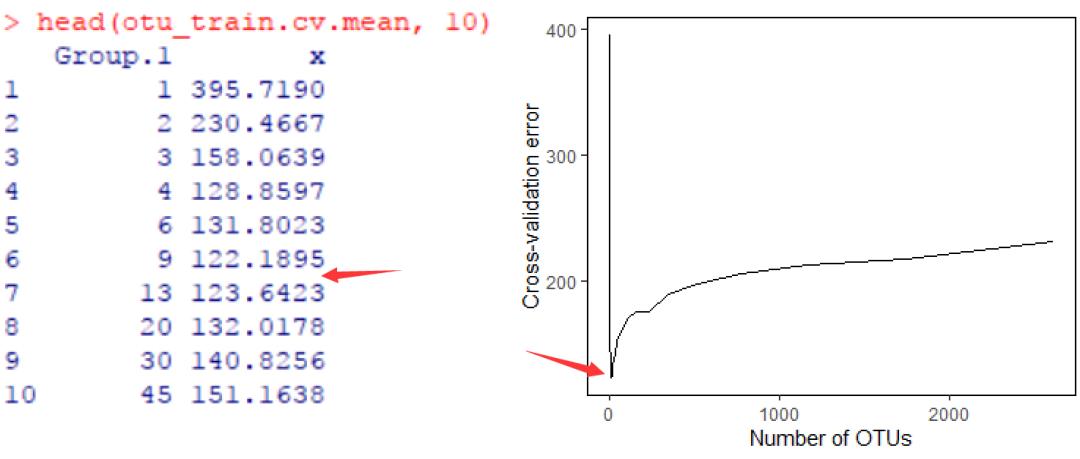
交叉验证曲线展示了模型误差与用于拟合的OTU数量之间的关系。误差首先会随OTUs数量的增加而减少,开始时下降非常明显,但到了特定范围处,下降幅度将不再有显著变化,甚至有所增加。
根据交叉验证曲线,提示保留9-13个重要的OTU即可以获得理想的回归结果,因为此时的误差达到最小。
因此,根据计算得到的各OUT重要性的值(如“IncNodePurity”),将OTU由高往低排序后,最后大约选择前9-13个OTU就可以了。
#提示保留 9-13 个重要的 OTU,可以使随机森林回归的精度最大化
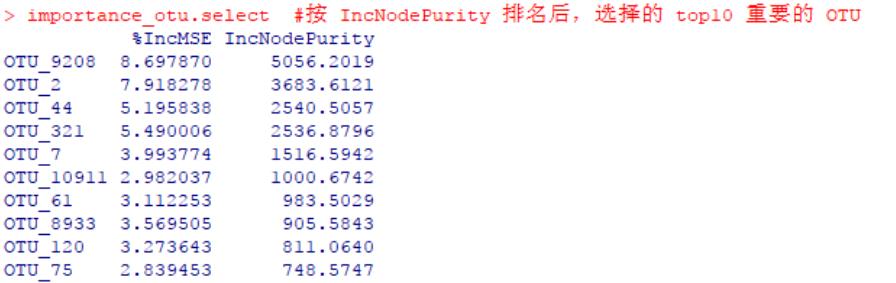
#首先根据某种重要性的高低排个序,例如根据“IncNodePurity”指标
importance_otu <- importance_otu[order(importance_otu$IncNodePurity, decreasing = TRUE), ]
#然后取出排名靠前的 OTU,例如 top10 最重要的 OTU
importance_otu.select <- importance_otu[1:10, ]
importance_otu.select
#输出表格
#write.table(importance_otu.select, 'importance_otu.select.txt', sep = ' ', col.names = NA, quote = FALSE)
#有关 OTU 的物种分类信息等其它细节,可后续自行补充
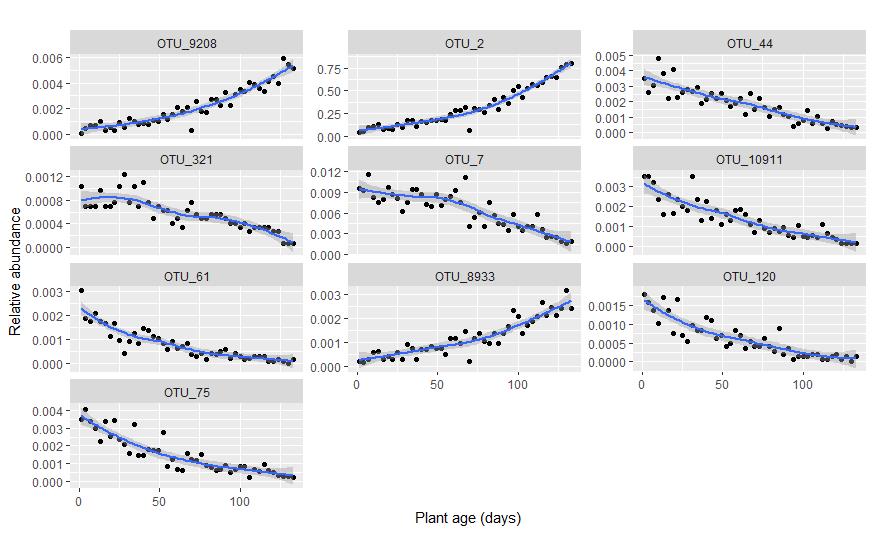
#不妨简单查看下这些重要的 OTU 丰度与植物生长时间的关系
#可以看到趋势非常明显,包括根际富集或排斥等都有涉及
otu_id.select <- rownames(importance_otu.select)
otu.select <- otu[ ,c(otu_id.select, 'plant_age')]
otu.select <- reshape2::melt(otu.select, id = 'plant_age')
ggplot(otu.select, aes(x = plant_age, y = value)) +
geom_point() +
geom_smooth() +
facet_wrap(~variable, ncol = 3, scale = 'free_y') +
labs(title = '',x = 'Plant age (days)', y = 'Relative abundance')
最终模型的确定
现在来看,只使用选择的10个重要的OTU构建回归模型,效果如何。
##只包含 10 个重要预测变量的简约回归
otu.select <- otu[ ,c(otu_id.select, 'plant_age')]
#为了方便后续评估随机森林模型的性能,将总数据集分为训练集(占 70%)和测试集(占 30%)
set.seed(123)
train <- sample(nrow(otu.select), nrow(otu.select)*0.7)
otu_train.select <- otu.select[train, ]
otu_test.select <- otu.select[-train, ]
#随机森林计算(默认生成 500 棵决策树),详情 ?randomForest
set.seed(123)
otu_train.select.forest <- randomForest(plant_age~., data = otu_train.select, importance = TRUE)
otu_train.select.forest
#使用训练集,查看预测精度
plant_predict <- predict(otu_train.select.forest, otu_train.select)
plot(otu_train.select$plant_age, plant_predict, main = '训练集',
xlab = 'Plant age (days)', ylab = 'Predict')
abline(1, 1)
#使用测试集,评估预测性能
plant_predict <- predict(otu_train.select.forest, otu_test.select)
plot(otu_test.select$plant_age, plant_predict, main = '测试集',
xlab = 'Plant age (days)', ylab = 'Predict')
abline(1, 1)
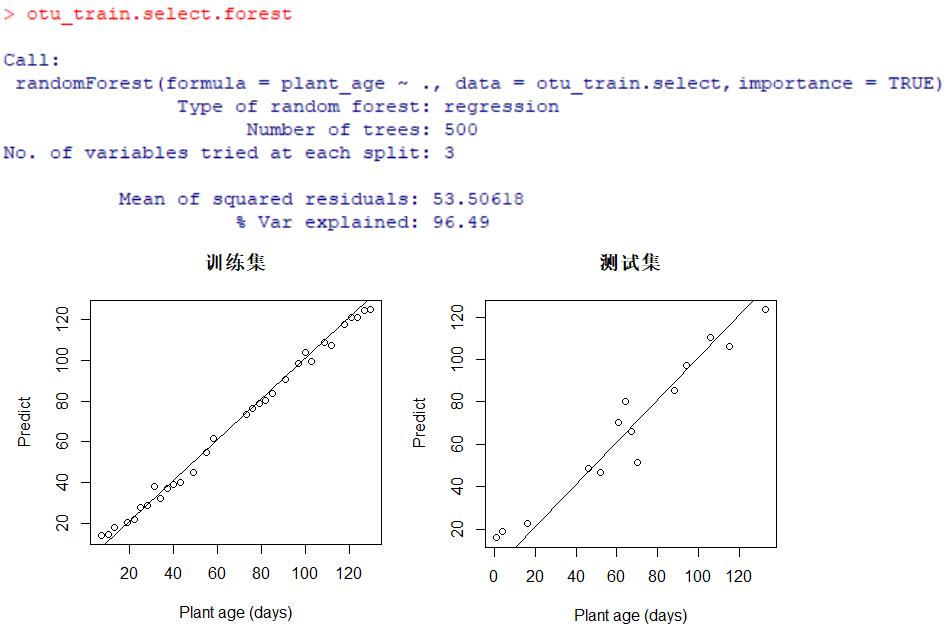
结果显示,与先前使用全部的OTU(去除低丰度后约2600多个)相比,只使用这10个更重要OTU的随机森林回归模型,获得了更出色的效果。
一是体现在% Var explained,模型中预测变量(这10个重要的OTU)对响应变量(植物年龄)有关方差的整体解释率达到了96.49%,大于先前的89.17%,这得益于排除了不重要或者高噪声的OTU。二是预测性能更佳,特别是对于测试集,与先前的分布图相比较(见上文),植物实际生长时间与通过根际菌群预测的植物年龄更趋一致。
如果不涉及预测,只是评估变量的重要性
上文过程中,为了评估随机森林回归模型的预测性能,将数据拆分为训练集和测试集分开讨论。但如果目的不涉及预测,只是为了寻找重要的预测变量,完全可以直接使用所有数据代入回归,无需再区分训练集与测试集,这也是很多文献中经常提到的方法。
例如另一篇类似的研究,Zhang等(2018)寻找水稻根系中与植物生长期密切相关的biomarkers时,同样使用的随机森林回归。但区别是文中没有涉及预测,关注的重点在于识别重要的微生物类群。
参考文献
以上是关于r语言randomforest包下载不了的主要内容,如果未能解决你的问题,请参考以下文章