几种常见的归一化方法
Posted 不想敲代码的小杨
tags:
篇首语:本文由小常识网(cha138.com)小编为大家整理,主要介绍了几种常见的归一化方法相关的知识,希望对你有一定的参考价值。
数据归一化是深度学习数据预处理中非常关键的步骤,可以起到统一量纲,防止小数据被吞噬的作用。
一:归一化的概念
归一化就是把所有数据都转化成[0,1]或者[-1,1]之间的数,其目的是为了取消各维数据之间的数量级差别,避免因为输入输出数据数量级差别大而造成网络预测误差过大。
二:归一化的作用
1)为了后面数据处理的方便,归一化可以避免一些不必要的数值问题。
2)为了程序运行时收敛速度更快
3)统一量纲。样本数据的评价标准不一样,需要对其量纲化,统一评价标准,这算是应用层面的需求。
4)避免神经元饱和。就是说当神经元的激活在接近0或者1时,在这些区域,梯度几乎为0,这样在反向传播过程中,局部梯度就会接近于0,这样非常不利于网络的训练。
5)保证输出数据中数值小的不被吞食。
三:归一化的类型
1:线性归一化
线性归一化也被称为最小-最大规范化;离散标准化,是对原始数据的线性变换,将数据值映射到[0,1]之间。用公式表示为:
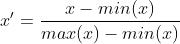
差标准化保留了原来数据中存在的关系,是消除量纲和数据取值范围影响的最简单的方法。代码实现如下:
def MaxMinNormalization(x,Max,Min):
x = (x - Min) / (Max - Min);
return x适用范围:比较适用在数值比较集中的情况
缺点:1)如果max和min不稳定,很容易使得归一化的结果不稳定,使得后续使用效果也不稳定。如果遇到超过目前属性[min,max]取值范围的时候,会引起系统报错。需要重新确定min和max。
2)如果数值集中的某个数值很大,则规范化后各值接近于0,并且将会相差不大。(如 1,1.2,1.3,1.4,1.5,1.6,10)这组数据。
2:零-均值归一化(Z-score标准化)
Z-score标准化也被称为标准差标准化,经过处理的数据的均值为0,标准差为1。其转化公式为:
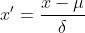
其中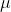 为原始数据的均值,
为原始数据的均值,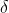 为原始数据的标准差,是当前用的最多的标准化公式
为原始数据的标准差,是当前用的最多的标准化公式
这种方法给予原始数据的均值(mean)和标准差(standard deviation)进行数据的标准化。经过处理的数据符合标准正态分布,即均值为0,标准差为1,这里的关键在于复合标准正态分布
代码实现如下:
def Z_ScoreNormalization(x,mu,sigma):
x = (x - mu) / sigma;
return x3:小数定标规范化
这种方法通过移动属性值的小数数位,将属性值映射到[-1,1]之间,移动的小数位数取决于属性值绝对值的最大值。转换公式为:
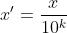
4:非线性归一化
这个方法包括log,指数,正切
适用范围:经常用在数据分析比较大的场景,有些数值很大,有些很小,将原始值进行映射。
四:批归一化(BatchNormalization)
1:引入
在以往的神经网络训练时,仅仅只对输入层数据进行归一化处理,却没有在中间层进行归一化处理。虽然我们对输入数据进行了归一化处理,但是输入数据经过了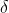 这样的矩阵乘法之后,其数据分布很可能发生很大改变,并且随着网络的层数不断加深。数据分布的变化将越来越大。因此这种在神经网络中间层进行的归一化处理,使得训练效果更好的方法就被称为批归一化(BN)
这样的矩阵乘法之后,其数据分布很可能发生很大改变,并且随着网络的层数不断加深。数据分布的变化将越来越大。因此这种在神经网络中间层进行的归一化处理,使得训练效果更好的方法就被称为批归一化(BN)
2:BN算法的优点
1)减少了人为选择参数
2)减少了对学习率的要求,我们可以使用初始状态下很大的学习率或者当使用较小的学习率时,算法也能够快速训练收敛。
3)破换了原来的数据分布,一定程度上缓解了过拟合(防止每批训练中某一个样本经常被挑选到)
4)减少梯度消失,加快收敛速度,提高训练精度。
3:批归一化(BN)算法流程
输入:上一层输出结果X=x1,x2,.....xm,学习参数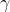 ,
,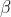
算法流程:
1)计算上一层输出数据的均值:

其中,m是此次训练样本batch的大小。
2)计算上一层输出数据的标准差:

3)归一化处理得到
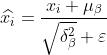
公式中的 是为了避免分母为0而加进去接近于0的很小的值。
是为了避免分母为0而加进去接近于0的很小的值。
4)重构,对经过上面归一化处理得到的数据进行重构,得到:
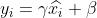
其中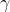 ,
,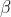 为可学习的参数。
为可学习的参数。
详细理解可参考:深度学习基础之归一化
图像的归一化互信息Normlized Mutual Information
紧接上文: 计算二维离散随机变量的联合概率分布
我们知道了上文提到的几种计算二维概率密度分布中, accumarray 方法是最快的.
那么就使用accumarray来求计算两幅相同大小图像的归一化互信息.
一. 互信息的定义
离散变量的互信息定义为:

求联合分布和边缘分布会用到了上文的方法.
或者使用熵来定义:
其中,
H
是熵. 熵是测量信号或者图像中信息量大小的量. 常用定义式:
归一化互信息定义为:
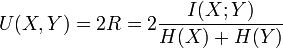
所以, 不论是求互信息还是求归一化互信息, 都要把两个随机变量的联合分布和边缘分布求出来.
而边缘分布可以从联合分布求出来. 所以只要求出来联合分布就可以了. 这正是我们上文的主题!
二. 实现
matlab里log函数是自然对数
求互信息和归一化互信息的函数, 在子桥的cnblogs 基础上修改:
function [MI,NMI] = NormMutualInfo( A, B ,method)
% NMI Normalized mutual information
% http://en.wikipedia.org/wiki/Mutual_information
% http://nlp.stanford.edu/IR-book/html/htmledition/evaluation-of-clustering-1.html
% % Example :
% % (http://nlp.stanford.edu/IR-book/html/htmledition/evaluation-of-clustering-1.html)
% % A = [1 1 1 1 1 1 2 2 2 2 2 2 3 3 3 3 3];
% % B = [1 2 1 1 1 1 1 2 2 2 2 3 1 1 3 3 3];
% % nmi(A,B)
%
% % ans = 0.3646
switch method
case 1
% 参考http://www.cnblogs.com/ziqiao/archive/2011/12/13/2286273.html#3350670
if length( A ) ~= length( B)
error('length( A ) must == length( B)');
end
total = length(A);
A_ids = unique(A);
B_ids = unique(B);
% Mutual information
MI = 0;
for idA = A_ids
for idB = B_ids
idAOccur = find( A == idA );
idBOccur = find( B == idB );
idABOccur = intersect(idAOccur,idBOccur);
px = length(idAOccur)/total;
py = length(idBOccur)/total;
pxy = length(idABOccur)/total;
MI = MI + pxy*log2(pxy/(px*py)+eps); % eps : the smallest positive number
end
end
% Normalized Mutual information
Hx = 0; % Entropies
for idA = A_ids
idAOccurCount = length( find( A == idA ) );
Hx = Hx - (idAOccurCount/total) * log2(idAOccurCount/total + eps);
end
Hy = 0; % Entropies
for idB = B_ids
idBOccurCount = length( find( B == idB ) );
Hy = Hy - (idBOccurCount/total) * log2(idBOccurCount/total + eps);
end
NMI = 2 * MI / (Hx+Hy);
case 2
% 使用accumarray方法
A=A(:);
B=B(:);
if length(A)~=length(B)
error('A B must be the SAME length!\\n');
end
H=accumarray([A B],ones(1,size(A,1)));
Pab=H/length(A);
pa=sum(Pab,2);
pb=sum(Pab,1);
Pa=repmat(pa,1,size(Pab,2));
Pb=repmat(pb,size(Pab,1),1);
MI=sum(sum(Pab.*log2((Pab+eps)./(Pa.*Pb+eps)+eps)));
Ha=-sum(pa.*log2(pa+eps));% 熵
Hb=-sum(pb.*log2(pb+eps));
NMI=2*MI/(Ha+Hb);
end
end
测试脚本:
A = randi(256,1,1e3);
B = randi(256,1,1e3);
tic
[mi1,nmi1]=NormMutualInfo(A,B,1)
toc
tic
[mi2,nmi2]=NormMutualInfo(A,B,2)
toc输出:
mi1 =
5.6394
nmi1 =
0.7231
Elapsed time is 5.087573 seconds.
mi2 =
5.6394
nmi2 =
0.7231
Elapsed time is 0.006722 seconds.结果相同.
多试几次, 基本上method2 比method1快了1000多倍!
三. 求归一化互信息矩阵
类似matlab中的normxcorr2函数, 如果输入两个矩阵的大小
(M∗N)
和
(m∗n)
)不同, 那么输出一个NMI矩阵.
为了节省不必要的计算. NMI矩阵大小为
(M−m+1,N−n+1)
NMI(1,1)代表两个矩阵左上角(1,1)元素对齐求出的NMI. NMI(end,end)代表两个矩阵右下角对齐.
method1, 为了进一步节省计算量, template矩阵的边缘分布和熵事先求出.
method2, 直接使用相同大小的矩阵调用accumarray.
matlab函数
function [ MI,NMI] = MI_matrix( A,B,L,method )
switch method
case 1
% 求NMI矩阵, 事先求出template边缘分布Pb和熵Hb
[M,N]=size(A);
[m,n]=size(B);
pb=hist(B(:),1:L)/(m*n);
Hb=-sum(pb.*log(pb+eps));
Pb=repmat(pb,L,1);
MI=nan(M-m+1,N-n+1);
NMI=nan(M-m+1,N-n+1);
for i=1:M-m+1
for j=1:N-n+1
ImgSub=A(i:i+m-1,j:j+n-1);
H=accumarray([ImgSub(:) B(:)],ones(1,m*n));
Pab=H/(m*n);
pa=sum(Pab,2);
Pa=repmat(pa,1,size(Pab,2));
MI(i,j)=sum(sum(Pab.*log2((Pab+eps)./(Pa.*Pb+eps)+eps)));
Ha=-sum(pa.*log2(pa+eps));% 熵
NMI(i,j)=2*MI(i,j)/(Ha+Hb);
end
end
case 2
% 求NMI矩阵
[M,N]=size(A);
[m,n]=size(B);
MI=nan(M-m+1,N-n+1);
NMI=nan(M-m+1,N-n+1);
for i=1:M-m+1
for j=1:N-n+1
ImgSub=A(i:i+m-1,j:j+n-1);
H=accumarray([ImgSub(:) B(:)],ones(1,m*n));
Pab=H/(m*n);
pa=sum(Pab,2);
pb=sum(Pab,1);
Pa=repmat(pa,1,size(Pab,2));
Pb=repmat(pb,size(Pab,1),1);
MI(i,j)=sum(sum(Pab.*log2((Pab+eps)./(Pa.*Pb+eps)+eps)));
Ha=-sum(pa.*log2(pa+eps));% 熵
Hb=-sum(pb.*log2(pb+eps));% 熵
NMI(i,j)=2*MI(i,j)/(Ha+Hb);
end
end
end测试代码:
A = randi(256,256,256);%256*256大小
B = randi(256,200,200);%200*200大小
tic
[ MI1,NMI1] = MI_matrix( A,B,256,1);
toc
tic
[ MI2,NMI2] = MI_matrix( A,B,256,2);
toc
all(abs(MI1(:)-MI2(:))<1e-5)
all(abs(NMI1(:)-NMI2(:))<1e-5)输出:
Elapsed time is 7.564679 seconds.
Elapsed time is 8.167062 seconds.
ans =
1
ans =
1实验表明, A和B大小相差越大, method1节省时间的优势越大.
如果A和B大小相差不大, method2更快一些.
以上是关于几种常见的归一化方法的主要内容,如果未能解决你的问题,请参考以下文章

