openVINO+paddleCPU部署新冠肺炎CT图像分类识别与病害分割
Posted 翼达口香糖
tags:
篇首语:本文由小常识网(cha138.com)小编为大家整理,主要介绍了openVINO+paddleCPU部署新冠肺炎CT图像分类识别与病害分割相关的知识,希望对你有一定的参考价值。
【openVINO+paddle】CPU部署新冠肺炎CT图像分类识别与病害分割
在这个项目中是我在看到一位大佬代码生成器的项目文章时想要尝试开发的一个项目。主要是想要在飞桨上通过Cla与Seg(分类和分割)模型对CT图像进行处理,然后将他们导出onnx模型下载到自己的设备上,通过openVINO转化为IR模型后,能够在CPU上就能够实现对新冠肺炎CT图片进行处理。
这里我会提供所有的数据和已经跑通的代码,我已经把我的源代码和相关数据资料全部上传到百度aistudio上,你可以直接在下面的链接搜索到:
https://aistudio.baidu.com/aistudio/projectdetail/3460633
因为考虑到OpenVINO这部分比较简洁,这篇文章先展示CPU OpenVINO上的效果,然后再展示如何在飞桨上进行模型和导出。
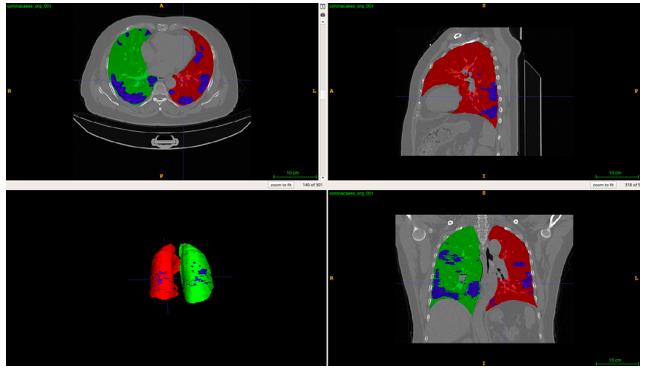
先看OpenVINO的效果图:
OpenVINO进行推理
这里直接提供IR模型,下面会教你在飞桨训练如何导出ONNX模型并转化为IR模型,你可以在这个链接下载到模型链接
你只需要下载下来放到和自己的jupyter notebook上即可。
首先你需要引入所需要的库,这里涉及到Open VINO的notebook安装,这个可以参考我的另一篇博客查看OpenVINO的notebook以及环境配置。这里可以先看这个代码,不算太难。
首先import所需要的库
import os
import sys
import zipfile
from pathlib import Path
from openvino.inference_engine import IECore
sys.path.append("../utils")
from models.custom_segmentation import SegmentationModel
from notebook_utils import benchmark_model, download_file, show_live_inference
这里如果已经下载好链接中的模型,你就将这个模型的路径配置到双引号中。将IR_PATH设置为"pretrained_model/unet44.xml"。
MODEL_PATH = "pretrained_model/quantized_unet_kits19.xml"
我们需要调用好所需的硬件,不仅可以使用CPU,还可以使用GPU等。
ie = IECore()
device = "MULTI:CPU,GPU" if "GPU" in ie.available_devices else "CPU"
为了测量模型推理性能,这里使用了OpenVINO的推理性能测量工具Benchmark Tool。你可以直接在note book上用命令! benchmark_app or %sx benchmark_app来启动。这里我们直接使用Notebook Utils中的包装器函数。
benchmark_model(model_path=MODEL_PATH, device=device, seconds=15)
下载和准备数据
这里的数据集直接提供了一个链接来下载我们的数据集。注意这里不是训练,而是简单地下载一个小小的训练集,因为这里如果路径下没有就可以直接下载。
BASEDIR = Path("kits19_frames_1")
CASE = 117
case_path = BASEDIR / f"case_CASE:05d"
if not case_path.exists():
filename = download_file(
f"https://storage.openvinotoolkit.org/data/test_data/openvino_notebooks/kits19/case_CASE:05d.zip"
)
with zipfile.ZipFile(filename, "r") as zip_ref:
zip_ref.extractall(path=BASEDIR)
os.remove(filename) # remove zipfile
print(f"Downloaded and extracted data for case_CASE:05d")
else:
print(f"Data for case_CASE:05d exists")
显示生活推理
为了在笔记本上显示实时推理,我们使用了OpenVINO推理引擎的异步处理的特点。(推理有多种方式,异步同步可以见我的博客那个OPENVINO的课程下)
我们使用Notebook Utils中的show_live_inference函数来显示实时推理的参数。这个函数使用Open Model Zoo的AsyncPipeline和Model API来执行异步推理。当对指定CT扫描的推理完成后,在结果图上打出包括预处理和显示在内的总时间和吞吐量(fps)。
ie = IECore()
segmentation_model = SegmentationModel(ie=ie, model_path=Path(MODEL_PATH), sigmoid=True)
image_paths = sorted(case_path.glob("imaging_frames/*jpg"))
print(f"case_path.name, len(image_paths) images")
进行推理
这里我们运行show live_inference函数,该函数将图像分割加载到指定的设备、加载图像、执行推理,并实时地在图像中加载的帧上显示结果。
device = "MULTI:CPU,GPU" if "GPU" in ie.available_devices else "CPU"
show_live_inference(
ie=ie, image_paths=image_paths, model=segmentation_model, device=device
)
如果中间有一些不会的地方可以看下这期间参考的一些大佬项目与技术文档。
- https://aistudio.baidu.com/aistudio/projectdetail/3459413
- https://aistudio.baidu.com/aistudio/projectdetail/3460443?forkThirdPart=1
- https://aistudio.baidu.com/aistudio/projectdetail/3460337?contributionType=1
- https://aistudio.baidu.com/aistudio/projectdetail/3461846
- https://aistudio.baidu.com/aistudio/projectdetail/3460268?forkThirdPart=1
- https://aistudio.baidu.com/aistudio/projectdetail/3460317?contributionType=1
数据集获取
这个就是用来分类的图片,下面这张图片是新冠肺炎患者的肺部CT图,第二张图片时正常人体的CT图片,下部有很明显的不同。
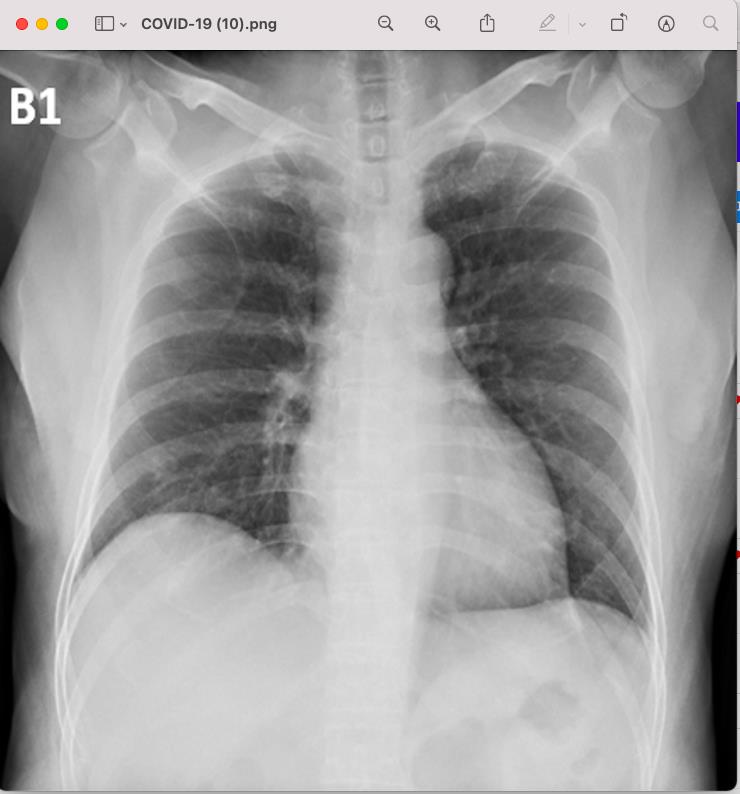
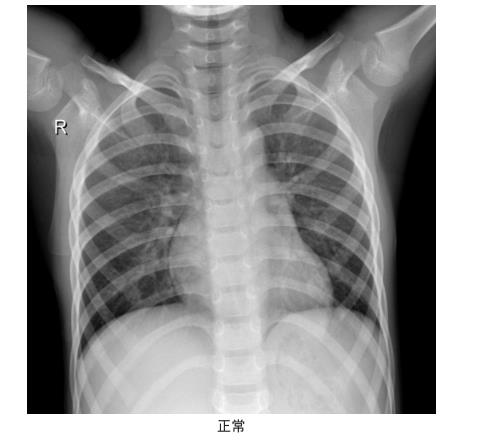
使用的数据集是 covid19-radiography-database。下面是数据的链接,COVID-19 RADIOGRAPHY DATABASE组合了意大利,Ieee8023和40余篇论文中的CT扫描,形成了一个有219张新冠病例,1341张正常扫描和1345张肺炎扫描的数据集,点开数据集直接引用。
https://aistudio.baidu.com/aistudio/datasetdetail/34241
数据自带的医学免责声明: 97% 仅为实验数据集上的结果,任何临床使用的算法需要在实际使用环境下进行实验,本模型分类结果不可作为临床诊疗依据。
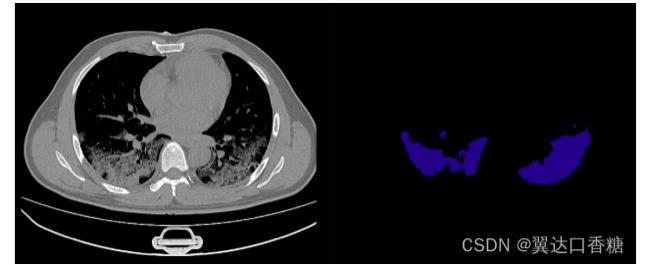
这里用的数据集是 covid19-ct-scans,数据集包含Ieee8023收集的20组新冠扫描,并对其进行了左右肺和感染区的标注。下面是标注的示例,
下面是数据的链接,只需要直接下载或者直接引用到自己aistudio中即可。
https://aistudio.baidu.com/aistudio/datasetdetail/34221
数据的医学免责声明: 任何临床使用的算法需要在实际使用环境下进行测试,本模型结果不可作为临床诊疗依据。
paddlecla-新冠CT分类模型训练与导出
1、数据集预处理
运行下面的代码解压数据集,一定要注意一点是,在解压后会得到三个txt文件在data目录下,是和我们创建的image同一个目录的。因为我们这个项目是要解压两个数据集,所以在结束第一个模型训练后要这个三个txt文件删掉。如果你使用的是aistudio,你可以去查看一下~/data/images 目录下有三个文件夹,COVID-19,Viral Pneumonia和NORMAL,分别存放着三个类别的图像。
!mkdir /home/aistudio/data/images
!unzip -q /home/aistudio/data/data34241/covid19-combo.zip -d /home/aistudio/data/images
!mv /home/aistudio/data/images/'COVID-19 Radiography Database'/* /home/aistudio/data/images
!rm -rf /home/aistudio/data/images/'COVID-19 Radiography Database'
!ls ~/data/images
PaddleClas还需要提供一个数据列表文件,里面每条数据按照 “文件路径 类别” 的格式标记,以供后续训练。同时还要要做的是把数据分组,你需要训练、评估和测试都需要数据集,代码如下
%cd ~
import os
base_dir = "/home/aistudio/data/images/" # CT图片所在路径
img_dirs = ["COVID-19", "NORMAL", "Viral Pneumonia"] # 三类CT图片文件夹名
file_names = ["train_list.txt", "val_list.txt", "test_list.txt"]
splits = [0, 0.6, 0.8, 1] # 按照 6 2 2 的比例对数据进行分组
for split_ind, file_name in enumerate(file_names):
with open(os.path.join("./data", file_name), "w") as f:
for type_ind, img_dir in enumerate(img_dirs):
imgs = os.listdir(os.path.join(base_dir, img_dir) )
for ind in range( int(splits[split_ind]* len(imgs)), int(splits[split_ind + 1] * len(imgs)) ):
print("|".format(img_dir + "/" + imgs[ind], type_ind), file = f)
文件列表制作完成后可以用head查看一下前10行。
! head /home/aistudio/data/train_list.txt
2、paddleclas配置
我把飞桨的一个压缩包已经上传到了平台上,可以直接点开我的项目下载解压就行。这个其实就是git从飞桨下载的代码包,但是经过裁剪不到150M,方便传输。解压后你需要运行第二行代码,用来进行环境初始化的,其实你可以打开看看,里面写着一些需要安装的依赖。
!unzip -q pdclas.zip
%cd pdclas
!pip install -r requirements.txt
接下里我是要利用GPU进行模型训练,所以在训练前需要初始化环境
!python -m pip install paddlepaddle-gpu==2.1.3.post101 -f https://www.paddlepaddle.org.cn/whl/linux/mkl/avx/stable.html
3、模型训练
下面就到了模型训练这一个关键一步。用PaddleClas训练模型需要编写config文件,这个文件是对训练步骤的细节进行定义,比如说epochs要多少,batch size要多少。这里可以根据你的硬件需求修改这个covid-2.yaml配置文件,如果文件丢失也可以复制下面的代码。但是一定要注意的是,我提供的文件由于我是使用32G内存的显卡,所以我的文件中的batch_size:给到了16。同时为了获取精确度高达95%以上的模型,我的配置文件是epoch是15步,训练时长是半个小时左右。
mode: 'train'
ARCHITECTURE:
# 使用的模型结构,可以参照 pdclas/config 下其他模型结构的cofig文件修改模型名称
# 比如 ResNet101
name: 'ResNet50_vd'
pretrained_model: "" # 通常使用预训练模型迁移能在小数据集上取得好的效果,但是预训练模型都是针对自然图像,因此没有使用
model_save_dir: "./output/"
classes_num: 3
total_images: 2905
save_interval: 1
validate: True
valid_interval: 1
epochs: 20
topk: 2
image_shape: [3, 1024, 1024]
LEARNING_RATE:
function: 'Cosine'
params:
lr: 0.00375
OPTIMIZER:
function: 'Momentum'
params:
momentum: 0.9
regularizer:
function: 'L2'
factor: 0.000001
TRAIN:
batch_size: 4 # 训练过程中一个batch的大小,如果你有幸分到32g显卡这个参数最高开到16
num_workers: 4
file_list: "/home/aistudio/data/train_list.txt"
data_dir: "/home/aistudio/data/images/"
delimiter: "|"
shuffle_seed: 0
transforms:
- DecodeImage:
to_rgb: True
to_np: False
channel_first: False
- RandFlipImage:
flip_code: 1
- NormalizeImage:
scale: 1./255.
- ToCHWImage:
VALID:
batch_size: 20
num_workers: 4
file_list: "/home/aistudio/data/val_list.txt"
data_dir: "/home/aistudio/data/images/"
delimiter: "|"
shuffle_seed: 0
transforms:
- DecodeImage:
to_rgb: True
to_np: False
channel_first: False
- ResizeImage:
resize_short: 1024
- NormalizeImage:
scale: 1.0/255.0
- ToCHWImage:
接下来开始训练,一般pdclas是在命令行环境下使用的,这里需要注意的是启动训练之前需要设置一个环境变量,代码如下。值得注意的是,如果出现报错说到这个paddle.enable_static()的话,你需要顺着路径打开这个文件,比如说train.py和export.py(导出那里),在上方补充下面这两段代码。
import paddle
paddle.enable_static()
%cd ~/pdclas/
import os
os.environ['PYTHONPATH']="/home/aistudio/pdclas"
!python -m paddle.distributed.launch --selected_gpus="0" tools/train.py -c ../covid-2.yaml
4、模型导出
训练好这是提前指定的存储路径,如果你没有更改的话可以运行下面的代码查看好的模型。
!ls ~/pdclas/output/ResNet50_vd
通过pdclas中提供的模型转换脚本将训练模型转换为推理模型,可以看到转换之后生成了两个文件,model是模型结构,params是模型权重。这里要提一下Paddle框架保存的权重文件分为两种:支持前向推理和反向梯度的训练模型 和 只支持前向推理的推理模型。二者的区别是推理模型针对推理速度和显存做了优化,裁剪了一些只在训练过程中才需要的tensor,降低显存占用,并进行了一些类似层融合,kernel选择的速度优化。ppcls在训练过程中保存的模型属于训练模型,在这个过程我们一般使用推理模型比较方便去导出onnx模型,第二个也要考虑到推理模型体量比较小方便传输。
!python tools/export_model.py --m=ResNet50_vd --p=output/ResNet50_vd/best_model_in_epoch_0/ppcls --o=../inference
!ls -lh /home/aistudio/inference/
这里我回到原路来展示一下现在我的文件夹,是这个项目在飞桨这一侧的所有文件了。%cd ~是为了回到根目录等会要进行推理。
%cd ~/
!ls
你可以使用导出的模型对任意一张数据集的图片进行推理,对照上面生成训练文件时的脚本,新冠类别为0,正常类别为1,其他肺炎类别为2,一般这里的分类都是这个患者是新冠肺炎患者,概率为百分百。
!python /home/aistudio/pdclas/tools/infer/predict.py --use_gpu=0 -i="/home/aistudio/COVID-19 (10).png" -m=/home/aistudio/inference/model -p=/home/aistudio/inference/params
由于我的环境安装在了paddleSeg这部分了,所以这里先留着到你下面导出了分割模型再回来导这个,这样子就不会出现报错。
!paddle2onnx \\
--model_dir inference/ \\
--model_filename model \\
--params_filename params \\
--save_file model_1.onnx \\
--opset_version 12
新冠CT分割部分与onnx导出
1、数据集预处理
安装 nibabel 库用于读取 nii 格式数据
!pip install --upgrade nibabel -i https://mirror.baidu.com/pypi/simple
对数据集进行解压
%cd ~/data/data34221/
!unzip -q -d .. 20_ncov_scan.zip # 扫描数据
!unzip -q -d ../Infection_Mask Infection_Mask.zip # 感染病灶分割标签
!unzip -qd ../Lung_Mask Lung_Mask.zip # 左右肺分割标签
# !unzip -qd ../Lung_Infection Lung_Infection.zip # 合并肺部和感染病灶标签,项目中没有用上
!ls ~/data/20_ncov_scan
上面可以看到解压出了20组扫描。PaddleSeg框架只接受图片格式的输入,因此我们需要对nii格式的CT扫描进行一点预处理,将他们转换为图片。此外在这个过程中我们将扫描数据 clip 到 [-512, 512] 的范围,防止强度过大或过小的噪点对训练产生影响。
import os
import nibabel as nib
import numpy as np
from tqdm import tqdm
import cv2
def listdir(path):
dirs = os.listdir(path)
dirs.sort() # 扫描和标签的文件名不完全相同,对两个目录下的所有文件进行排序可以保证二者能匹配上
return dirs
scan_dir = "/home/aistudio/data/20_ncov_scan" # CT扫描数据路径
label_dir = "/home/aistudio/data/Infection_Mask" # 病灶分割标签所在路径
output_dir = "/home/aistudio/data/prep"
scan_output = os.path.join(output_dir, "image") # CT图片输出路径
label_output = os.path.join(output_dir, "annotation") # 标签图片输出路径
if not os.path.exists(scan_output):
os.makedirs(scan_output)
if not os.path.exists(label_output):
os.makedirs(label_output)
wl, wh = (-512, 512) # 对CT进行窗口化的强度范围
scan_fnames = listdir(scan_dir)
label_fnames = listdir(label_dir)
for case_ind in tqdm( range(len(scan_fnames)) ):
scan_fname = scan_fnames[case_ind]
label_fname = label_fnames[case_ind]
scanf = nib.load(os.path.join(scan_dir, scan_fname)) # 使用nibabel库读入数据
scan = scanf.get_fdata()
labelf = nib.load(os.path.join(label_dir, label_fname))
label = labelf.get_fdata()
scan = np.rot90(scan) # 对读入数据的方向进行矫正,逆时针旋转90度
label = np.rot90(label)
# 窗口化操作,将范围转换到 0~255,便于存入图片
scan = scan.clip(wl, wh).astype("float16")
scan = ( (scan - wl)/(wh - wl) * 256)
for sli_ind in range(label.shape[2]):
scan_slice_path = os.path.join(scan_output, "-.png".format(scan_fname.rstrip(".nii.gz"), sli_ind ) )
label_slice_path = os.path.join(label_output, "-.png".format(scan_fname.rstrip(".nii.gz"), sli_ind ) )
cv2.imwrite(scan_slice_path, scan[:,:,sli_ind])
cv2.imwrite(label_slice_path, label[:,:,sli_ind])
print("图片转换完成")
! ls ~/data/prep/image -l | wc -l # 可以看到共生成了3500多张图片
这里一定要注意再注意,由于我们这个项目解压了两个数据集,并且需要生成的文件是在同一个路径下,所以你需要data三个文件删除后再执行接下来的操作。数据预处理完后,PaddleSeg需要我们为训练集,验证集和测试集分别提供一个文件列表 txt。下面代码的实际功能是将所有训练数据的路径按照三个集合的划分比例写入三个txt文件。
import os
data_base_dir = "/home/aistudio/data/prep"
scan_folder = "image"
label_folder = "annotation"
txt_path = "/home/aistudio/data/"
split = [0, 0.7, 0.9, 1.0] # 训练,验证和测试集的划分比例为 7:2:1
list_names = ["train_list.txt", "val_list.txt", "test_list.txt"]
curr_type = 0
img_count = len(os.listdir( os.path.join(data_base_dir, scan_folder ) ) )
split = [int(x * img_count) for x in split]
f = open(os.path.join(txt_path, list_names[curr_type]), "w")
for ind, slice_name in enumerate(os.listdir( os.path.join(data_base_dir, scan_folder)) ):
if ind < img_count - 1 and ind == split[curr_type + 1]:
curr_type += 1
f.close()
f = open(os.path.join(txt_path, list_names[curr_type]), "w")
print("|".format(os.path.join(scan_folder, slice_name), os.path.join(label_folder, slice_name)), file=f)
f.close()
# 可以通过 head 命令看一下生成的结果
!head ~/data/train_list.txt
2、paddleSeg配置
这里解压一下提供的飞桨分割代码文件,其实你也可以通过git下载,这个文件已经上传到项目中了
%cd /home/aistudio
!unzip paddleSeg.zip
这一步和上面一步是一样的,如果你已经初始化了GPU的环境就可以不用再执行了。
!python -m pip install paddlepaddle-gpu==2.1.3.post101 -f https://www.paddlepaddle.org.cn/whl/linux/mkl/avx/stable.html
这个需要执行,初始化分割的环境
!pip install -r paddleSeg/requirements.txt
3、模型训练
如果运行代码报错后出现了paddle.enable_static()这个字样,你需要打开训练文件trai.py加入下面代码。
import paddle
paddle.enable_static()
在这里给出了一根文件,我把我自己的配置文件也上传到了项目中。因为我需要高精度模型并且我使用的是32G的显卡,所以我的参数会和下面有所不同。epoch给到18,BATCH_SIZE给到20.
# 数据集配置
DATASET:
DATA_DIR: "/home/aistudio/data/prep" # 数据基路径,这个路径和文件列表中的路径 join 成实际的文件路径
NUM_CLASSES: 2 # 分割分为病灶和不是病灶两类
TRAIN_FILE_LIST: "/home/aistudio/data/train_list.txt" # 训练,验证和测试集的文件列表路径
VAL_FILE_LIST: "/home/aistudio/data/val_list.txt"
TEST_FILE_LIST: "/home/aistudio/data/test_list.txt"
SEPARATOR: "|" # 文件列表中用 | 分割训练数据和标签路径
IMAGE_TYPE: "gray" # 使用灰度图,单通道进行训练
# 预训练模型配置
MODEL:
MODEL_NAME: "unet" # 使用unet网络结构,可选的网络结构包括 deeplabv3p, unet, icnet,pspnet,hrnet
DEFAULT_NORM_TYPE: "bn"
# 其他配置
TRAIN_CROP_SIZE: (512, 512) # 训练输入数据大小
EVAL_CROP_SIZE: (512, 512)
AUG:
AUG_METHOD: "unpadding"
FIX_RESIZE_SIZE: (512, 512)
MIRROR: True # 左右镜像数据增强
BATCH_SIZE: 8 # 如果你有幸分到32g显卡,这个参数最高可以开到大概 20
TRAIN:
MODEL_SAVE_DIR: "./saved_model/unet_covid/"
SNAPSHOT_EPOCH: 1
TEST:
TEST_MODEL: "./saved_model/unet_covid/final"
SOLVER:
NUM_EPOCHS: 20 # 训练的时间较长,为了便于执行下面的代码这里只写了 1 个epoch。大概在15~20个epoch可以做到 85% 左右的准确率
LR: 0.001
LR_POLICY: "poly"
OPTIMIZER: "adam"
开始训练
%cd ~/paddleSeg
!python pdseg/train.py --cfg ~/covid.yaml --use_gpu --use_mpio --do_eval --use_vdl --vdl_log_dir ~/log
4、模型导出
导出代码,如果还是出现报错就打开这个导出文件,然后添加上面的两段代码
!python pdseg/export_model.py --cfg ~/covid.yaml TEST.TEST_MODEL ./saved_model/unet_covid/final/
安装paddle2onnx和他们的相关工具
!pip install pycocotools paddle2onnx
!pip install onnx==1.9.0
从GitHub下载好paddle2onnx的代码文件
%cd ~/
!git clone https://github.com/paddlepaddle/paddle2onnx --depth 1
初始化环境
!cd ~/paddle2onnx/ && python setup.py install
%cd ~/paddleSeg/
!ls
在这里我们导出这个我们分割模型的onnx模型,你可以指定导出的路径,然后下载到自己的电脑上。
!paddle2onnx \\
--model_dir freeze_model \\
--model_filename __model__ \\
--params_filename __params__ \\
--save_file model.onnx \\
--opset_version 12
当然,虽然我们的主要任务是导出ONNX模型,你也可以推理一下这个模型,你可以得到患者的肺部的分割图像
!python infer.py --conf=/home/aistudio/infer.yaml --input_dir=/home/aistudio/inference --image_dir="/home/aistudio/"
以上是关于openVINO+paddleCPU部署新冠肺炎CT图像分类识别与病害分割的主要内容,如果未能解决你的问题,请参考以下文章