优化预测基于matlab贝叶斯优化LSTM预测含Matlab源码 1329期
Posted 紫极神光
tags:
篇首语:本文由小常识网(cha138.com)小编为大家整理,主要介绍了优化预测基于matlab贝叶斯优化LSTM预测含Matlab源码 1329期相关的知识,希望对你有一定的参考价值。
一、贝叶斯网络及LSTM简介
1 贝叶斯网络
贝叶斯网络(Bayesian network),又称信念网络(Belief Network),或有向无环图模型(directed acyclic graphical model),是一种概率图模型,于1985年由Judea Pearl首先提出。它是一种模拟人类推理过程中因果关系的不确定性处理模型,其网络拓朴结构是一个有向无环图(DAG)。

贝叶斯网络的有向无环图中的节点表示随机变量{ X 1 , X 2 , . . . , X n }
它们可以是可观察到的变量,或隐变量、未知参数等。认为有因果关系(或非条件独立)的变量或命题则用箭头来连接。若两个节点间以一个单箭头连接在一起,表示其中一个节点是“因(parents)”,另一个是“果(children)”,两节点就会产生一个条件概率值。
例如,假设节点E直接影响到节点H,即E→H,则用从E指向H的箭头建立结点E到结点H的有向弧(E,H),权值(即连接强度)用条件概率P(H|E)来表示,如下图所示:

简言之,把某个研究系统中涉及的随机变量,根据是否条件独立绘制在一个有向图中,就形成了贝叶斯网络。其主要用来描述随机变量之间的条件依赖,用圈表示随机变量(random variables),用箭头表示条件依赖(conditional dependencies)。
此外,对于任意的随机变量,其联合概率可由各自的局部条件概率分布相乘而得出:

2.4.1 贝叶斯网络的结构形式
(1) head-to-head

依上图,所以有:P(a,b,c) = P(a)*P(b)P(c|a,b)成立,即在c未知的条件下,a、b被阻断(blocked),是独立的,称之为head-to-head条件独立。
(2) tail-to-tail
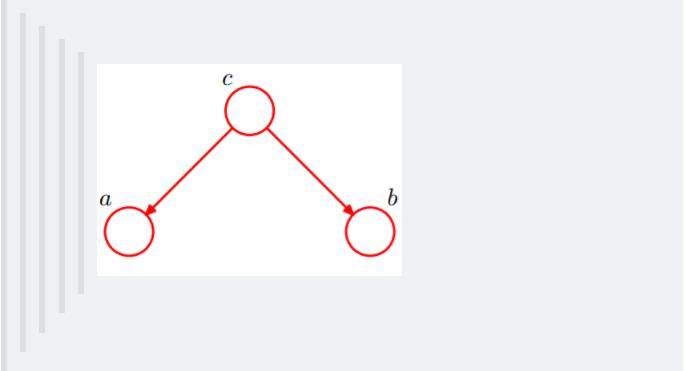
考虑c未知,跟c已知这两种情况:
在c未知的时候,有:P(a,b,c)=P©P(a|c)P(b|c),此时,没法得出P(a,b) = P(a)P(b),即c未知时,a、b不独立。
在c已知的时候,有:P(a,b|c)=P(a,b,c)/P©,然后将P(a,b,c)=P©P(a|c)P(b|c)带入式子中,得到:P(a,b|c)=P(a,b,c)/P© = P©P(a|c)*P(b|c) / P© = P(a|c)*P(b|c),即c已知时,a、b独立。
(3)head-to-tail

还是分c未知跟c已知这两种情况:
c未知时,有:P(a,b,c)=P(a)*P(c|a)*P(b|c),但无法推出P(a,b) = P(a)P(b),即c未知时,a、b不独立。
c已知时,有:P(a,b|c)=P(a,b,c)/P©,且根据P(a,c) = P(a)P(c|a) = P©P(a|c),可化简得到:

所以,在c给定的条件下,a,b被阻断(blocked),是独立的,称之为head-to-tail条件独立。
这个head-to-tail其实就是一个链式网络,如下图所示:

根据之前对head-to-tail的讲解,我们已经知道,在xi给定的条件下,xi+1的分布和x1,x2…xi-1条件独立。意味着啥呢?意味着:xi+1的分布状态只和xi有关,和其他变量条件独立。通俗点说,当前状态只跟上一状态有关,跟上上或上上之前的状态无关。这种顺次演变的随机过程,就叫做马尔科夫链(Markov chain)。对于马尔科夫链我们下一节再细讲。
2 LSTM简介
2.1 LSTM控制流程
LSTM的控制流程:是在前向传播的过程中处理流经细胞的数据,不同之处在于 LSTM 中细胞的结构和运算有所变化。

这一系列运算操作使得 LSTM具有能选择保存信息或遗忘信息的功能。咋一看这些运算操作时可能有点复杂,但没关系下面将带你一步步了解这些运算操作。
2.2 核心概念
LSTM 的核心概念在于细胞状态以及“门”结构。细胞状态相当于信息传输的路径,让信息能在序列连中传递下去。你可以将其看作网络的“记忆”。理论上讲,细胞状态能够将序列处理过程中的相关信息一直传递下去。
因此,即使是较早时间步长的信息也能携带到较后时间步长的细胞中来,这克服了短时记忆的影响。信息的添加和移除我们通过“门”结构来实现,“门”结构在训练过程中会去学习该保存或遗忘哪些信息。
2.3 Sigmoid
门结构中包含着 sigmoid 激活函数。Sigmoid 激活函数与 tanh 函数类似,不同之处在于 sigmoid 是把值压缩到 0~1 之间而不是 -1~1 之间。这样的设置有助于更新或忘记信息,因为任何数乘以 0 都得 0,这部分信息就会剔除掉。同样的,任何数乘以 1 都得到它本身,这部分信息就会完美地保存下来。这样网络就能了解哪些数据是需要遗忘,哪些数据是需要保存。

2.4 LSTM门结构
LSTM 有三种类型的门结构:遗忘门、输入门和输出门。
2.4.1 遗忘门
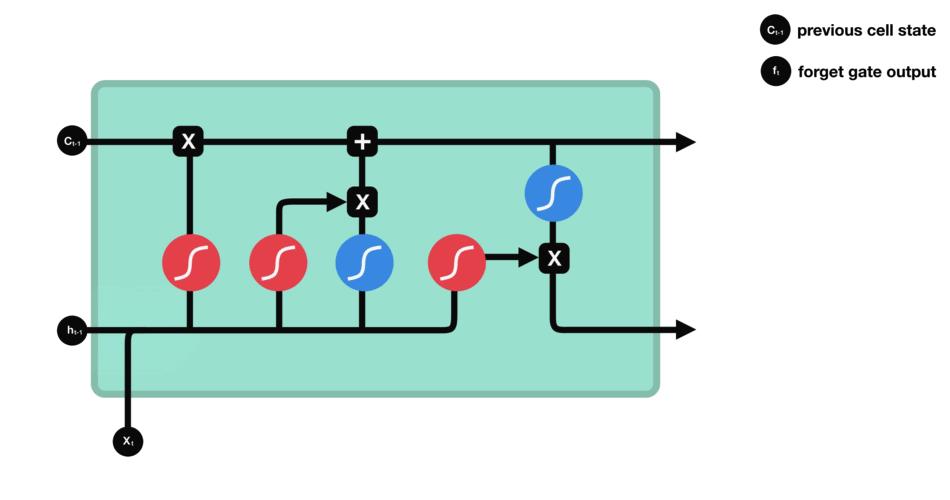
遗忘门的功能是决定应丢弃或保留哪些信息。来自前一个隐藏状态的信息和当前输入的信息同时传递到 sigmoid 函数中去,输出值介于 0 和 1 之间,越接近 0 意味着越应该丢弃,越接近 1 意味着越应该保留。
2.4.2 输入门
输入门用于更新细胞状态。首先将前一层隐藏状态的信息和当前输入的信息传递到 sigmoid 函数中去。将值调整到 0~1 之间来决定要更新哪些信息。0 表示不重要,1 表示重要。
其次还要将前一层隐藏状态的信息和当前输入的信息传递到 tanh 函数中去,创造一个新的侯选值向量。最后将 sigmoid 的输出值与 tanh 的输出值相乘,sigmoid 的输出值将决定 tanh 的输出值中哪些信息是重要且需要保留下来的。

2.4.3 细胞状态
下一步,就是计算细胞状态。首先前一层的细胞状态与遗忘向量逐点相乘。如果它乘以接近 0 的值,意味着在新的细胞状态中,这些信息是需要丢弃掉的。然后再将该值与输入门的输出值逐点相加,将神经网络发现的新信息更新到细胞状态中去。至此,就得到了更新后的细胞状态。

2.4.4 输出门
输出门用来确定下一个隐藏状态的值,隐藏状态包含了先前输入的信息。首先,我们将前一个隐藏状态和当前输入传递到 sigmoid 函数中,然后将新得到的细胞状态传递给 tanh 函数。
最后将 tanh 的输出与 sigmoid 的输出相乘,以确定隐藏状态应携带的信息。再将隐藏状态作为当前细胞的输出,把新的细胞状态和新的隐藏状态传递到下一个时间步长中去。

让我们再梳理一下。遗忘门确定前一个步长中哪些相关的信息需要被保留;输入门确定当前输入中哪些信息是重要的,需要被添加的;输出门确定下一个隐藏状态应该是什么。
二、部分源代码
clc; clear; close all;
%% ---------------------------- init Variabels ----------------------------
opt.Delays = 1:30;
opt.dataPreprocessMode = 'Data Standardization'; % 'None' 'Data Standardization' 'Data Normalization'
opt.learningMethod = 'LSTM';
opt.trPercentage = 0.80; % divide data into Test and Train dataset
% ---- General Deep Learning Parameters(LSTM and CNN General Parameters)
opt.maxEpochs = 400; % maximum number of training Epoch in deeplearning algorithms.
opt.miniBatchSize = 32; % minimum batch size in deeplearning algorithms .
opt.executionEnvironment = 'cpu'; % 'cpu' 'gpu' 'auto'
opt.LR = 'adam'; % 'sgdm' 'rmsprop' 'adam'
opt.trainingProgress = 'none'; % 'training-progress' 'none'
% ------------- BILSTM parameters
opt.isUseBiLSTMLayer = true; % if it is true the layer turn to the Bidirectional-LSTM and if it is false it will turn the units to the simple LSTM
opt.isUseDropoutLayer = true; % dropout layer avoid of bieng overfit
opt.DropoutValue = 0.5;
% ------------ Optimization Parameters
opt.optimVars = [
optimizableVariable('NumOfLayer',[1 4],'Type','integer')
optimizableVariable('NumOfUnits',[50 200],'Type','integer')
optimizableVariable('isUseBiLSTMLayer',[1 2],'Type','integer')
optimizableVariable('InitialLearnRate',[1e-2 1],'Transform','log')
optimizableVariable('L2Regularization',[1e-10 1e-2],'Transform','log')];
opt.isUseOptimizer = true;
opt.MaxOptimizationTime = 14*60*60;
opt.MaxItrationNumber = 60;
opt.isDispOptimizationLog = true;
opt.isSaveOptimizedValue = false; % save all of Optimization output on mat files
opt.isSaveBestOptimizedValue = true; % save Best Optimization output oد a mat file
%% --------------- load Data
data = loadData(opt);
if ~data.isDataRead
return;
end
%% --------------- Prepair Data
[opt,data] = PrepareData(opt,data);
%% --------------- Find Best LSTM Parameters with Bayesian Optimization
[opt,data] = OptimizeLSTM(opt,data);
%% --------------- Evaluate Data
[opt,data] = EvaluationData(opt,data);
%% ---------------------------- Local Functions ---------------------------
function data = loadData(opt)
[chosenfile,chosendirectory] = uigetfile({'*.xlsx';'*.csv'},...
'Select Excel time series Data sets','data.xlsx');
filePath = [chosendirectory chosenfile];
if filePath ~= 0
data.DataFileName = chosenfile;
data.CompleteData = readtable(filePath);
if size(data.CompleteData,2)>1
warning('Input data should be an excel file with only one column!');
disp('Operation Failed... '); pause(.9);
disp('Reloading data. '); pause(.9);
data.x = [];
data.isDataRead = false;
return;
end
data.seriesdataHeder = data.CompleteData.Properties.VariableNames(1,:);
data.seriesdata = table2array(data.CompleteData(:,:));
disp('Input data successfully read.');
data.isDataRead = true;
data.seriesdata = PreInput(data.seriesdata);
figure('Name','InputData','NumberTitle','off');
plot(data.seriesdata); grid minor;
title({['Mean = ' num2str(mean(data.seriesdata)) ', STD = ' num2str(std(data.seriesdata)) ];});
if strcmpi(opt.dataPreprocessMode,'None')
data.x = data.seriesdata;
elseif strcmpi(opt.dataPreprocessMode,'Data Normalization')
data.x = DataNormalization(data.seriesdata);
figure('Name','NormilizedInputData','NumberTitle','off');
plot(data.x); grid minor;
title({['Mean = ' num2str(mean(data.x)) ', STD = ' num2str(std(data.x)) ];});
elseif strcmpi(opt.dataPreprocessMode,'Data Standardization')
data.x = DataStandardization(data.seriesdata);
figure('Name','NormilizedInputData','NumberTitle','off');
plot(data.x); grid minor;
title({['Mean = ' num2str(mean(data.x)) ', STD = ' num2str(std(data.x)) ];});
end
else
warning(['In order to train network, please load data.' ...
'Input data should be an excel file with only one column!']);
disp('Operation Cancel.');
data.isDataRead = false;
end
end
function data = PreInput(data)
if iscell(data)
for i=1:size(data,1)
for j=1:size(data,2)
if strcmpi(data{i,j},'#NULL!')
tempVars(i,j) = NaN; %#ok
else
tempVars(i,j) = str2num(data{i,j}); %#ok
end
end
end
data = tempVars;
end
end
function vars = DataStandardization(data)
for i=1:size(data,2)
x.mu(1,i) = mean(data(:,i),'omitnan');
x.sig(1,i) = std (data(:,i),'omitnan');
vars(:,i) = (data(:,i) - x.mu(1,i))./ x.sig(1,i);
end
end
function vars = DataNormalization(data)
for i=1:size(data,2)
vars(:,i) = (data(:,i) -min(data(:,i)))./ (max(data(:,i))-min(data(:,i)));
end
end
% --------------- data preparation for LSTM ---
function [opt,data] = PrepareData(opt,data)
% prepare delays for time serie network
data = CreateTimeSeriesData(opt,data);
% divide data into test and train data
data = dataPartitioning(opt,data);
% LSTM data form
data = LSTMInput(data);
end
% ----Run Bayesian Optimization Hyperparameters for LSTM Network Parameters
function [opt,data] = OptimizeLSTM(opt,data)
if opt.isDispOptimizationLog
isLog = 2;
else
isLog = 0;
end
if opt.isUseOptimizer
opt.ObjFcn = ObjFcn(opt,data);
BayesObject = bayesopt(opt.ObjFcn,opt.optimVars, ...
'MaxTime',opt.MaxOptimizationTime, ...
'IsObjectiveDeterministic',false, ...
'MaxObjectiveEvaluations',opt.MaxItrationNumber,...
'Verbose',isLog,...
'UseParallel',false);
end
end
% ---------------- objective function
function ObjFcn = ObjFcn(opt,data)
ObjFcn = @CostFunction;
function [valError,cons,fileName] = CostFunction(optVars)
inputSize = size(data.X,1);
outputMode = 'last';
numResponses = 1;
dropoutVal = .5;
if optVars.isUseBiLSTMLayer == 2
optVars.isUseBiLSTMLayer = 0;
end
if opt.isUseDropoutLayer % if dropout layer is true
if optVars.NumOfLayer ==1
if optVars.isUseBiLSTMLayer
opt.layers = [ ...
sequenceInputLayer(inputSize)
bilstmLayer(optVars.NumOfUnits,'OutputMode',outputMode)
dropoutLayer(dropoutVal)
fullyConnectedLayer(numResponses)
regressionLayer];
else
opt.layers = [ ...
sequenceInputLayer(inputSize)
lstmLayer(optVars.NumOfUnits,'OutputMode',outputMode)
dropoutLayer(dropoutVal)
fullyConnectedLayer(numResponses)
regressionLayer];
end
elseif optVars.NumOfLayer==2
if optVars.isUseBiLSTMLayer
opt.layers = [ ...
sequenceInputLayer(inputSize)
bilstmLayer(optVars.NumOfUnits,'OutputMode','sequence')
dropoutLayer(dropoutVal)
bilstmLayer(optVars.NumOfUnits,'OutputMode',outputMode)
dropoutLayer(dropoutVal)
fullyConnectedLayer(numResponses)
regressionLayer];
else
opt.layers = [ ...
sequenceInputLayer(inputSize)
lstmLayer(optVars.NumOfUnits,'OutputMode','sequence')
dropoutLayer(dropoutVal)
lstmLayer(optVars.NumOfUnits,'OutputMode',outputMode)
dropoutLayer(dropoutVal)
fullyConnectedLayer(numResponses)
regressionLayer];
end
三、运行结果


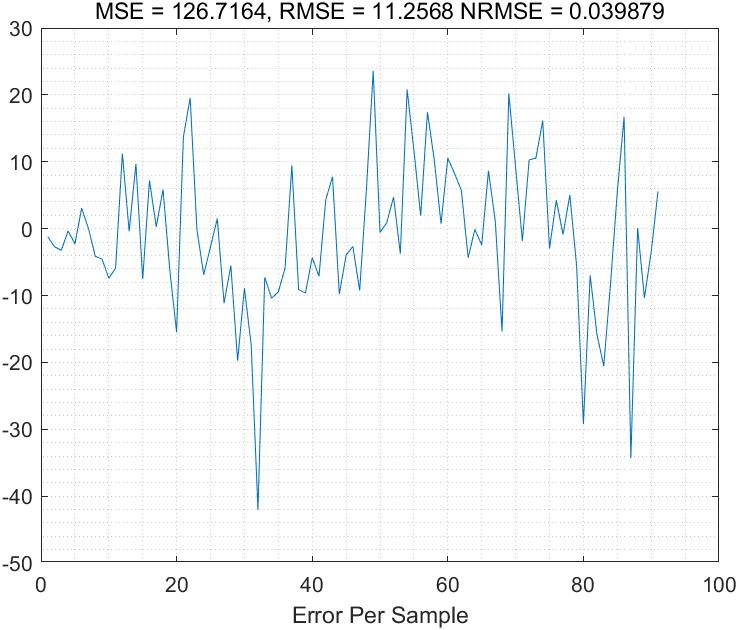
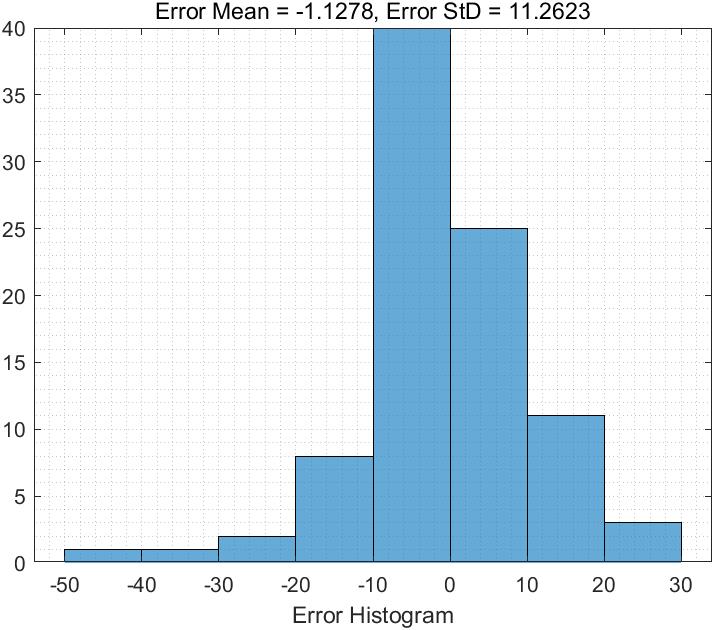

四、matlab版本及参考文献
1 matlab版本
2014a
2 参考文献
[1] 包子阳,余继周,杨杉.智能优化算法及其MATLAB实例(第2版)[M].电子工业出版社,2016.
[2]张岩,吴水根.MATLAB优化算法源代码[M].清华大学出版社,2017.
[3]周品.MATLAB 神经网络设计与应用[M].清华大学出版社,2013.
[4]陈明.MATLAB神经网络原理与实例精解[M].清华大学出版社,2013.
[5]方清城.MATLAB R2016a神经网络设计与应用28个案例分析[M].清华大学出版社,2018.
以上是关于优化预测基于matlab贝叶斯优化LSTM预测含Matlab源码 1329期的主要内容,如果未能解决你的问题,请参考以下文章
时间序列预测基于matlab贝叶斯优化LSTM时间序列预测(单变量单输出)含Matlab源码 651期
LSTM预测基于matlab贝叶斯网络改进LSTM预测含Matlab源码 1158期
LSTM回归预测基于matlab灰狼算法优化LSTM回归预测含Matlab源码 2038期
LSTM回归预测基于matlab灰狼算法优化LSTM回归预测含Matlab源码 2038期