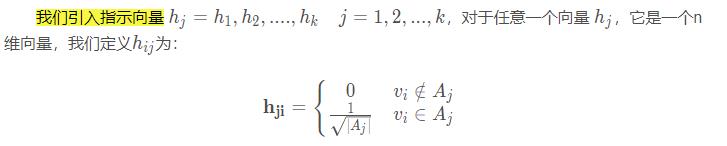谱聚类初探
Posted UQI-LIUWJ
tags:
篇首语:本文由小常识网(cha138.com)小编为大家整理,主要介绍了谱聚类初探相关的知识,希望对你有一定的参考价值。
1, 谱聚类引入

对于这样分布的样本,我们怎么进行聚类呢?我们可以使用核函数+K-means来聚类,当然也可以使用本节将要介绍的谱聚类。
2,模型介绍
谱聚类是一种基于图模型的聚类方法(一般图是带权重的无向图)。比起传统的K-Means算法,谱聚类对数据分布的适应性更强,聚类效果也很优秀,同时聚类的计算量也小很多。

谱聚类的主要思想是把所有的数据看做空间中的点,这些点之间可以用边连接起来。距离较远的两个点之间的边权重值较低,而距离较近的两个点之间的边权重值较高,通过对所有数据点组成的图进行切图,让切图后不同的子图间边权重和尽可能的低,而子图内的边权重和尽可能的高,从而达到聚类的目的。
2.1 几个图的概念
先定义几个概念:
图:G={V,E}
节点集合:V={1,2,……,N}
2.1.1 边集合与邻接矩阵
边集合: E:W=[wij] 1≤i,j≤N ,这里我们用一个相似度矩阵W来表达节点与节点之间,即边的信息。其中相似度的定义有以下几种:
1、ϵ-邻近法

从上式可见,两点的权重要不就是ϵ, 要不就是0, 没有其他信息了。距离远近度量很不精确,因此在实际应用中,我们很少使用ϵ-邻近法。
2、K邻近法
利用KNN算法遍历所有的样本点,取每个样本最近的k个点作为近邻,只有和样本距离最近的k个点之间的边权重大于0。但是这种方法会造成重构之后的邻接矩阵W非对称,我们后面的算法需要对称邻接矩阵。为了解决这种问题,一般采取下面两种方法之一:
3、全连接法
相比前两种方法,第三种方法所有的点之间的权重值都大于0,因此称之为全连接法。在实际的应用中,使用第三种全连接法来建立邻接矩阵是最普遍的。
可以选择不同的核函数来定义边权重,常用的有多项式核函数,高斯核函数和Sigmoid核函数。最常用的是高斯核函数RBF。
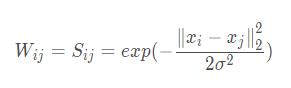
2.1.2 度与度矩阵
度:度为该顶点与其他顶点连接权值之和。度矩阵是一个对角矩阵。
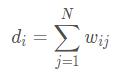
2.2 切割
2.2.1 两类的切割
我们先看切割成两类的情况
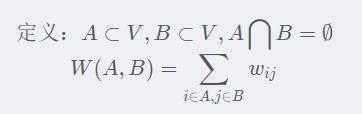
也就是两个切割A,B之间的度量是任意A中的点和任意B中的点之间的相似度。也就是“切口”两侧的点之间的相似度权重。
2.2.2 K类的切割
然后我们推广到K个类别
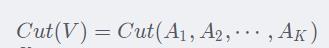
那么这个K类切割怎么度量呢?从在两类切割的情况我们知道,切割的度量只取决于“切口”两侧点之间的相似度权重。那么K类切割也是同理,只不过原来是切一刀,现在是切K刀,每一刀切下Ai和其他部分。
于是我们就有:
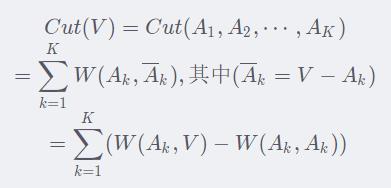
那么我们可以通过调整Ai的分配,获得最小的Cut。

2.3 Ncut
但是如果仅最优化这边的cut,会有一个不足。就是我们希望分割效果更好,希望每一个分割“紧凑”一点。也就是类间的相似度低(w小),类内的相似度大(w大)。那么Cut的定义就无法满足我们这个需求了。为了满足这个“分割紧凑”的需求,我们对Cut进行归一化操作:
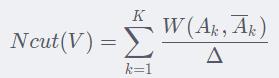
这里的Δ定义为某一类内点的度(虽然我觉得可能是类内点之间的度更合理一点?欢迎讨论)

整合一下,Normalized Cut (NCut)的定义为
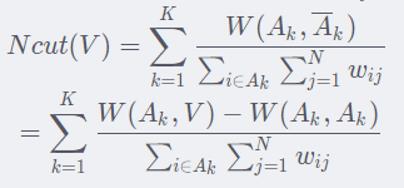
我们的目标是
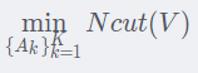
3, Ncut模型的矩阵表示
通过上一小节,我们知道
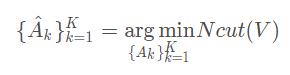
为了更方便地求解这个优化问题,我们一般要将它转化为矩阵形式
3.1 指示向量
我们先来看一下等号左边,我们定义一个指示向量indicator vector,它类似于one-hot向量
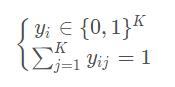

3.2 Ncut
接下来我们要对Ncut进行矩阵表示
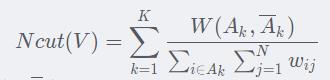
NCut是一个求和的数值,那么我们可以将其转化为对角矩阵求迹(trace)

然后把分子分母分离(对角矩阵求逆相当于对每一个对角线元素求逆):

整合一下,即:
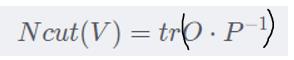
但是,我们目前知道的是相似度矩阵W(N×N维)和指示向量Y(N×K维)【这里我们先给定一个特定的Y,然后将O,P用Y来表示。之后再用优化问题求解Y,所以此时Y暂定为已知的】
3.2.1 求P
我们先看一下 (K×K维):
(K×K维):

对于每一个 ,
, 的结果是一个K×K的矩阵。如果
的结果是一个K×K的矩阵。如果 中第j维是1(其他是0),那么
中第j维是1(其他是0),那么 中只有第(j,j)坐标元素为1,其余为0。
中只有第(j,j)坐标元素为1,其余为0。
那么这样所有的 求和的结果,元素值为1的部分全在对角线上.
求和的结果,元素值为1的部分全在对角线上.
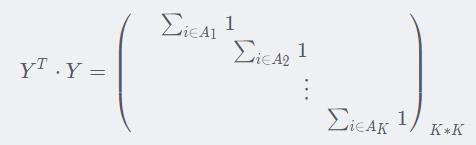
对角线上的每个元素都是这一类的元素个数。这个已经和P(下图)很像了,唯一的区别就是求和的内容。

我们在前面又有

所以前面的P也可以写成
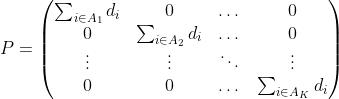

整合一下,有:
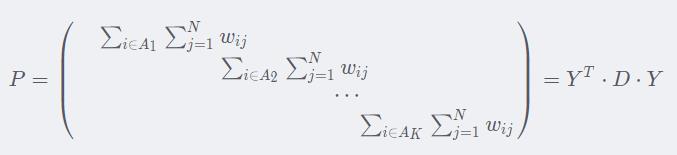

3.2.2. 求O
最后我们来看O
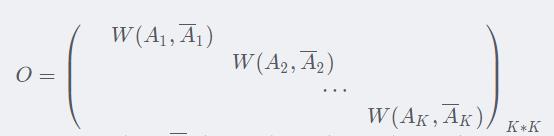
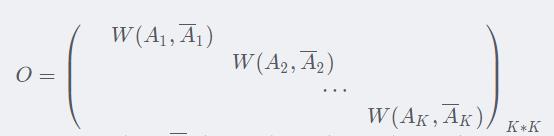
对于第一项,也就是组别k内所有点的度之和,即
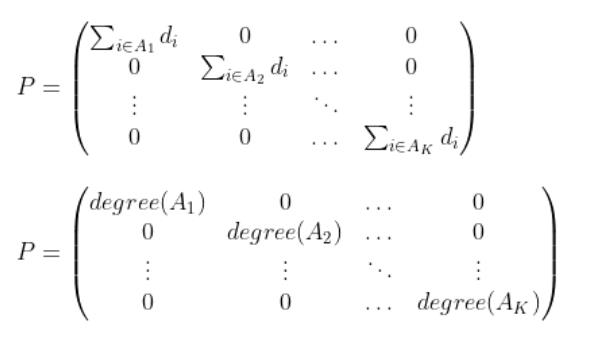
而我们前面已经求得了

而对于第二项

我们先猜测一下,因为我们现在就只知道W和Y,我们先试试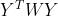 行不行.
行不行.
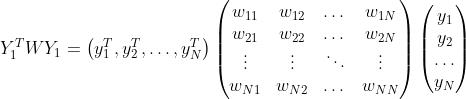
因为我们只是需要求迹,而

是对角矩阵,所以这里我们也只需要验证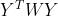 的对角线部分即可。
的对角线部分即可。
我们取 ,什么时候元素会到对角线上呢?那就是yi和yj在一组里面(比如第k组)。此时第k行第k列的元素+
,什么时候元素会到对角线上呢?那就是yi和yj在一组里面(比如第k组)。此时第k行第k列的元素+
所以最后,只看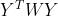 对角线上的元素,那么这个矩阵就是
对角线上的元素,那么这个矩阵就是

3.3 结论
所以
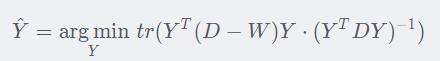
其中D-W我们称之为拉普拉斯矩阵
3.4 求最优方案的方法
在之后会涉及的RatioCut中,我们去求拉普拉斯矩阵L的最小的前k个特征值,然后求出对应的特征向量并标准化,然后对结果进行一次传统的聚类。
Ncut和RatioCut唯一的区别就是分母的D,所以仿RatioCut方法,我们这里进行特征分解的矩阵为标准化的拉普拉斯矩阵,即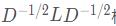
4 拉普拉斯矩阵
拉普拉斯矩阵(Laplacian matrix)),也称为基尔霍夫矩阵, 是表示图的一种矩阵。给定一个有n个顶点的图,其拉普拉斯矩阵被定义为:L=D−W 其中D为图的度矩阵,W为图的邻接矩阵。
4.1 拉普拉斯矩阵举例
以下图为例:

| 邻接矩阵 |  |
| 度矩阵 |  |
| 拉普拉斯矩阵 |  |
4.2 拉普拉斯矩阵性质
证明:
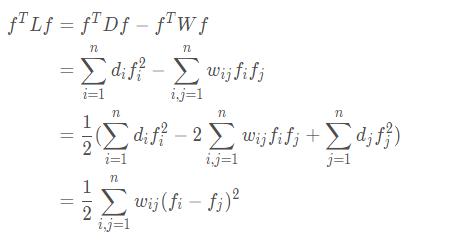
5 RatioCut切图
大体思路和Ncut是一样的,不过分母除的不是某个分割的度之和,而是某个分割中点的个数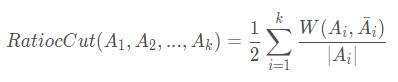
和Ncut一样,这边的指示向量也表示了第i个元素(i∈[1,n])在第j个分割中(j∈[1,k])
那么我们对每一个分割Ai有:
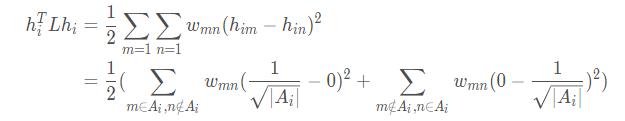
(展开的时候,都属于Ai的和都不属于Ai的相减为0,所以不显示出来)

(Ai分割边缘上所有点的权重之和就是cut(Ai,~Ai))

汇总到一块,总的分割的RatioCut为





通过找到L的最小的k个特征值,可以得到对应的k个特征向量,这k个特征向量组成一个nxk维度的矩阵,即为我们的H。
由于我们在使用维度规约的时候损失了少量信息,导致得到的优化后的指示向量h对应的H现在不能完全指示各样本的归属,因此一般在得到 n × k维度的矩阵H后还需要对每一行进行一次传统的聚类,比如使用K-Means聚类。
6 谱聚类算法流程汇总
以Ncut为例
输入:样本集D=(x1,x2,x3,......,xn)、相似矩阵的生成方式、 降维后的维度k1(需要分成几个分割)、聚类方法、聚类后的维度k2
输出: 簇划分C=(c1,c2,.....ck2)
流程:
1)根据输入的相似矩阵的生成方式构建样本的相似矩阵S
2)根据相似矩阵S构建邻接矩阵W ,构建度矩阵D
3)计算出拉普拉斯矩阵L
4)构建标准化后的拉普拉斯矩阵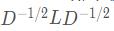
5)计算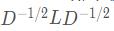 最小的k1个特征值所各自对应的特征向量f
最小的k1个特征值所各自对应的特征向量f
6) 将各自对应的特征向量f组成的矩阵按行标准化,最终组成n × k1的特征矩阵F(第j个矩阵(j∈[1,n])属于第i个分割(i∈[1,k1]))
7)对F中的每一行作为一个k1维的样本,共n个样本,用输入的聚类方法进行聚类,聚类维数为k2
8)得到簇划分C=(c1,c2,.....ck2)
7 谱聚类python实现
为了方便记忆,我按照流程1~8依次实现
7-0-1 导入库
import numpy as np
from sklearn import datasets7-0-2 加载鸢尾花数据
#加载鸢尾花数据
iris=datasets.load_iris()
'''
print(iris['data'].shape)
(150, 4)
print(iris['data'][0])
array([5.1, 3.5, 1.4, 0.2])
'''
m=iris['data'].shape[0]
n=iris['data'].shape[1]7-1 构建样本的相似矩阵S

#相似矩阵
AMatrix=np.zeros((m,m))
#求两条记录之间的距离(相似度),用高斯核函数
def AffMatrix(arrA,arrB):
sigma=0.5
diff=arrA-arrB #两个ndarray逐元素相减
diff=np.square(diff)#相减的结果逐元素平方
a=np.sum(diff)#到这里就完成了||x-x'||^2这一部分了
b=2*(np.square(sigma))#到这里完成了2*σ^2
w=np.exp(-a/b) #高斯核函数
#print(w)
return w
7-2 根据相似矩阵S构建邻接矩阵W ,构建度矩阵D
7-2-1 邻接矩阵
#计算邻接矩阵
def M_Matrix(m,AMatrix):
for i in range(m):
for j in range(m):
AMatrix[i][j]=AffMatrix(iris['data'][i],iris['data'][j])
#计算两两的相似度
AMatrix=AMatrix-np.identity(m)
#我们认为自己到自己是没有边相连的,所以需要让对角线元素变为0
#自己和自己的高斯核结果为0
return AMatrix
w=M_Matrix(m,AMatrix)
'''
w.shape
(150, 150)
'''7-2-2 度矩阵
D=np.diag(np.sum(w,axis=0))这里复习一个知识,就是axis的事情
axis=0 第0个坐标的元素叠加,其他坐标的元素不变 sum([0,1,2,....,n][x][y])
axis=1 第1个坐标的元素叠加,其他坐标的元素不变 sum([x][0,1,2,....,n][y])
7.3 计算出拉普拉斯矩阵L=D−W
L=D-w7.4 构建标准化后的拉普拉斯矩阵
#np.power(np.sum(w,axis=0),-0.5)
#D的对角线元素开根号
Dn=np.diag(np.power(np.sum(w,axis=0),-0.5))
#Dn=D^(-1/2)
L=Dn.dot(L).dot(Dn)7.5 计算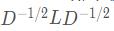 最小的k1个特征值所各自对应的特征向量f
最小的k1个特征值所各自对应的特征向量f
eigvals, eigvecs = np.linalg.eig(L)
# 规范化后的拉普拉斯矩阵 特征分解
k = 3
indices = eigvals.argsort()[:k]
#最小的三个特征值
k_max_eigvecs = eigvecs[:,indices]
#最小的三个特征值对应的特征向量
'''
k_max_eigvecs.shape
(150,3)
'''7.7 对F中的每一行作为一个k1维的样本,共n个样本,用输入的聚类方法进行聚类,聚类维数为k2
#KNN进行聚类
from sklearn.cluster import KMeans
res = KMeans(n_clusters=k).fit_predict(k_max_eigvecs)
7.9 可视化
def draw():
colorSet = ["black", "blue", "green", "yellow", "red", "purple", "orange", "brown"]
for i in range(m):
x = iris['data'][i][0]
y = iris['data'][i][1]
color = colorSet[res[i] % len(colorSet)]
plt.scatter(x,y, marker="o", c=color, s=20)
plt.xlabel("x")
plt.ylabel("y")
plt.show()
draw()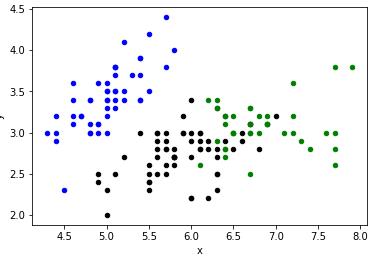
8 sklearn库中的谱聚类使用
在scikit-learn的类库中,sklearn.cluster.SpectralClustering实现了基于Ncut的谱聚类,没有实现基于RatioCut的切图聚类。同时,对于相似矩阵的建立,也只是实现了基于K邻近法和全连接法的方式,没有基于ϵ-邻近法的相似矩阵。
8.1 SpectralClustering中的参数
8.1.1 n_clusters
代表我们在对谱聚类切图时降维到的维数,同时也是最后一步聚类算法聚类到的维数。
8.1.2 affinity:
我们的相似矩阵的建立方式。可以选择的方式有三类,
第一类是 **‘nearest_neighbors’**即K邻近法。
第二类是**‘precomputed’**即自定义相似矩阵。选择自定义相似矩阵时,需要自己调用set_params来自己设置相似矩阵。
第三类是全连接法,可以使用各种核函数来定义相似矩阵,还可以自定义核函数。
最常用的是内置高斯核函数’rbf’。其他比较流行的核函数有‘linear’即线性核函数, ‘poly’即多项式核函数, ‘sigmoid’即sigmoid核函数。
如果选择了这些核函数, 对应的核函数参数在后面有单独的参数需要设置。
affinity默认是高斯核’rbf’。
8.1.3 核函数参数gamma:
如果我们在affinity参数使用了多项式核函数 ‘poly’,高斯核函数‘rbf’, 或者’sigmoid’核函数,那么我们就需要对这个参数进行调参。
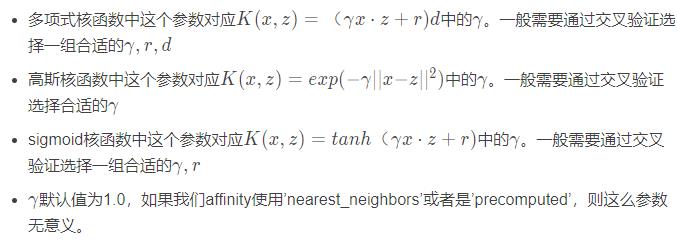
8.1.4 核函数参数degree
如果我们在affinity参数使用了多项式核函数 ‘poly’,那么我们就需要对这个参数进行调参。这个参数对应 中的d。默认是3。
中的d。默认是3。
8.1.5 核函数参数coef0:
如果我们在affinity参数使用了多项式核函数 ‘poly’,或者sigmoid核函数,那么我们就需要对这个参数进行调参。
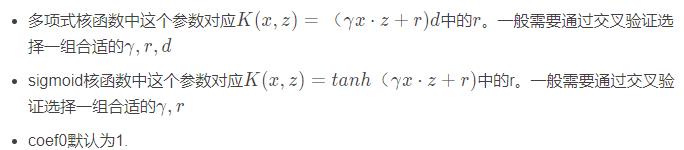
8.1.6 kernel_params
如果affinity参数使用了自定义的核函数,则需要通过这个参数传入核函数的参数。
8.1.7 n_neighbors
如果我们affinity参数指定为’nearest_neighbors’即K邻近法,则我们可以通过这个参数指定KNN算法的K的个数。默认是10
8.2 代码
from sklearn.cluster import SpectralClustering
from sklearn import metrics
iris=datasets.load_iris()
y_pred = SpectralClustering(gamma=0.1,n_clusters=3).fit_predict(iris['data'])8.3 可视化
可以看到结果和我们之前自己实现的是差不多的
def draw():
colorSet = ["black", "blue", "green", "yellow", "red", "purple", "orange", "brown"]
for i in range(m):
x = iris['data'][i][0]
y = iris['data'][i][1]
color = colorSet[y_pred[i] % len(colorSet)]
plt.scatter(x,y, marker="o", c=color, s=20)
plt.xlabel("x")
plt.ylabel("y")
plt.show()
draw()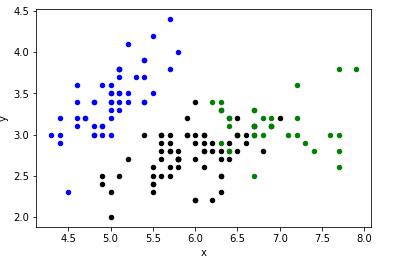
以上是关于谱聚类初探的主要内容,如果未能解决你的问题,请参考以下文章