附源码医学图像分割入门实践
Posted LYNNzZ361
tags:
篇首语:本文由小常识网(cha138.com)小编为大家整理,主要介绍了附源码医学图像分割入门实践相关的知识,希望对你有一定的参考价值。
有一定深度学习图像分割基础,至少阅读过部分语义分割或者医学图像分割文献
前面的一篇 医学图像分割多目标分割(多分类)实践文章记录了笔者在医学图像分割踩坑入门的实践,但当时的源码不够完整。通过博客的评论互动和私信发现有很多同学同样在做这个方向,最近空闲的时间也让我下定决心重新复现之前代码并进行一些注释和讲解,希望能对该方向入坑的同学提供一些帮助。
先上源码。
1 完整源码
【完整源码地址】: pytorch-medical-image-segmentation
重新整理了之前的代码,利用其中一个数据集(前面文章提到的基于磁共振成像的膀胱内外壁分割与肿瘤检测,)作为案例,但由于没有官方的数据授权,我仅将该数据集的一小部分数据拿来做演示。
我将代码托管到了国内的Gitee上(主要觉得比Github速度快点),源码 pytorch-medical-image-segmentation可直接下载运行。
【代码目录结构】:
pytorch-medical-image-segmentation/
|-- checkpoint # 存放训练好的模型
|-- dataprepare # 数据预处理的一些方法
|-- datasets # 数据加载的一些方法
|-- log # 日志文件
|-- media
| |-- Datasets # 存放数据集
|-- networks # 存放模型
|-- test # 测试相关
|-- train # 训练相关
|-- utils # 一些工具函数
|-- validate # 验证相关
|-- README.md
2 数据集
来自ISICDM 2019 临床数据分析挑战赛的基于磁共振成像的膀胱内外壁分割与肿瘤检测数据集。
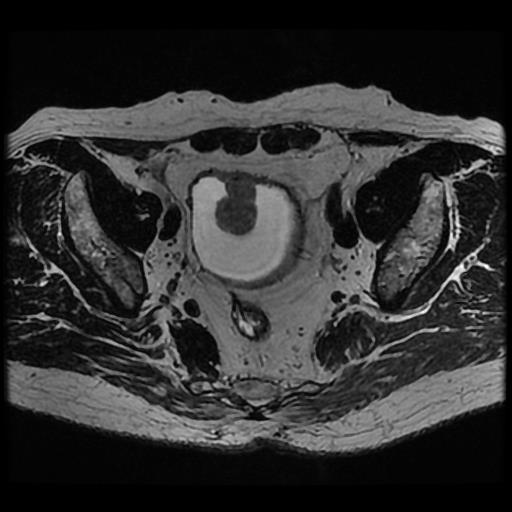

【说明】:笔者没有权限公开分享该数据集,需要完整数据集可通过官网获取。若官网数据集也不能获取,可利用其他数据集代替,本教程主要是提供分割的大体代码思路,不局限于某一个具体的数据集。
【灰度值】:灰色128为膀胱内外壁,白色255为肿瘤。
【分割任务】:同时分割出膀胱内外壁和肿瘤部分
【分析】:我们需要分割出膀胱内外壁和肿瘤,再加上黑色背景,相当于是一个三分类问题。
3 分割任务的思路
根据笔者做分割的一些经验,医学图像分割任务的步骤大体是以下几个步骤:
- 数据预处理
- 模型设计
- 评估指标和损失函数选择
- 训练
- 验证
- 测试
接下来我们通过代码一步步完成分割的过程。
4 代码实现
4.1 数据预处理
此次的膀胱数据集本身是官方处理好的png图像,不像常规的MRI和CT图像是nii格式的,因此数据处理起来相对容易。
为了简单起见,笔者主要对原始数据做了数据集划分、对标签进行One-hot、裁剪等操作。由于不同的数据集做的数据增广操作(一般会有旋转、缩放、弹性形变等)不太一样,本案例中省略了数据增广的操作。
首先,我们对原始数据集进行重新数据划分,这里使用了五折交叉验证(5-fold validation)的方法对数据进行划分,不了解交叉验证的同学可以先去网上搜索了解一下。
这里是将数据集的名字划分到不同txt文件中,而不是真正的将原始数据划分到不同的文件夹中,后面读取的时候也是通过名字来读取,这样更加方便。
# /dataprepare/kfold.py
import os, shutil
from sklearn.model_selection import KFold
# 按K折交叉验证划分数据集
def dataset_kfold(dataset_dir, save_path):
data_list = os.listdir(dataset_dir)
kf = KFold(5, False, 12345) # 使用5折交叉验证
for i, (tr, val) in enumerate(kf.split(data_list), 1):
print(len(tr), len(val))
if os.path.exists(os.path.join(save_path, 'train{}.txt'.format(i))):
# 若该目录已存在,则先删除,用来清空数据
print('清空原始数据中...')
os.remove(os.path.join(save_path, 'train{}.txt'.format(i)))
os.remove(os.path.join(save_path, 'val{}.txt'.format(i)))
print('原始数据已清空。')
for item in tr:
file_name = data_list[item]
with open(os.path.join(save_path, 'train{}.txt'.format(i)), 'a') as f:
f.write(file_name)
f.write('\\n')
for item in val:
file_name = data_list[item]
with open(os.path.join(save_path, 'val{}.txt'.format(i)), 'a') as f:
f.write(file_name)
f.write('\\n')
if __name__ == '__main__':
# 膀胱数据集划分
# 首次划分数据集或者重新划分数据集时运行
dataset_kfold(os.path.join('..\\media\\Datasets\\Bladder', 'raw_data\\Labels'),
os.path.join('..\\media\\Datasets\\Bladder', 'raw_data'))
运行后会生成以下文件,相当于是将数据集5份,每一份对应自己的训练集和验证集。
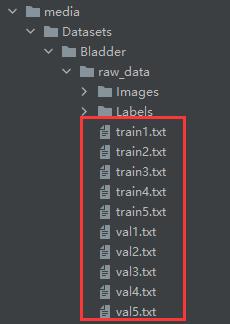
数据集划分好了,接下来就要写数据加载的类和方法,以便在训练的时候加载我们的数据。
# /datasets/bladder.py
import os
import cv2
import numpy as np
from PIL import Image
from torch.utils import data
from utils import helpers
'''
128 = bladder
255 = tumor
0 = background
'''
palette = [[0], [128], [255]] # one-hot的颜色表
num_classes = 3 # 分类数
def make_dataset(root, mode, fold):
assert mode in ['train', 'val', 'test']
items = []
if mode == 'train':
img_path = os.path.join(root, 'Images')
mask_path = os.path.join(root, 'Labels')
if 'Augdata' in root: # 当使用增广后的训练集
data_list = os.listdir(os.path.join(root, 'Labels'))
else:
data_list = [l.strip('\\n') for l in open(os.path.join(root, 'train{}.txt'.format(fold))).readlines()]
for it in data_list:
item = (os.path.join(img_path, it), os.path.join(mask_path, it))
items.append(item)
elif mode == 'val':
img_path = os.path.join(root, 'Images')
mask_path = os.path.join(root, 'Labels')
data_list = [l.strip('\\n') for l in open(os.path.join(
root, 'val{}.txt'.format(fold))).readlines()]
for it in data_list:
item = (os.path.join(img_path, it), os.path.join(mask_path, it))
items.append(item)
else:
img_path = os.path.join(root, 'Images')
data_list = [l.strip('\\n') for l in open(os.path.join(
root, 'test.txt')).readlines()]
for it in data_list:
item = (os.path.join(img_path, 'c0', it))
items.append(item)
return items
class Dataset(data.Dataset):
def __init__(self, root, mode, fold, joint_transform=None, center_crop=None, transform=None, target_transform=None):
self.imgs = make_dataset(root, mode, fold)
self.palette = palette
self.mode = mode
if len(self.imgs) == 0:
raise RuntimeError('Found 0 images, please check the data set')
self.mode = mode
self.joint_transform = joint_transform
self.center_crop = center_crop
self.transform = transform
self.target_transform = target_transform
def __getitem__(self, index):
img_path, mask_path = self.imgs[index]
file_name = mask_path.split('\\\\')[-1]
img = Image.open(img_path)
mask = Image.open(mask_path)
if self.joint_transform is not None:
img, mask = self.joint_transform(img, mask)
if self.center_crop is not None:
img, mask = self.center_crop(img, mask)
img = np.array(img)
mask = np.array(mask)
# Image.open读取灰度图像时shape=(H, W) 而非(H, W, 1)
# 因此先扩展出通道维度,以便在通道维度上进行one-hot映射
img = np.expand_dims(img, axis=2)
mask = np.expand_dims(mask, axis=2)
mask = helpers.mask_to_onehot(mask, self.palette)
# shape from (H, W, C) to (C, H, W)
img = img.transpose([2, 0, 1])
mask = mask.transpose([2, 0, 1])
if self.transform is not None:
img = self.transform(img)
if self.target_transform is not None:
mask = self.target_transform(mask)
return (img, mask), file_name
def __len__(self):
return len(self.imgs)
if __name__ == '__main__':
np.set_printoptions(threshold=9999999)
from torch.utils.data import DataLoader
import utils.image_transforms as joint_transforms
import utils.transforms as extended_transforms
def demo():
train_path = r'../media/Datasets/Bladder/raw_data'
val_path = r'../media/Datasets/Bladder/raw_data'
test_path = r'../media/Datasets/Bladder/test'
center_crop = joint_transforms.CenterCrop(256)
test_center_crop = joint_transforms.SingleCenterCrop(256)
train_input_transform = extended_transforms.NpyToTensor()
target_transform = extended_transforms.MaskToTensor()
train_set = Dataset(train_path, 'train', 1,
joint_transform=None, center_crop=center_crop,
transform=train_input_transform, target_transform=target_transform)
train_loader = DataLoader(train_set, batch_size=1, shuffle=False)
for (input, mask), file_name in train_loader:
print(input.shape)
print(mask.shape)
img = helpers.array_to_img(np.expand_dims(input.squeeze(), 2))
gt = helpers.onehot_to_mask(np.array(mask.squeeze()).transpose(1, 2, 0), palette)
gt = helpers.array_to_img(gt)
cv2.imshow('img GT', np.uint8(np.hstack([img, gt])))
cv2.waitKey(1000)
demo()
通常我会在数据预处理和加载类已写好后,运行代码测试数据的加载过程,看加载的数据是否有问题。通过可视化的结果可以看到加载的数据是正常的。
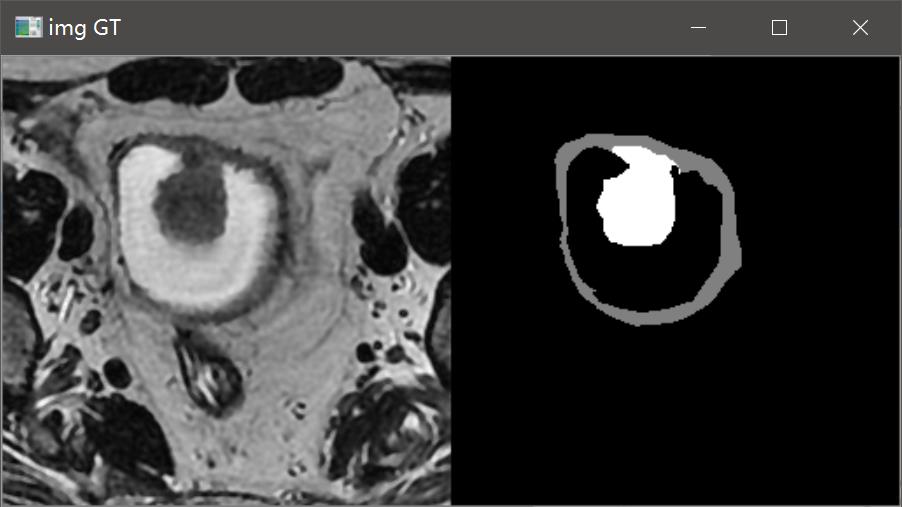
我们在对ground truth反one-hot进行可视化时,改变颜色表palette中的颜色值,就可以将ground truth重新映射成我们想要的颜色,例如:
我们修改上面的部分代码,将颜色表palette修改成三色值([x, x, x]里边有三个数字,单色[x]就对应灰色图像)将gt映射成彩色图像。
for (input, mask), file_name in train_loader:
print(input.shape)
print(mask.shape)
img = helpers.array_to_img(np.expand_dims(input.squeeze(), 2))
# 将gt反one-hot回去以便进行可视化
palette = [[0, 0, 0], [246, 16, 16], [16, 136, 246]]
gt = helpers.onehot_to_mask(np.array(mask.squeeze()).transpose(1, 2, 0), palette)
gt = helpers.array_to_img(gt)
# cv2.imshow('img GT', np.uint8(np.hstack([img, gt])))
cv2.imshow('img GT', np.uint8(gt))
cv2.waitKey(1000)
可视化的结果如下

4.2 模型设计
直接用经典的U-Net作为演示模型。注意输入的图像是1个通道,输出是3个通道。
# /networks/u_net.py
from networks.custom_modules.basic_modules import *
from utils.misc import initialize_weights
class Baseline(nn.Module):
def __init__(self, img_ch=1, num_classes=3, depth=2):
super(Baseline, self).__init__()
chs = [64, 128, 256, 512, 512]
self.maxpool = nn.MaxPool2d(kernel_size=2, stride=2)
self.enc1 = EncoderBlock(img_ch, chs[0], depth=depth)
self.enc2 = EncoderBlock(chs[0], chs[1], depth=depth)
self.enc3 = EncoderBlock(chs[1], chs[2], depth=depth)
self.enc4 = EncoderBlock(chs[2], chs[3], depth=depth)
self.enc5 = EncoderBlock(chs[3], chs[4], depth=depth)
self.dec4 = DecoderBlock(chs[4], chs[3])
self.decconv4 = EncoderBlock(chs[3] * 2, chs[3])
self.dec3 = DecoderBlock(chs[3], chs[2])
self.decconv3 = EncoderBlock(chs[2] * 2, chs[2])
self.dec2 = DecoderBlock(chs[2], chs[1])
self.decconv2 = EncoderBlock(chs[1] * 2, chs[1])
self.dec1 = DecoderBlock(chs[1], chs[0])
self.decconv1 = EncoderBlock(chs[0] * 2, chs[0])
self.conv_1x1 = nn.Conv2d(chs[0], num_classes, 1, bias=False)
initialize_weights(self)
def forward(self, x):
# encoding path
x1 = self.enc1(x)
x2 = self.maxpool(x1)
x2 = self.enc2(x2)
x3 = self.maxpool(x2)
x3 = self.enc3(x3)
x4 = self.maxpool(x3)
x4 = self.enc4(x4)
x5 = self.maxpool(x4)
x5 = self.enc5(x5)
# decoding + concat path
d4 = self.dec4(x5)
d4 = torch.cat((x4, d4), dim=1)
d4 = self.decconv4(d4)
d3 = self.dec3(d4)
d3 = torch.cat((x3, d3), dim=1)
d3 = self.decconv3(d3)
d2 = self.dec2(d3)
d2 = torch.cat((x2, d2), dim=1)
d2 = self.decconv2(d2)
d1 = self.dec1(d2)
d1 = torch.cat((x1, d1), dim=1)
d1 = self.decconv1(d1)
d1 = self.conv_1x1(d1)
return d1
if __name__ == '__main__':
# from torchstat import stat
import torch
以上是关于附源码医学图像分割入门实践的主要内容,如果未能解决你的问题,请参考以下文章