五自适应阈值与轮廓检测应用实例——细胞计数统计
Posted 舞刀书生
tags:
篇首语:本文由小常识网(cha138.com)小编为大家整理,主要介绍了五自适应阈值与轮廓检测应用实例——细胞计数统计相关的知识,希望对你有一定的参考价值。
教程汇总:python基础入门系列
假设有这样一个任务,通过显微镜拍下的某藻类细胞的玻片图,需要统计图中细胞个数。如果每天都要人工处理这项工作,那么将会浪费大量的时间与人力,我们既然学习了python那么自然可以想办法自动化高效率(偷懒)的完成这项任务。显微镜下藻类细胞图如下:

图像预处理
这种图是必须要进行处理过的,滤除掉无关多余信息才能更有针对性的进行细胞检测任务,首先想到的便是灰度,二值化处理。上代码:
import cv2
src=cv2.imread('cell0.jpg')
# 鉴于后续的图像处理操作可能会破坏原图,我们这里另拷贝一份src来用
img = src.copy()
# 转灰度图
gray = cv2.cvtColor(img,cv2.COLOR_BGR2GRAY)
# 均值模糊,除去噪音
gray = cv2.blur(gray, (2, 2))
# 二值化
retval,threshold = cv2.threshold(gray,150,255,cv2.THRESH_BINARY)
cv2.imshow("gray", gray)
cv2.imshow("threshold", threshold)
cv2.waitKey(0)
cv2.destroyAllWindows()
处理结果如下图:

物体轮廓检测
代表细胞的黑点已经处理出来了,那么就要进行轮廓检测与统计了。这里我们会用到cv2.findContours()函数来查找检测物体的轮廓。需要注意的是cv2.findContours()函数接受的参数为二值图,即黑白的(不是灰度图),所以使用该方法前二值化处理是比不可少的。在之前二值化处理代码基础上增加轮廓检测代码:
contours,hierarchy = cv2.findContours(threshold,cv2.RETR_LIST,cv2.CHAIN_APPROX_SIMPLE)
注意,findcontours函数会“原地”修改输入的图像。这一点可通过下面的语句验证:
cv2.imshow("threshold", threshold)
contours,hierarchy = cv2.findContours(threshold,cv2.RETR_LIST,cv2.CHAIN_APPROX_SIMPLE)
cv2.imshow("threshold2", threshold)
cv2.findContours()函数
函数的原型为
cv2.findContours(image, mode, method[, contours[, hierarchy[, offset ]]])
opencv2返回两个值:contours,hierarchy。注:opencv3会返回三个值,分别是img, countours, hierarchy
参数
-
第一个参数是寻找轮廓的图像;
-
第二个参数表示轮廓的检索模式,有四种(本文介绍的都是新的cv2接口):
cv2.RETR_EXTERNAL 表示只检测外轮廓
cv2.RETR_LIST 检测的轮廓不建立等级关系
cv2.RETR_CCOMP 建立两个等级的轮廓,上面的一层为外边界,里面的一层为内孔的边界信息。如果内孔内还有一个连通物体,这个物体的边界也在顶层。
cv2.RETR_TREE 建立一个等级树结构的轮廓。 -
第三个参数method为轮廓的近似办法
cv2.CHAIN_APPROX_NONE 存储所有的轮廓点,相邻的两个点的像素位置差不超过1,即max(abs(x1-x2),abs(y2-y1))==1
cv2.CHAIN_APPROX_SIMPLE 压缩水平方向,垂直方向,对角线方向的元素,只保留该方向的终点坐标,例如一个矩形轮廓只需4个点来保存轮廓信息
cv2.CHAIN_APPROX_TC89_L1,CV_CHAIN_APPROX_TC89_KCOS 使用teh-Chinl chain 近似算法
返回值
cv2.findContours()函数返回两个值,一个是轮廓本身(contours),还有一个是每条轮廓对应的属性(hierarchy)。
- contours:
我们主要用到第一个返回值,该参数是一个list,list中每个元素都是图像中的一个轮廓,用numpy中的ndarray表示。这个概念非常重要。在下面drawContours中会看见。通过print (type(contours)) print (type(contours[0])) print (len(contours)) - hierarchy:
此外,该函数还可返回一个可选的hiararchy结果,这是一个ndarray,其中的元素个数和轮廓个数相同,每个轮廓contours[i]对应4个hierarchy元素hierarchy[i][0] ~hierarchy[i][3],分别表示后一个轮廓、前一个轮廓、父轮廓、内嵌轮廓的索引编号,如果没有对应项,则该值为负数。
通过
可以看出,hierarchy本身包含两个ndarray,每个ndarray对应一个轮廓,每个轮廓有四个属性。print (type(hierarchy)) print (hierarchy.ndim) print (hierarchy[0].ndim) print (hierarchy.shape)
轮廓的绘制
我们需要在原图上绘制出检测出来的轮廓,方便验证检测出来的轮廓是否满足我们的要求。OpenCV中可以使用cv2.drawContours()函数在图像上绘制轮廓。
函数原型如下:
cv2.drawContours(image, contours, contourIdx, color[, thickness[, lineType[, hierarchy[, maxLevel[, offset ]]]]])
- 第一个参数是指明在哪幅图像上绘制轮廓;
- 第二个参数是轮廓本身,在Python中是一个list。
- 第三个参数指定绘制轮廓list中的哪条轮廓,如果是-1,则绘制其中的所有轮廓。后面的参数很简单。其中thickness表明轮廓线的宽度,如果是-1(cv2.FILLED),则为填充模式。绘制参数将在以后独立详细介绍。
完整代码如下:
import cv2
#打开图片
src=cv2.imread('cell0.jpg')
# 鉴于后续的图像处理操作可能会破坏原图,我们这里另拷贝一份src来用
img = src.copy()
# 转灰度图
gray = cv2.cvtColor(img,cv2.COLOR_BGR2GRAY)
# 均值模糊,除去噪音
gray = cv2.blur(gray, (2, 2))
# 二值化
retval,threshold = cv2.threshold(gray,150,255,cv2.THRESH_BINARY)
#轮廓检测
contours,hierarchy = cv2.findContours(threshold,cv2.RETR_LIST,cv2.CHAIN_APPROX_SIMPLE)
# 标记圆点轮廓
cv2.drawContours(img,contours,-1,(255,0,0,),1)
# 标记圆点序号
n = 0
for cnt in contours:
font = cv2.FONT_HERSHEY_PLAIN
cv2.putText(img,str(n+1),(contours[n][0][0][0],contours[n][0][0][1]),font,1,(200,200,0),1,cv2.LINE_AA)
n += 1
# 显示总个数
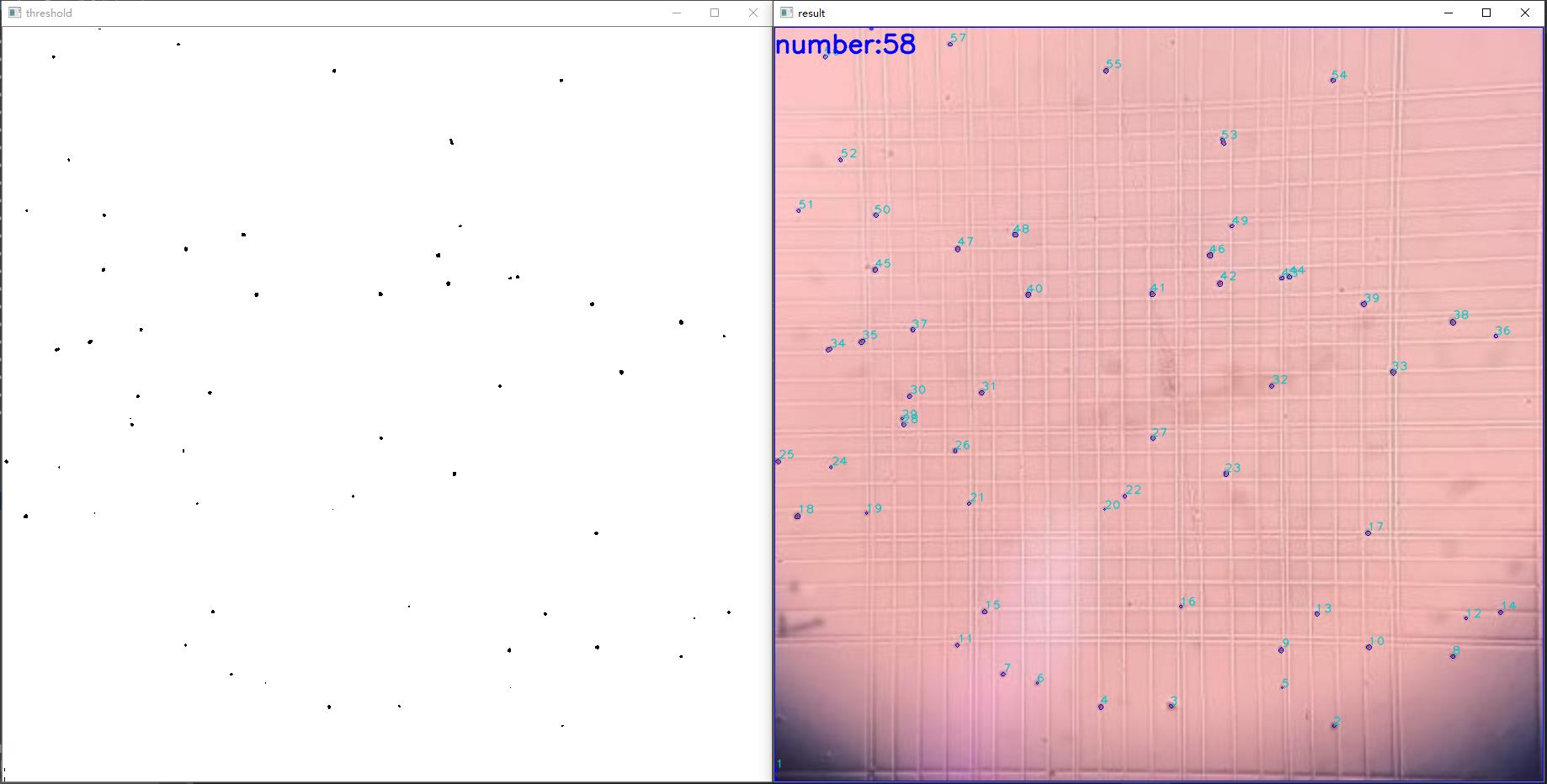
font = cv2.FONT_HERSHEY_SIMPLEX
cv2.putText(img,'number:'+str(len(contours)-1),(0,30),font,1,(255,0,0),2,cv2.LINE_AA)
cv2.imshow("source",src)
cv2.imshow("result", img)
cv2.waitKey(0)
cv2.destroyAllWindows()
结果如下图:

自适应阈值优化
上面的处理我们只是用了简单阈值处理也足够了,可如果是下面这张图呢:

可以发现图中边缘有大面积深色部分,简单阈值的话势必会导致出现误判。通过前面 三、OpenCV图像的预处理——二值化与自适应阈值的章节学习,我们想到了采用自适应阈值来处理。
只需要上面代码中二值化处理代码改为下句:
threshold = cv2.adaptiveThreshold(gray, 255, cv2.ADAPTIVE_THRESH_GAUSSIAN_C, cv2.THRESH_BINARY, 21, 35)
结果如图:
以上是关于五自适应阈值与轮廓检测应用实例——细胞计数统计的主要内容,如果未能解决你的问题,请参考以下文章