CT片居然可以这么玩:用头部CT断层扫描片复原三维头像
Posted AI科技大本营
tags:
篇首语:本文由小常识网(cha138.com)小编为大家整理,主要介绍了CT片居然可以这么玩:用头部CT断层扫描片复原三维头像相关的知识,希望对你有一定的参考价值。


作者:天元浪子
来源:Python作业辅导员
前言
CT是现代医学影像的主力设备,寻常百姓并不陌生。通常,一张CT片由多张连续断层扫描的图像组成。在医生眼中,CT片展示了人体器官的形态和性质,是判断病人健康状况的重要依据。对于普通人而言,CT片则像天书,几无大用。不过呢,总有不甘寂寞喜欢折腾的程序员,把看似无用的CT片玩出了新花样——用头部CT断层扫描片成功复原出了逼真的三维头像。

CT是如何变成三维头像的呢?
说白了,其实很简单,只需要3步:
准备一套头部CT断层扫描片。怎么准备?要么去网上找(我就是从网上下载的),要么自己扫描胶片。
按照断层顺序逐张读入图片,并垂直堆叠成一个numpy数组。
导入3D工具库WxGL,调用capsule函数,绘制三维头像。
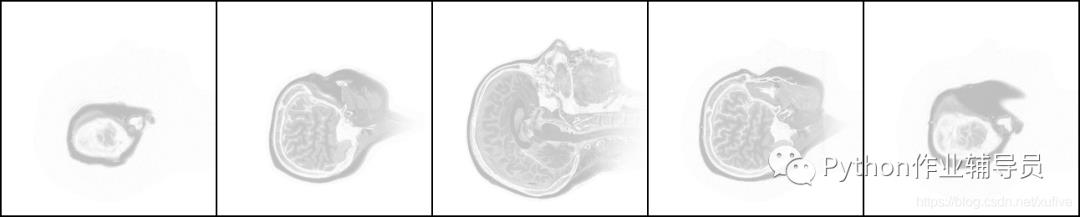
我手头这套头部CT断层扫描片共有109张,是侧卧位的水平断层扫描,从一侧开始至另一侧结束。下图从左至右分别是第11张、第21张、第55张、第89张、第99张。不难看出,中间的一张面积最大,是鼻梁正中的断层图片,鼻尖朝向图片上方,越往两侧的图片,面积越小。
有了CT断层扫描片,接下来就是打开文件读取数据了。做这一步之前需要先导入pillow模块和numpy模块。假定CT文件保存在:
D:\\XufiveGithub\\wxgl\\res\\headCT这个路径下,每个文件名都以head+序号的方式命名,下面是读取数据的代码。
>>> from PIL import Image
>>> import numpy as np
>>> base_dir = r'D:\\XufiveGithub\\wxgl\\res\\headCT'
>>> data = list()
>>> for i in range(109):
im = np.array(Image.open(r'%s\\head%d.png'%(base_dir, i)))
data.append(im)
>>> data = np.stack(data, axis=0)
其实,读取数据的代码可以写得更简洁,使用列表推导式,只用一行就可以了。
>>> data = np.stack([np.array(Image.open(r'%s\\head%d.png'%(base_dir, i))) for i in range(109)], axis=0)
>>> data.shape
(109, 256, 256, 4)
从data.shape可以看出,总共有109个断层,每个断层分辨率是256x256像素,每个像素有RGBA四个通道。比较每个通道的数据,就会发现,每个断层的RGBA四个通道是完全相同的,这是胶片扫描的特点决定的。
三维重建的基本原理是滤除完全透明的像素,用具有相同透明度且位置相邻的像素构建surface,因此数组data中只有透明通道(A通道)是我们需要的。
data = data[...,3] # 提取透明通道
>>> data.shape
(109, 256, 256)
现在data变成一个三维数组了。数组的0轴,即第1张到第109张CT片的方向,对应着头像的左右;数组的1轴,即每张CT片自上而下的方向,对应着头像的前后;数组的2轴,即每张CT片从左至右的方向,对应着头像的上下。3D工具库WxGL习惯将数据数组的0轴视为高度方向,不做调整的话,绘制出的头像将是水平侧卧的,就像CT片展示的那样。为了让头像立起来,需要将头像的上下方对应的数组2轴翻转到0轴之后,再上下翻转。
>>> data = np.rollaxis(data, 2, 0) # 2轴翻转至0轴,让头像立起来(倒立)
>>> data = np.flipud(data) # 上下翻转,头像正立
>>> data.shape
(256, 109, 256)
至此,数据处理算是完成了,不过,这些数据还没有空间定位,只是保持了相对的位置关系,我们需要给数据限定一个空间位置。由于CT片没有给出头部尺寸和分层厚度,只能按照通常的头像比例,给出头像在xyz轴上的空间范围。
x = np.linspace(-1, 1, data.shape[2]) # 头像前后范围:[-1, 1]
y = np.linspace(-0.65, 0.65, data.shape[1]) # 头像左右范围:[-0.65, 0.65]
z = np.linspace(-1, 1, data.shape[0]) # 头像高度范围:[-1, 1]
另外,还要滤除几乎完全透明的那些像素。我们暂且以最大值的1/8作为阈值,调整该阈值将会得到不同的重建效果。
level = data.max()/8 # 忽略低于此阈值的数据
万事俱备,只欠东风。是时候让wxgl一显身手了。
plt.capsule(data, x=x, y=y, z=z, level=level, color='#EEE9D4')
plt.show()
不要惊讶,只需要两行代码,一个逼真的三维头像呈现在眼前。这一切,仅仅依赖一套CT断层扫描片和WxGL这个3D工具库。

刚刚发布的0.7.3版3D工具库WxGL,新增了几个有趣的功能,其中之一就是刚刚用到的capsule函数。capsule意为胶囊,顾名思义,capsule函数可以从三维数据中找出等值面,并尝试构建出囊性结构(如果存在的话)。WxGL的安装非常简单,直接使用pip命令即可。如果在安装和使用WxGL的过程中遇到问题,请在文后留言,也可以直接到我的GitHub反馈问题。
pip install wxgl
最后,附上用头部CT断层扫描片复原三维头像的完整代码。
import numpy as np
from PIL import Image
from wxgl import wxplot as plt
base = r'D:\\XufiveGithub\\wxgl\\res\\headCT'
data = np.stack([np.array(Image.open(r'%s\\head%d.png'%(base, i))) for i in range(109)], axis=0)
data = data[...,3] # 提取透明通道
data = np.rollaxis(data, 2, 0) # 2轴翻转至0轴,让头像立起来(倒立)
data = np.flipud(data) # 上下翻转,头像正立
x = np.linspace(-1, 1, data.shape[2]) # 头像前后范围:[-1, 1]
y = np.linspace(-0.65, 0.65, data.shape[1]) # 头像左右范围:[-0.65, 0.65]
z = np.linspace(-1, 1, data.shape[0]) # 头像高度范围:[-1, 1]
level = data.max()/8 # 忽略低于此阈值的数据
plt.capsule(data, x=x, y=y, z=z, level=level, color='#EEE9D4')
plt.show(rotate=-1)
下图直观显示了不同的阈值对于三维重建结果的影响。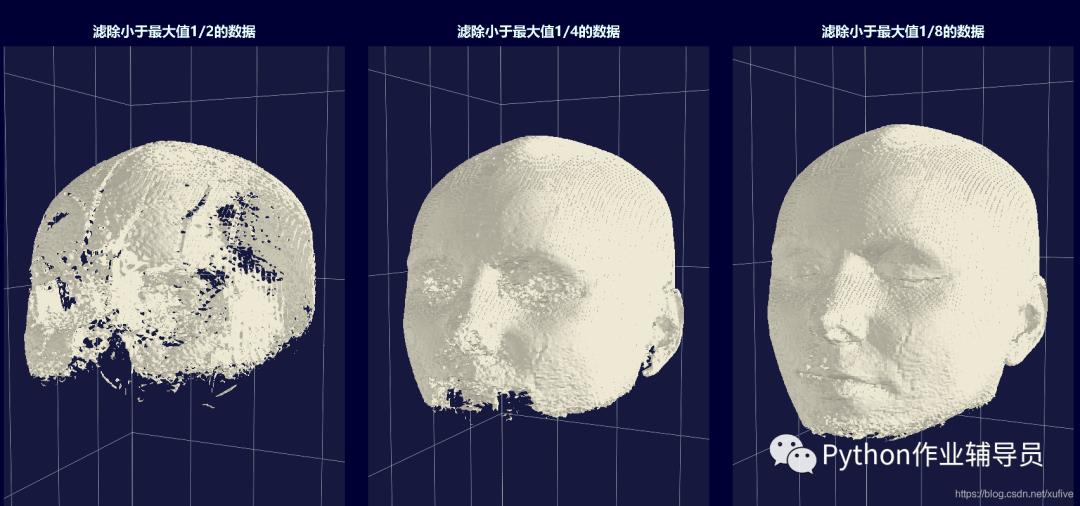

END


往
期
回
顾
新闻
技术
技术
新闻

分享

点收藏

点点赞

点在看
以上是关于CT片居然可以这么玩:用头部CT断层扫描片复原三维头像的主要内容,如果未能解决你的问题,请参考以下文章