遗传算法GA--TSP旅行商问题(Python)
Posted 桃陉
tags:
篇首语:本文由小常识网(cha138.com)小编为大家整理,主要介绍了遗传算法GA--TSP旅行商问题(Python)相关的知识,希望对你有一定的参考价值。
一.基础介绍
遗传算法的来源、定义、特点见之前的文章【遗传算法GA】–计算函数最值(Python)。
下面我们先来看本次需要实现的内容:我们随机生成一些城市的坐标,然后找一条最短路径通过所有城市。
最重要的还是对染色体DNA的编码以及适应度函数的确定。对于本题来说可以先将所以城市进行编号,然后对这些编号进行排序,排好的顺序就是旅行的路线。对于适应度函数来说就是将路程加起来,总路程最小,适应度越高。
参数:
| 参数名称 | 含义 |
|---|---|
| citys | 城市个数 |
| pc | 交叉概率 |
| pm | 变异概率 |
| popsize | 种群规模 |
| iternum | 迭代次数 |
| pop | 种群 |
| city_position | 城市坐标 |
二.分布实现
∙
\\bullet
∙ 参数:
citys = 20 #染色体DNA长度
pc = 0.1 #交叉概率
pm = 0.02 #变异概率
popsize = 500 #种群规模
iternum = 100 #迭代次数
GA类:
∙
\\bullet
∙ 将种群中排好的序列横纵坐标分别提取出来transltaeDNA函数:参数DNA为种群pop,参数city_position为所有城市坐标
def translateDNA(self,DNA,city_position):
#生成等长的空列表
lineX = np.empty_like(DNA,dtype=np.float64)
lineY = np.empty_like(DNA,dtype=np.float64)
#将下标和值同时提取出来
for i,d in enumerate(DNA):
city_coord = city_position[d]
lineX[i,:] = city_coord[:,0]
lineY[i,:] = city_coord[:,1]
return lineX,lineY
∙
\\bullet
∙ 求适应度函数getFiness:参数lineX、lineY分别为城市坐标,返回fitness为每个个体的适应度,totalDis为每个个体的总路程。
def getFitness(self,lineX,lineY):
totalDis = np.empty((lineX.shape[0],),dtype=np.float64)
for i,(xval,yval) in enumerate(zip(lineX,lineY)):
totalDis[i]=np.sum(np.sqrt(np.square(np.diff(xval)) + np.square(np.diff(yval))))
fitness = np.exp(self.citys*2/totalDis)
return fitness,totalDis
∙
\\bullet
∙ 选择函数selection:参数fitness为适应度,选择适应度更高的个体。
def selection(self,fitness):
idx = np.random.choice(np.arange(self.popsize),size=self.popsize,replace=True,p=fitness/fitness.sum())
return self.pop[idx]
∙
\\bullet
∙ 交叉函数selection:参数parent为父本中一个个体,pop为种群。
交叉规则:在交叉概率内随机选择种群中一个体,随机选择一些位置,将这些位置的数提取出来放到数组前面,然后将母本中除这些数之外的数按顺序放入数组后面,组成新个体。
def crossover(self,parent,pop):
if np.random.rand() < self.pc:
i = np.random.randint(0, self.popsize, size=1) #随机选取一个个体进行交换
cross_points = np.random.randint(0, 2, self.citys).astype(np.bool) #随机选择个体中的一些位置
keep_city = parent[~cross_points] #将parent中False的位置返给keep_city
swap_city = pop[i, np.isin(pop[i].ravel(), keep_city, invert=True)] #将keep_city中没有出现的数赋给swap_city
parent[:] = np.concatenate((keep_city, swap_city)) #拼接形成新个体
return parent
∙
\\bullet
∙ 变异函数mutation:在变异范围内随机选取一个位置与下标位置的数进行互换。
def mutation(self,child):
for point in range(self.citys):
if np.random.rand()<self.pm:
swap_point = np.random.randint(0,self.citys)
swapa,swapb = child[point],child[swap_point]
child[point],child[swap_point] = swapb,swapa
return child
∙
\\bullet
∙ 进化函数evolve:调用交叉函数和变异函数。
def evolve(self,fitness):
pop = self.selection(fitness)
pop_copy = pop.copy()
for parent in pop:
child = self.crossover(parent,pop_copy)
child = self.mutation(child)
parent[:] = child
self.pop = pop
TSP类:
∙
\\bullet
∙ 构造函数:随机生成城市坐标
def __init__(self,citys):
#生成每个城市的横纵坐标
self.city_position = np.random.rand(citys,2)
plt.ion()
∙
\\bullet
∙ 绘图函数plotting:参数lx、ly为城市横纵坐标,total_d最优路线。
def plotting(self,lx,ly,total_d):
plt.cla()
plt.scatter(self.city_position[:, 0].T, self.city_position[:, 1].T, s=100, c='k') #画散点图
plt.plot(lx.T, ly.T, 'r-') #连线
plt.text(-0.05, -0.05, "Total distance=%.2f" % total_d, fontdict={'size': 20, 'color': 'red'})
plt.xlim((-0.1, 1.1))
plt.ylim((-0.1, 1.1))
plt.pause(0.01)
∙
\\bullet
∙ 主函数:
if __name__=='__main__':
ga = GA(citys=citys,pc=pc,pm=pm,popsize=popsize)
env = TSP(citys=citys)
for gen in range(iternum):
lx,ly = ga.translateDNA(ga.pop,env.city_position)
fitness,total_distance = ga.getFitness(lx,ly)
ga.evolve(fitness)
best_idx = np.argmax(fitness) #最优解的下标
print("Gen:", gen," | best fit: %.2f"%fitness[best_idx],)
env.plotting(lx[best_idx],ly[best_idx],total_distance[best_idx])
plt.ioff()
plt.show()
三.完整代码
import numpy as np
import matplotlib.pyplot as plt
#参数
citys = 20 #染色体DNA长度
pc = 0.1 #交叉概率
pm = 0.02 #变异概率
popsize = 500 #种群规模
iternum = 100 #迭代次数
class GA(object):
def __init__(self,citys,pc,pm,popsize,):
self.citys = citys
self.pc = pc
self.pm = pm
self.popsize = popsize
#vstck纵向拼接数组,permutaion将数字0-(city-1)进行随机排序
#生成种群,dna序列为0到city的随机序列
self.pop = np.vstack([np.random.permutation(citys) for _ in range(popsize)])
#将种群中排好的序列横纵坐标分别提取出来
def translateDNA(self,DNA,city_position):
#生成等长的空列表
lineX = np.empty_like(DNA,dtype=np.float64)
lineY = np.empty_like(DNA,dtype=np.float64)
#将下标和值同时提取出来
for i,d in enumerate(DNA):
city_coord = city_position[d]
lineX[i,:] = city_coord[:,0]
lineY[i,:] = city_coord[:,1]
return lineX,lineY
def getFitness(self,lineX,lineY):
totalDis = np.empty((lineX.shape[0],),dtype=np.float64)
for i,(xval,yval) in enumerate(zip(lineX,lineY)):
totalDis[i]=np.sum(np.sqrt(np.square(np.diff(xval)) + np.square(np.diff(yval))))
fitness = np.exp(self.citys*2/totalDis)
return fitness,totalDis
def selection(self,fitness):
idx = np.random.choice(np.arange(self.popsize),size=self.popsize,replace=True,p=fitness/fitness.sum())
return self.pop[idx]
def crossover(self,parent,pop):
if np.random.rand() < self.pc:
i = np.random.randint(0, self.popsize, size=1) #随机选取一个个体进行交换
cross_points = np.random.randint(0, 2, self.citys).astype(np.bool) #随机选择个体中的一些位置
keep_city = parent[~cross_points] #将parent中False的位置返给keep_city
swap_city = pop[i, np.isin(pop[i].ravel(), keep_city, invert=True)] #将keep_city中没有出现的数赋给swap_city
parent[:] = np.concatenate((keep_city, swap_city)) #拼接形成新个体
return parent
def mutation(self,child):
for point in range(self.citys):
if np.random.rand()<self.pm:
swap_point = np.random.randint(0,self.citys)
swapa,swapb = child[point],child[swap_point]
child[point],child[swap_point] = swapb,swapa
return child
def evolve(self,fitness):
pop = self.selection(fitness)
pop_copy = pop.copy()
for parent in pop:
child = self.crossover(parent,pop_copy)
child = self.mutation(child)
parent[:] = child
self.pop = pop
class TSP(object):
def __init__(self,citys):
#生成每个城市的横纵坐标
self.city_position = np.random.rand(citys,2)
plt.ion()
def plotting(self,lx,ly,total_d):
plt.cla()
plt.scatter(self.city_position[:, 0].T, self.city_position[:, 1].T, s=100, c='k') #画散点图
plt.plot(lx.T, ly.T, 'r-') #连线
plt.text(-0.05, -0.05, "Total distance=%.2f" % total_d, fontdict={'size': 20, 'color': 'red'})
plt.xlim((-0.1, 1.1))
plt.ylim((-0.1, 1.1))
plt.pause(0.01)
if __name__=='__main__':
ga = GA(citys=citys,pc=pc,pm=pm,popsize=popsize)
env = TSP(citys=citys)
for gen in range(iternum):
lx,ly = ga.translateDNA(ga.pop,env.city_position)
fitness,total_distance = ga.getFitness(lx,ly)
ga.evolve(fitness)
best_idx = np.argmax(fitness) #最优解的下标
print("Gen:", gen," | best fit: %.2f"%fitness[best_idx],)
env.plotting(lx[best_idx],ly[best_idx],total_distance[best_idx])
plt.ioff()
plt.show()
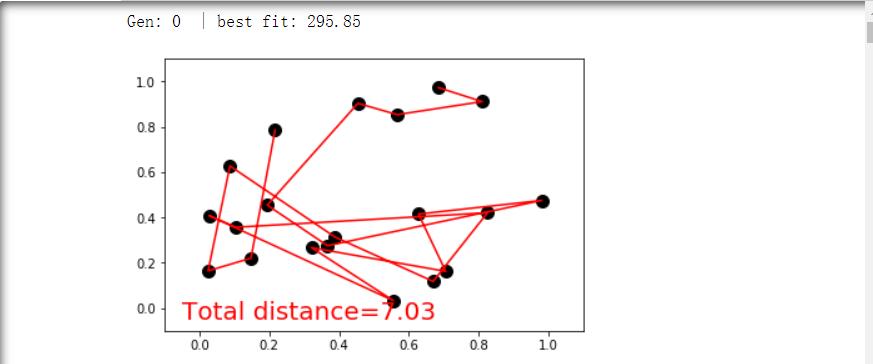
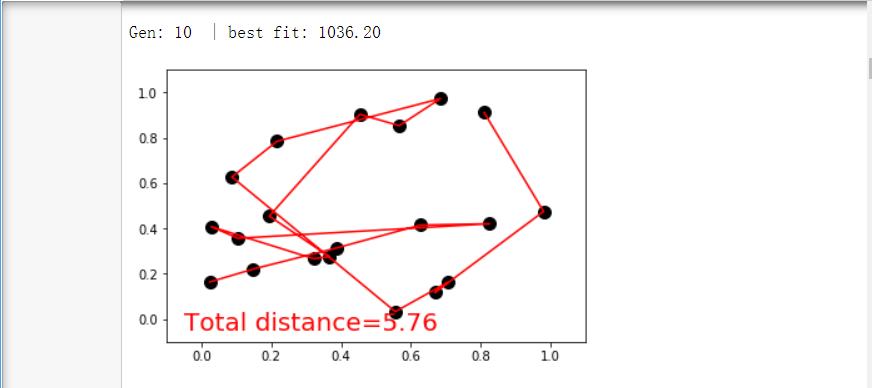
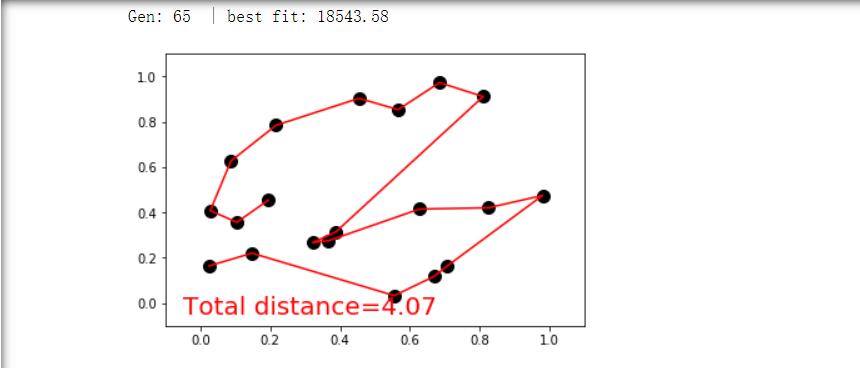
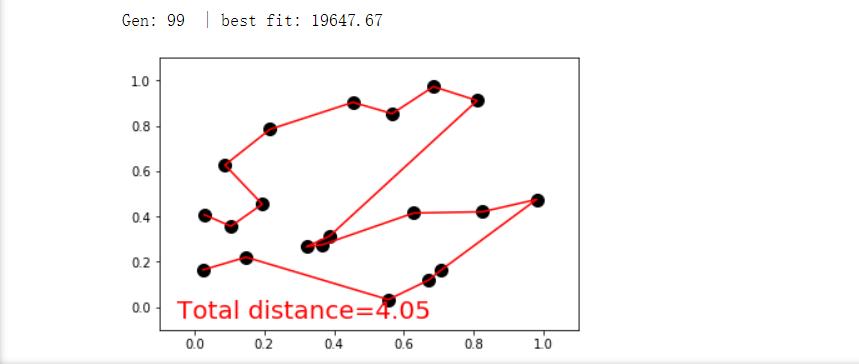
四.结果截图
初始情况(进行100次迭代):
以上是关于遗传算法GA--TSP旅行商问题(Python)的主要内容,如果未能解决你的问题,请参考以下文章
TSP基于matlab遗传算法求解旅行商问题含Matlab源码 1337期
MTSP基于matlab遗传算法求解多旅行商问题含Matlab源码 1325期
MTSP基于matlab遗传算法求解多旅行商问题含Matlab源码 1339期