R 语言 4.2.2安装 WGCNA
Posted LiQiang33
tags:
篇首语:本文由小常识网(cha138.com)小编为大家整理,主要介绍了R 语言 4.2.2安装 WGCNA相关的知识,希望对你有一定的参考价值。
文章目录
1 WGCNA库介绍
-
WGCNA是用于加权相关网络分析的R包,相关网络越来越多地用于生物信息学应用 -
加权基因共表达网络分析是一种系统生物学方法,用于描述微阵列样本中基因之间的相关性模式 -
加权相关网络分析(WGCNA)可用于发现高度相关基因的簇(模块),使用模块特征基因或模块内中枢基因总结此类簇,可以将模块彼此关联并与外部样本特征关联(使用特征基因网络方法),以及计算模块成员度量 -
相关网络促进了基于网络的基因筛选方法,可用于识别候选生物标志物或治疗靶点 -
这些方法已成功应用于各种生物学背景,例如癌症、小鼠遗传学、酵母遗传学和脑成像数据分析
-
WGCNA是R函数的综合集合,用于执行加权相关网络分析的各个方面。该软件包包括网络构建、模块检测、基因选择、拓扑特性计算、数据模拟、可视化以及与外部软件接口的功能
2 安装
踩坑
直接安装WGCNA,这样的方式会导致一些依赖包安装失败,导致WGCNA运行不起来;
install.packages('WGCNA')
报错信息:
Warning in install.packages :
没有'‘impute’, ‘preprocessCore’, ‘AnnotationDbi’'这种相依关系
trying URL 'https://cran.rstudio.com/bin/windows/contrib/4.2/WGCNA_1.71.zip'
Content type 'application/zip' length 3254830 bytes (3.1 MB)
downloaded 3.1 MB
---------------------------------------------------
> library(WGCNA)
载入需要的程辑包:dynamicTreeCut
载入需要的程辑包:fastcluster
载入程辑包:‘fastcluster’
The following object is masked from ‘package:stats’:
hclust
Error: package or namespace load failed for ‘WGCNA’ in loadNamespace(i, c(lib.loc, .libPaths()), versionCheck = vI[[i]]):
不存在叫‘AnnotationDbi’这个名字的程辑包
>
> library(GO.db)
Error: 找不到‘GO.db’所需要的程辑包‘AnnotationDbi’
> library(WGCNA)
Error: package or namespace load failed for ‘WGCNA’ in loadNamespace(i, c(lib.loc, .libPaths()), versionCheck = vI[[i]]):
不存在叫‘AnnotationDbi’这个名字的程辑包
对相应的依赖单独安装之后,仍然出现相关的问题,而且出现版本不匹配的问题,不能解决问题
还得是官方文档
link: https://horvath.genetics.ucla.edu/html/CoexpressionNetwork/Rpackages/WGCNA
由于作者基本不会R语言,所以不得不查找官方的文档,直接搜索WGCNA会有很多资料,找到官方文档
- 自动安装方法(本人尝试后失败)
install.packages("BiocManager")
BiocManager::install("WGCNA")
如果
BiocManager已经安装的情况下,第一个命令将被跳过
- 手动安装相关的包
install.packages(c("matrixStats", "Hmisc", "splines", "foreach", "doParallel", "fastcluster", "dynamicTreeCut", "survival", "BiocManager"))
BiocManager::install(c("GO.db", "preprocessCore", "impute"));
如果版本较老没有BiocManager,使用:
install.packages(c("matrixStats", "Hmisc", "splines", "foreach", "doParallel", "fastcluster", "dynamicTreeCut", "survival"))
source("http://bioconductor.org/biocLite.R")
biocLite(c("GO.db", "preprocessCore", "impute"))
这样安装我出现的问题
> library(WGCNA)
载入需要的程辑包:dynamicTreeCut
载入需要的程辑包:fastcluster
载入程辑包:‘fastcluster’
The following object is masked from ‘package:stats’:
hclust
Error: package or namespace load failed for ‘WGCNA’ in loadNamespace(j <- i[[1L]], c(lib.loc, .libPaths()), versionCheck = vI[[j]]):
不存在叫‘impute’这个名字的程辑包
问题原因:自从R版本 2.14.0起,CRAN已经将impute撤回,需要通过Biocondutor获取,在R shell中运行
source("http://bioconductor.org/biocLite.R")
biocLite("impute")
# 或者
BiocManager::install(c("GO.db", "preprocessCore", "impute"))
我在 R-4.2.2 中安装暂时没有出现其他ERROR
完成后信息:
> source("http://bioconductor.org/biocLite.R")
Error: With R version 3.5 or greater, install Bioconductor packages using BiocManager; see https://bioconductor.org/install
> BiocManager::install(c("GO.db", "preprocessCore", "impute"))
'getOption("repos")' replaces Bioconductor standard repositories, see '?repositories'
for details
replacement repositories:
CRAN: https://cran.rstudio.com/
Bioconductor version 3.16 (BiocManager 1.30.19), R 4.2.2 (2022-10-31 ucrt)
Installing package(s) 'preprocessCore', 'impute'
trying URL 'https://bioconductor.org/packages/3.16/bioc/bin/windows/contrib/4.2/preprocessCore_1.60.1.zip'
Content type 'application/zip' length 169168 bytes (165 KB)
downloaded 165 KB
trying URL 'https://bioconductor.org/packages/3.16/bioc/bin/windows/contrib/4.2/impute_1.72.2.zip'
Content type 'application/zip' length 667393 bytes (651 KB)
downloaded 651 KB
程序包‘preprocessCore’打开成功,MD5和检查也通过
程序包‘impute’打开成功,MD5和检查也通过
参考
WGCNA官网: https://horvath.genetics.ucla.edu/html/CoexpressionNetwork/Rpackages/WGCNA/
WGCNA包安装FAQ:https://horvath.genetics.ucla.edu/html/CoexpressionNetwork/Rpackages/WGCNA/faq.html
Appendix
A. 安装R
下载 R-windows 以及 RStudio :
R语言Windows安装: https://blog.csdn.net/weixin_44524441/article/details/114130789
安装RStudio: https://blog.csdn.net/qq_34848334/article/details/119762092
B. 配置环境
修改R语言环境:
RStudio -> Tools -> Global Options 中换掉相关的环境,并重启RStudio
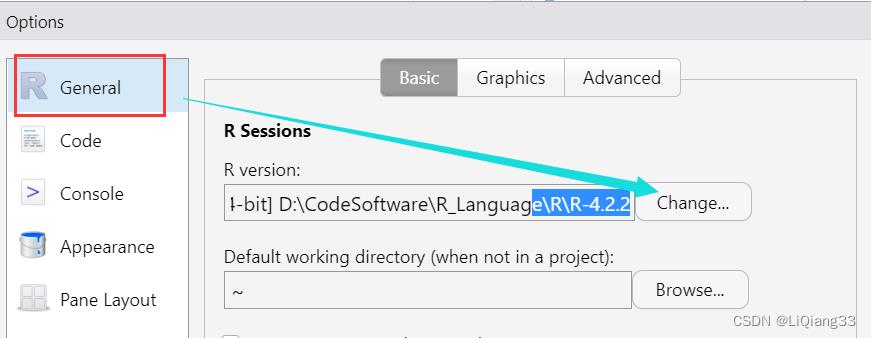
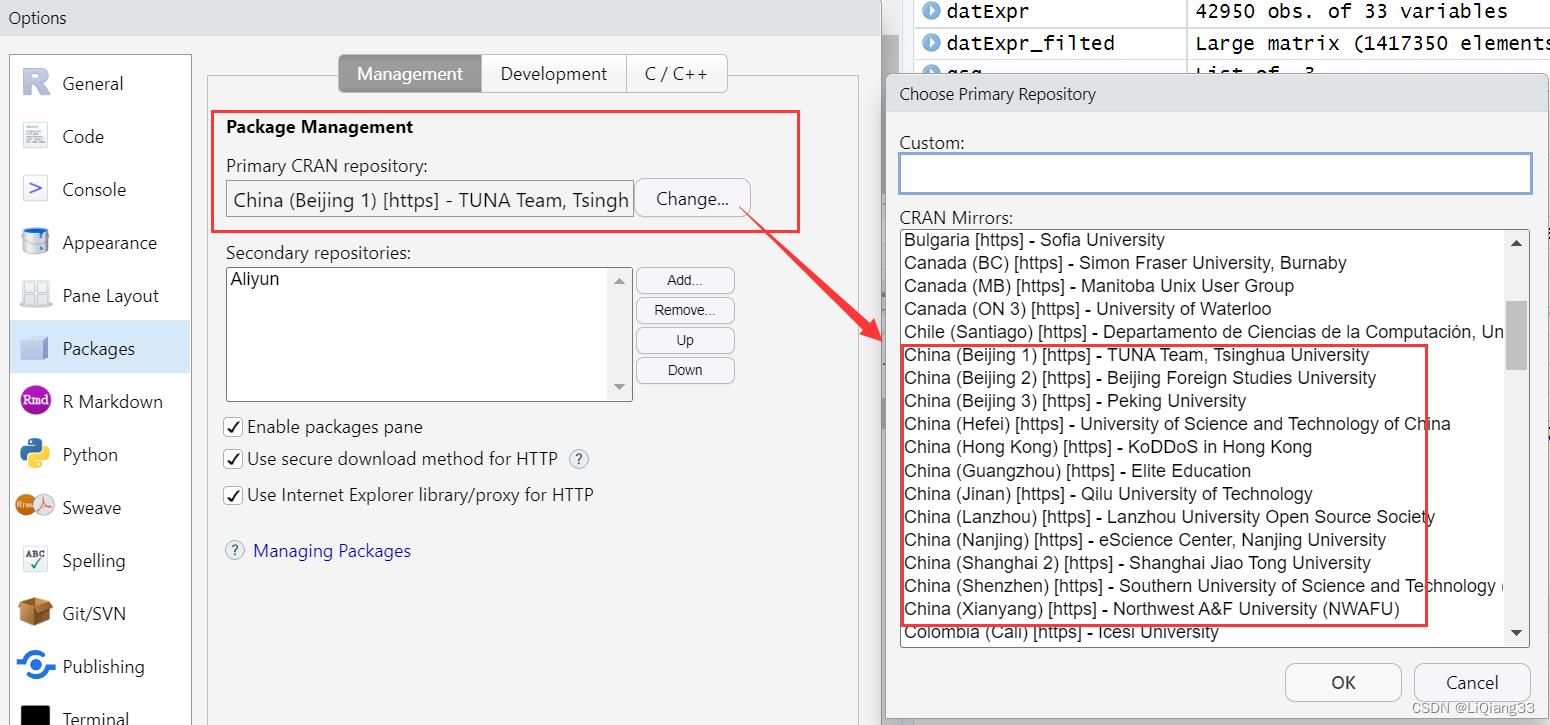
C. 修改镜像
RStudio -> Tools -> Global Options 中 Packages 选择中国镜像
以上是关于R 语言 4.2.2安装 WGCNA的主要内容,如果未能解决你的问题,请参考以下文章