数据挖掘专题 | GEO数据下载
Posted 生信控
tags:
篇首语:本文由小常识网(cha138.com)小编为大家整理,主要介绍了数据挖掘专题 | GEO数据下载相关的知识,希望对你有一定的参考价值。
对于GEO中的芯片数据来说,常包含两种类型的数据可供下载:
1、原始数据 -> Raw data 【Supplementary data files】
2、预处理数据 -> Series data 【Series Matirx Files】
以Affy HG-U133A平台数据的GSE994为例:
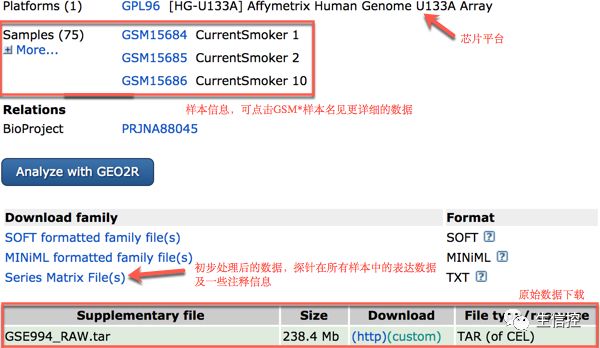
Series data和Raw data均可点击直接下载,但是有的数据集并未提供原始数据,如:

此时,只能下载预处理的数据,即Series data。当然,也有时候Supplementary下的数据并非最原始的数据...
In some cases there is a consensus in the field. For Affymetrix gene expression microarrays, "raw" files are so-called CEL files (a file format invented by Affymetrix) and "processed" data is normalized and quantified data, summarized at the probeset level.
小编在写这篇文章之前一直是手动下载的,因为反正要进到GEO数据库里看详细的数据信息,下载也是顺便的事情,并不繁琐。所以本次测试GEOquery是看看能不能更方便的得到更多的数据信息,或者有想要用R一次性解决所有问题的小伙伴呢?
在R中用GEOquery下载GEO数据库中的数据:
https://bioconductor.org/packages/release/bioc/html/GEOquery.html
The NCBI Gene Expression Omnibus (GEO) is a public repository of microarray data. Given the rich and varied nature of this resource, it is only natural to want to apply BioConductor tools to these data. GEOquery is the bridge between GEO and BioConductor.
### GEOquery 安装
source("https://bioconductor.org/biocLite.R")
biocLite("GEOquery") # 如果报错缺少依赖包时,安装一下缺的包即可
library(GEOquery)
### 使用
本次测试数据集GSE11675,包含HG_U95Av2芯片平台产生的6个样本数据,如下:
eList <- getGEO("GSE11675")
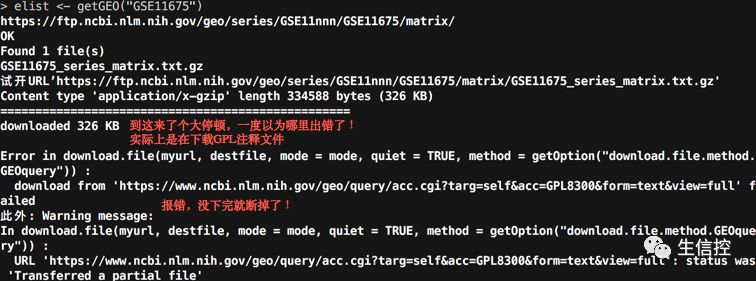
注意,对于该数据集,默认情况下,GSE11675_series_matrix.txt.gz 和 GPL8300.soft 文件会被下载在tempdir()目录下,可以通过destdir参数修改至自己预期的目录下,当然如果断开了,又重新运行命令的时候,可能会报如下错误:
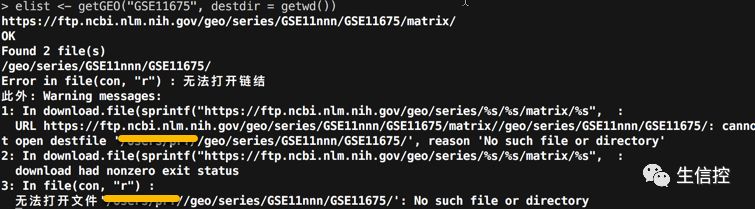
说实话,不知道问题出在哪里,换套数据也报错…【手残党一把辛酸泪啊!】
经过反复调试,目前已知的一个解决方案就是换个destdir目录(但凡用过的destdir就不要用了):
eList <- getGEO("GSE11675", destdir = "newdir")
# newdir换成自己的目录
GSE11675_series_matrix.txt.gz是下下来了,芯片注释soft文件还是下不动,网上给出的意见也很统一啊,手动下了再放到destdir目录下!
好吧,我用这个GEOquery就是为了下载方便,你让手动下...

在线下载注释文件:
https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GPL8300
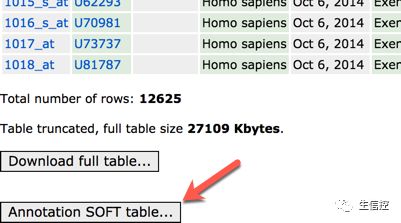
解压后将GPL8300.annot更名为GPL8300.soft,然后再放到destdir目录下:
eList <- getGEO("GSE11675", destdir = "newdir")
此时会自动识别并读取目录下的文件:
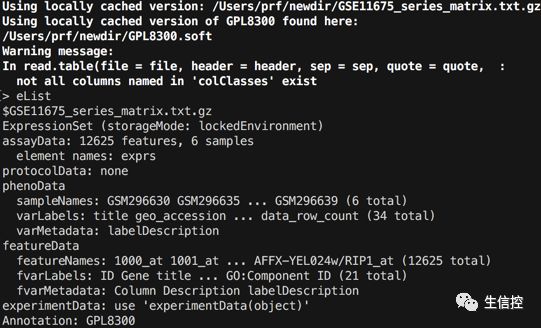
有个警告不管了(不同平台注释信息不同),算是读进来了!
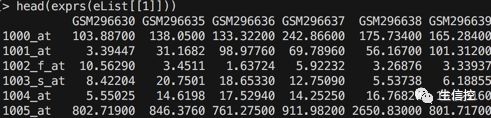
查看表达数据:
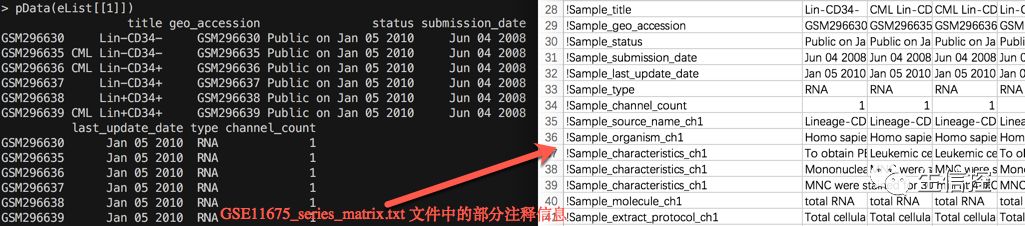
查看样本信息:
查看探针注释信息:
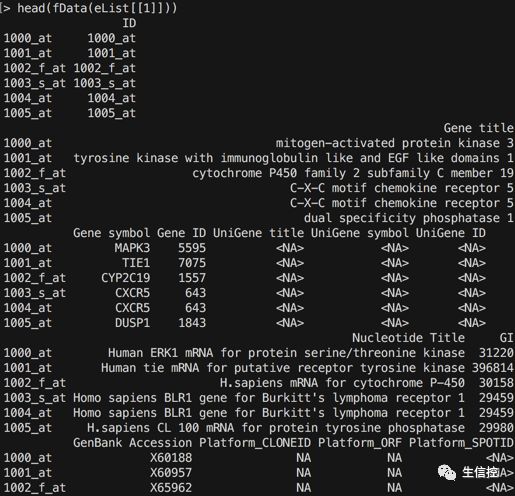
还可以下载原始数据:
gs = getGEOSuppFiles("GSE11675")
默认在当前目录下生成下载的原始数据文件,可使用baseDir参数修改路径。
如上,基本使用就是这些,其他用法详见:
https://bioconductor.org/packages/release/bioc/vignettes/GEOquery/inst/doc/GEOquery.html
综上,GEOquery的主要功能还是在R环境中实现GEO数据的下载,以及数据读入,更有利于流程化的数据分析。当然前提是要有一个较好的网络环境,如果像小编一样,数据下不动,建议还是手动下载吧,后续再读到R里进行整理分析!
关注生信控,解锁更多精彩!
以上是关于数据挖掘专题 | GEO数据下载的主要内容,如果未能解决你的问题,请参考以下文章