学徒数据挖掘第二期汇总之多分组基因注释代码大放送
Posted 生信菜鸟团
tags:
篇首语:本文由小常识网(cha138.com)小编为大家整理,主要介绍了学徒数据挖掘第二期汇总之多分组基因注释代码大放送相关的知识,希望对你有一定的参考价值。
学徒数据挖掘第二期目录如下:
如果仅仅是发一个目录,大家肯定觉得我们不走心,为了获得粉丝的喜爱,我仍然是安排学徒了一个数据挖掘任务。
本次需要完成的是非主流转录组数据分析图表,主流图表是:
表达矩阵的质控及可视化(PCA图及箱线图)
3大R包,edgeR, DEseq2, limma 做差异分析
差异分析的质控及可视化(热图及火山图)
差异基因的GO/KEGG数据库注释
表达矩阵及基因列表的GSEA分析
背景知识:
以前通常认为乳腺上皮包括两大类细胞:位于内层的分泌性管腔细胞(secretory luminal cells)和位于外层的基底/肌上皮细胞(basal/myoepithelial cells)
an inner layer of luminal cells that surround the lumen
an outer layer of myoepithelial cells that lie in a basal position adjacent to the basement membrane.
发表于2017年9月的NC,文章是;Construction of developmental lineage relationships in the mouse mammary gland by single-cell RNA profiling 的研究者系统性的跟踪检测了小鼠epithelial cells 的各种时期的单细胞转录组情况,包括:pre-puberty, puberty, adulthood and pregnancy, as well as at different points of the estrus cycle.
而我们今天要复现图表的文章(Cell Rep. 2016 Nov 15;) 提到正常的成年人的mammary gland 是由双层上皮细胞组成:
Bipotent and myoepithelial progenitors are prominent and unique components of the outer (basal) layer.
The inner (luminal) layer includes both luminal-restricted progenitors and a phenotypically separable fraction that lacks progenitor activity.
简而言之,外层是basal,内层是luminal。其中luminal还可以细分成两类。作者就研究了这3类细胞,再加上 stromal cells, 总共4类细胞。
发表于2018年的《Nature Communications》 , 题目为“Profiling human breast epithelial cells using single cell RNA sequencing identifies cell diversity”。作者从7个个体的乳腺上皮细胞提取25,790 个单细胞进行转录组测序。
鉴定出了三种不同的上皮细胞群:
1种基底细胞
2种管腔细胞,即L1型和L2型
L1型是分泌细胞
L2型在乳腺组织内扮演激素敏感元件功能。
下面看学徒的表演
学徒考核题
文章:Analysis of Normal Human Mammary Epigenomes Reveals Cell-Specific Active Enhancer States and Associated Transcription Factor Networks
需要重现的图片:
这是卖家秀 LP:luminal progenitors
LC:luminal cells
BC:basal cells
找了下面这张图片,没再考证是否还符合当下的流派(了解到是不同的细胞群体就好):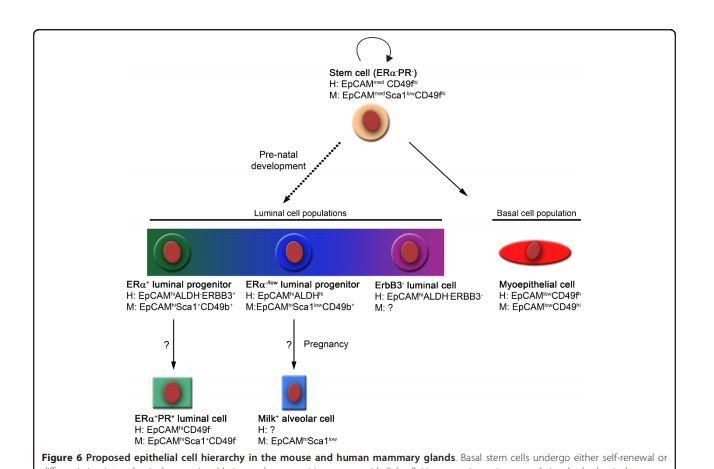
image.png
看着很简单,细节很愁人,最后还是不理想的图:
得到的附件是xls格式,这里是转为csv再读取的;
对图片细节的修饰是最考验耐心的了,我得慢慢练;(不知道是要练细节还是练耐心)
尝试下把几个图片组合到一起,用了
grid,用得不好,后续加强;
rm(list=ls())
a <- read.csv('mmc3.csv',header=T,row.names=1,fill=T)
data<- cbind(log10(a[,1:4]),a[,c(5,6,8)])
library(ggplot2)
library(grid)
library(gridExtra)
ph1<- ggplot(data,aes(x=LP, y=BC,color=BCvsLP))+geom_point()+scale_color_brewer(type='qual',palette=2)
ph2<- ggplot(data,aes(x=LP, y=LC,color=LCvsLP))+geom_point()+scale_color_brewer(type='qual',palette=2)
ph3<- ggplot(data,aes(x=LC, y=BC,color=BCvsLC))+geom_point()+scale_color_brewer(type='qual',palette=2)
data1<- cbind(log2(a[,1:4]+1),a[,c(5,6,8)])
data1$BCvsLP_log[!data1$BCvsLP==''] <- abs(data1$BC[!data1$BCvsLP=='']-data1$LP[!data1$BCvsLP==''])
data1$BCvsLC_log[!data1$BCvsLC==''] <- abs(data1$BC[!data1$BCvsLC=='']-data1$LC[!data1$BCvsLC==''])
data1$LCvsLP_log[!data1$LCvsLP==''] <- abs(data1$LC[!data1$LCvsLP=='']-data1$LP[!data1$LCvsLP==''])
data1[is.na(data1)] <- 0
####boxplot数据处理
tmp<- data1[,8:10]
tmp1<- data.frame(abslog2FC=c(data1[,8],data1[,8],data1[,8]),
group=rep(colnames(data1)[8:10],each=nrow(data1)))
library(ggplot2)
tmp1<- tmp1[!tmp1$abslog2FC==0,]
ph4<- ggplot(tmp1,aes(x = group, y = abslog2FC,color = group )) +geom_boxplot()+coord_flip()
####图片整合
grid.arrange(rectGrob(), rectGrob())
grobs = list(ph1,ph2,ph3,ph4)
marrangeGrob(grobs, nrow=2, ncol=2)
save(data1,file='data1.Rdata')
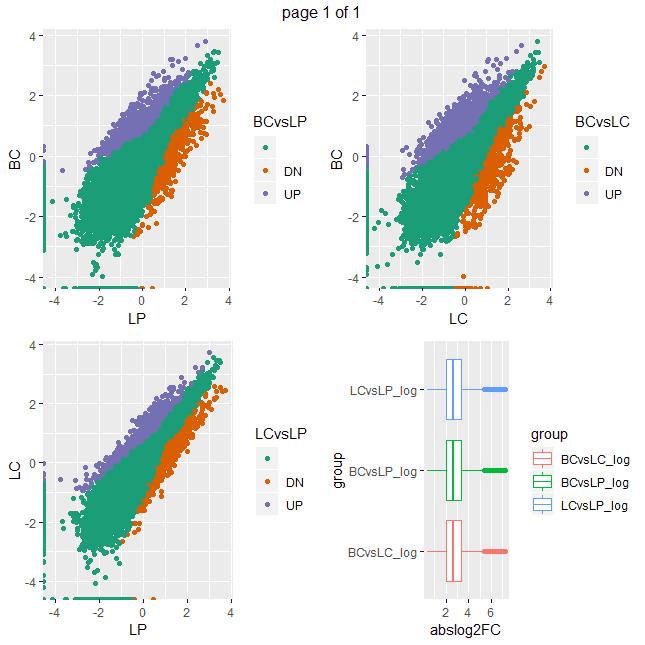
比较下差异基因好了(`不用找,这不是文章里的图`)
这里有个小插曲,运行起来时间比较长(多长呢?我都长草了,console的红灯还亮着),然后一动不敢动,真是最耐心的时候了;
实在是等不起了,索性把任务转到linux里用R操作了,同样有此困扰的,可以试试Move R to Linux[https://www.jianshu.com/p/c2bc2c5e532e],其实非可视化界面也好,挺酷的;
代码是跑完了,就compareCluster函数而已,里面的门道够喝一壶了;
library(clusterProfiler)
load('data1.Rdata')
library(org.Hs.eg.db)
en_sym<-select(org.Hs.eg.db,keys = keys(org.Hs.eg.db),columns = c('ENSEMBL','SYMBOL','ENTREZID'))
data1[,11:13]<- en_sym[match(rownames(data1),en_sym$ENSEMBL),]
BC_LP<- data1$ENTREZID[!data1$BCvsLP=='']
BS_LC<- data1$ENTREZID[!data1$BCvsLC=='']
LC_LP<- data1$ENTREZID[!data1$LCvsLP=='']
gc<- list(all=union(union(BC_LP,BS_LC),LC_LP),BC_LP=BC_LP,BS_LC=BS_LC,LC_LP=LC_LP)
xx <- compareCluster(gc,
fun="enrichGO",
OrgDb="org.Hs.eg.db",
ont= "BP")
dotplot(xx, showCategory=5, includeAll=FALSE)
■ ■ ■
全国巡讲约你
七月份我们不外出,只专注单细胞!
系统学习单细胞分析,报名生信技能树的线下培训,手慢无。
招生
以上是关于学徒数据挖掘第二期汇总之多分组基因注释代码大放送的主要内容,如果未能解决你的问题,请参考以下文章