科研套路不嫌多,数据挖掘发3分
Posted 解螺旋
tags:
篇首语:本文由小常识网(cha138.com)小编为大家整理,主要介绍了科研套路不嫌多,数据挖掘发3分相关的知识,希望对你有一定的参考价值。
做科研需要先学习套路,才能超越套路。今天给大家介绍的套路文献是今年发表在《Oncology reports》(IF= 3.041)上的一篇文章。
文章的标题虽然看上去比较泛,但也让读者一眼就能知道主题了,“molecular mechanism”、“potential drugs”、“papillary renal cell carcinoma(PRCC)”,
所用的研究数据就是“TCGA and Cmap datasets”了
。
当然,也有一些同学看到TCGA这几个字母,就敬而远之了,大家可以翻翻我们之前写的文章,抑或学习下我们的课程,让这些同学们不再只是“远观”,而且可以“亵玩”。
首先,给大家简单介绍下文章method的主要内容:
1. 作者首先用GEPIA做了个
差异表达
(没错,就是那个网页工具);
2. 用MetaScape进行
GO的富集分析
,用Webgestalt进行
KEGG通路富集分析
;
3. 根据上面得到的差异表达基因用Cmap和Drug Pair Seeker进行
药物分子预测
,前者是Broad下的网页工具,后者是需要下载使用的工具,不过两者都是基于Connectivity Map;
4.
构建药物-通路网络
:基于Cmap的数据找到受药物影响的差异表达基因,然后将这些基因做基因富集分析;
5.
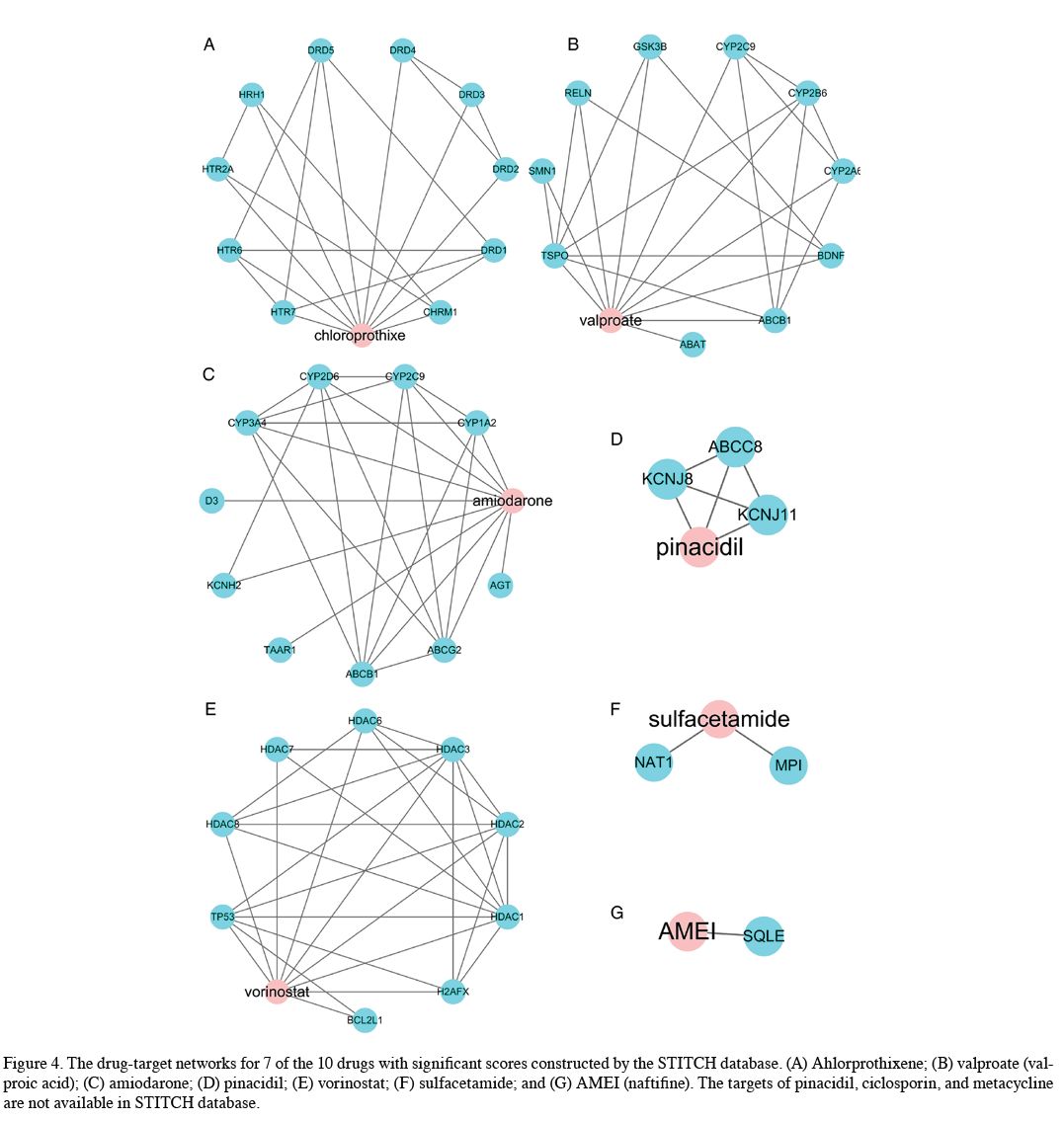
构建药物-靶标网络
:作者从DrugBank里弄到了潜力位于top10的药物的分子结构,然后去STITCH数据库里找他们的靶基因;
6.
进一步探索PRCC的潜在药物
:作者首先用STRING的数据和差异表达的基因,然后在Cytoscape里的CentiScape插件寻找其中的hub基因,再用GEPIA确认hub基因的表达水平。然后在Human Protein Atlas数据库里找了C3和ANXA1的免疫组化结果。最后作者用systemsDock(也是个网页版工具)做了个hub基因蛋白与药物之间的分子对接。
对于新手或者不太熟悉的同学,笔者建议将上述工具和数据库都去学一学用一用,技多真的不压身!(基本都是网站和一些简单的工具)
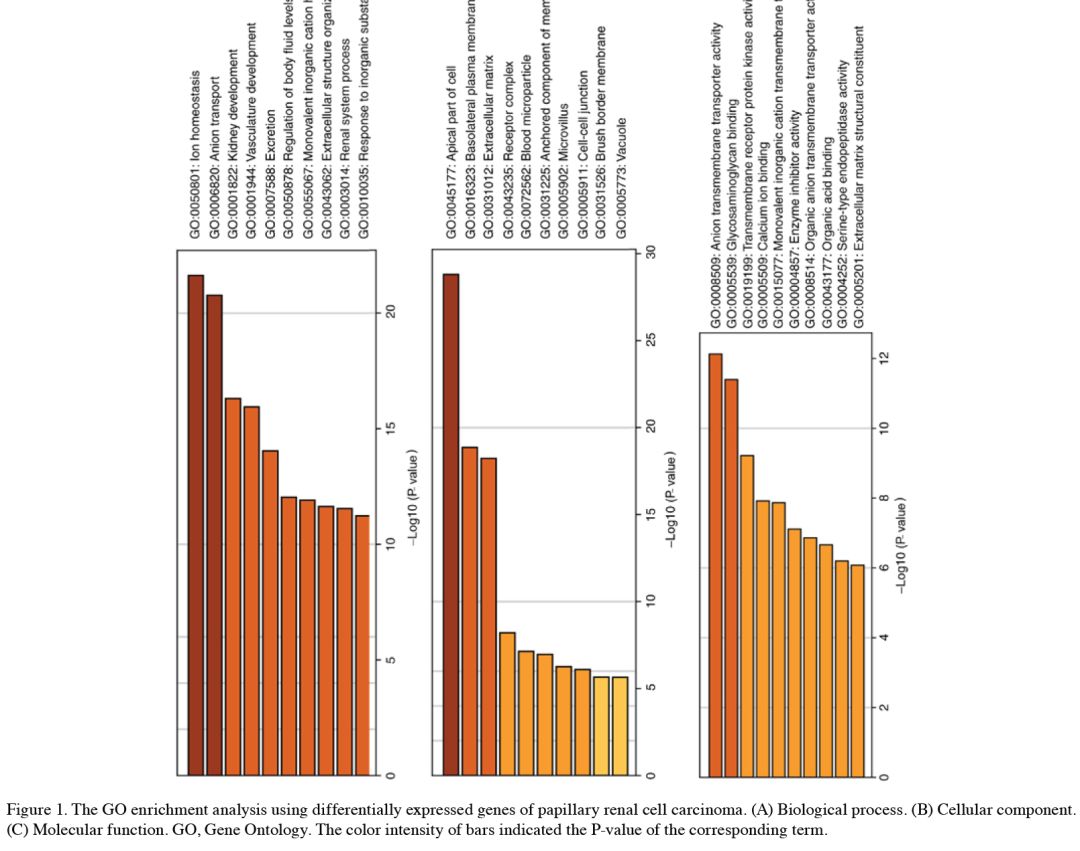
1. Table1和Table2分别展示了PRCC中差异表达基因的
GO和KEGG的富集分析结果
:
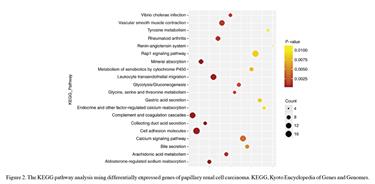
2. Figure1和Figure2也是展示PRCC中差异表达基因的
GO和KEGG的富集分析结果
:
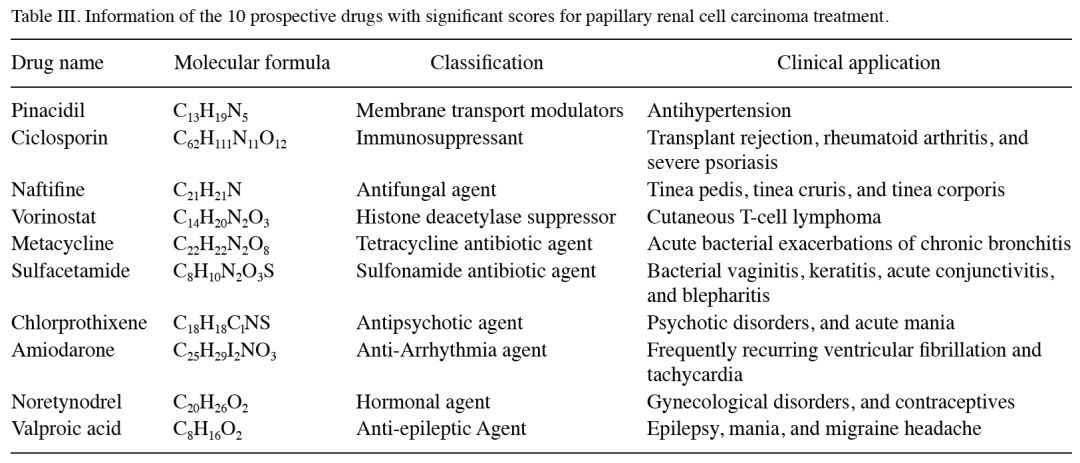
3. 展示了
打分最高最有潜力靶向
RPCC的10个
药物分子
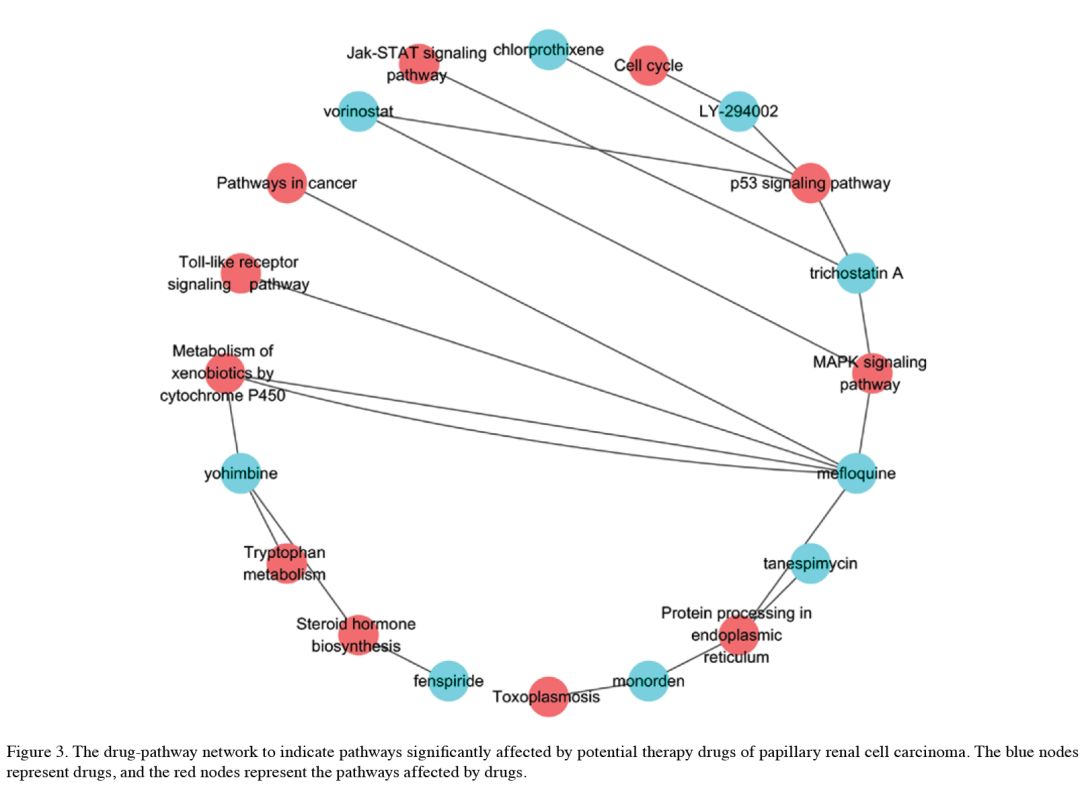
4. 展示了潜在的
靶向药物与信号通路之间的网络关系。
蓝色的表示药物,红色的表示受药物影响的通路。
5. 然后作者展示了基于STITICH数据库构建
药物分子和靶点之间网络关系
的结果。
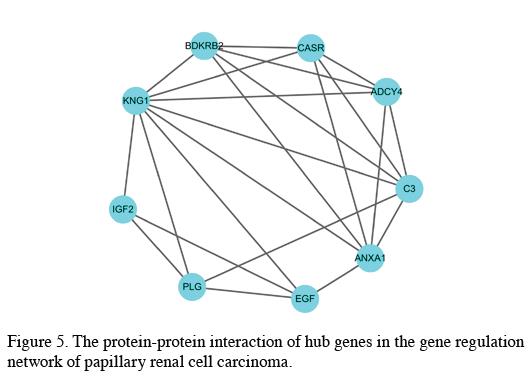
6. 作者在Cytoscape里找到了hub基因,并展示了这些
hub基因之间的PPI网络:
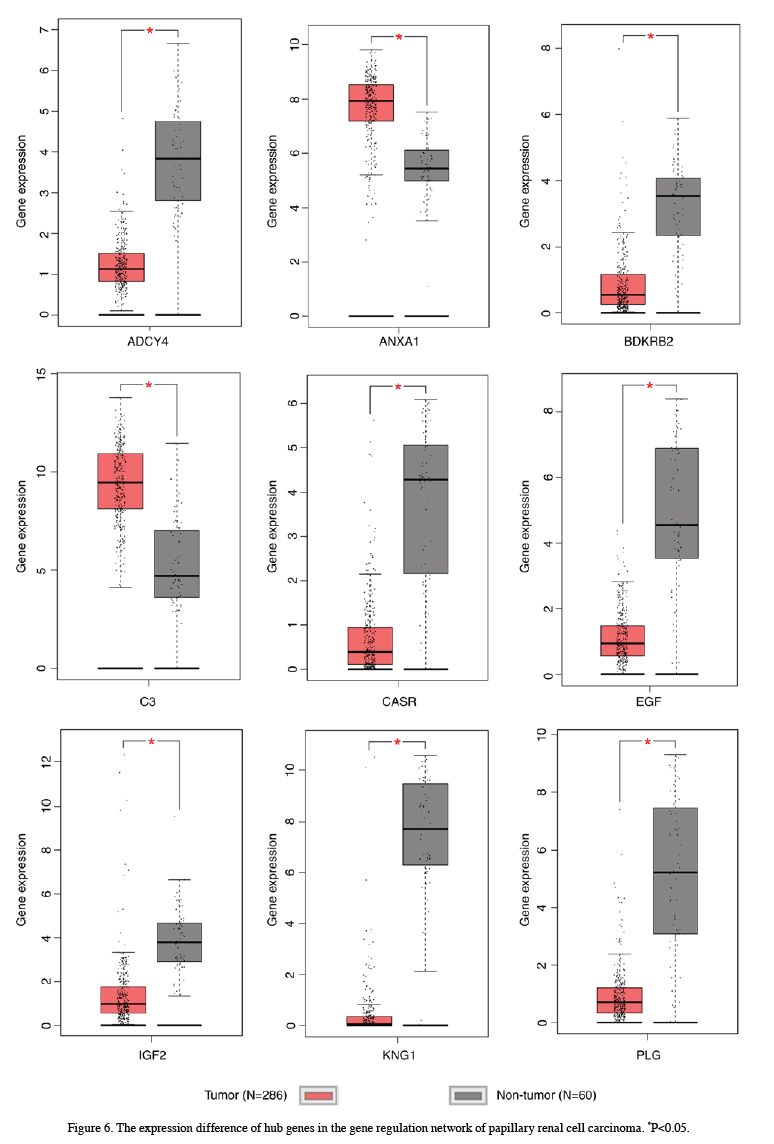
7. 然后作者用GEPIA验证了上面的hub基因在PRCC和癌旁组织中存在
差异表达
(没错,就是GEPIA!)
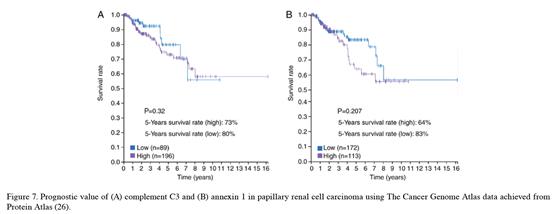
8. 然后作者在TCPA中用C3和ANXA1的蛋白数据做了个
生存分析
(然而两者都不显著啊,为什么不在GEPIA里也做一个mRNA的生存分析呢?)
9. 作者用Protein Atlas的数据
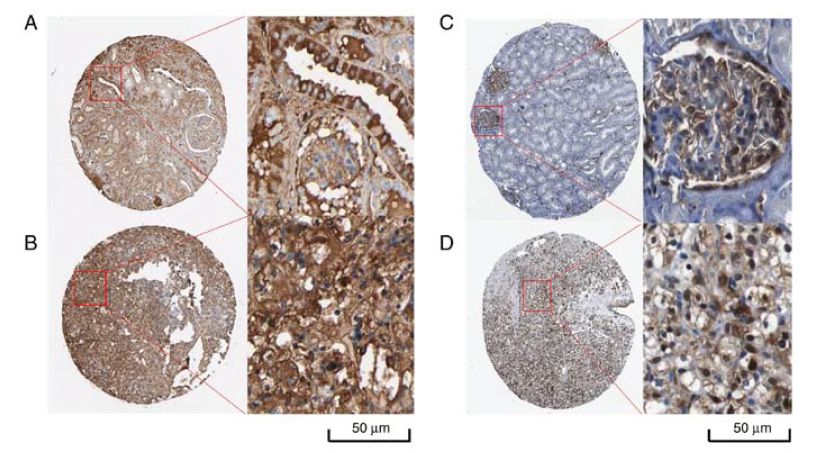
验证了C3和ANXA1两个基因的蛋白
在肿瘤中表达比较高,在60%以上的病人中都检测到了,位列前茅。
10. 然后作者展示了
免疫组化的结果
比较了正常组织和肿瘤组织中的差异(数据挖掘真好,抗体的费用都省了)
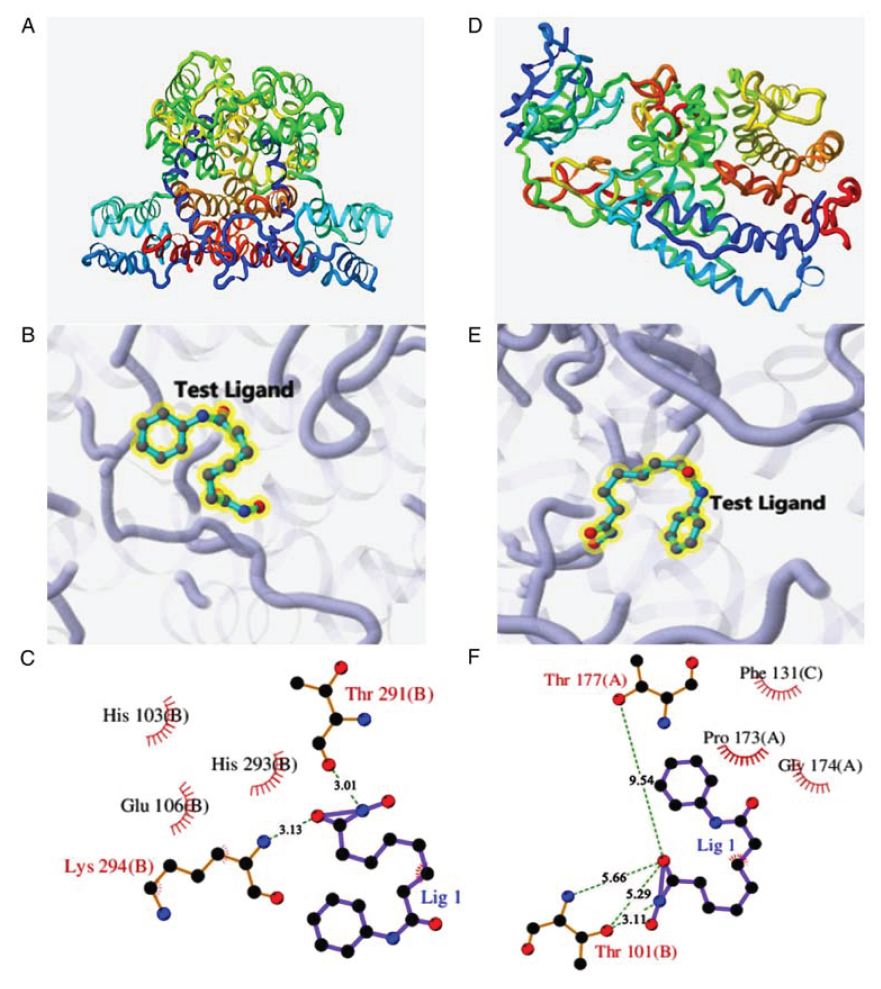
11. 随后作者秀了下C3和ANXA1两个蛋白与药物分子的对接效果图,展示了药物能结合的氨基酸残基和空间距离。
以上就是这篇文章的主要结果,这篇文章对于想要学习数据挖掘的小白们来说,还是值得学习的,至少其中所涉及到的工具还是有不少的,推荐大家去看看学习。
笔者也随手给大家提
两个建议
,如果大家对生信技能稍微熟悉一点,这篇文章里前面的
GO和KEGG可以用GSEA来作
,这样的结果会更靠谱一点,结果图也可以更多一些。
然后就是
可以用TCIA或者TIMER里的免疫细胞浸润数据分析
比较一下正常和肿瘤组织间的免疫浸润差异嘛。
以上是关于科研套路不嫌多,数据挖掘发3分的主要内容,如果未能解决你的问题,请参考以下文章
知网哭穷赔不起1200亿,网友:收钱时咋不嫌多
[培训班 北京] 小张聊科研-实用数据挖掘及案例实操班
python第一课
数据可视化基本套路总结
python大数据挖掘和分析的套路
2018医学方VIP科研沙龙火热开启!实现R语言-数据挖掘-生信文章思路解析三连撞!