TDC | 第一个机器学习在生物医药上的大规模数据集和Leaderboard
Posted DrugAI
tags:
篇首语:本文由小常识网(cha138.com)小编为大家整理,主要介绍了TDC | 第一个机器学习在生物医药上的大规模数据集和Leaderboard相关的知识,希望对你有一定的参考价值。
作者 | Kexin
单位 | Health Data Science @ Harvard
总结
生物医药是机器学习最重要的应用场景之一。但是生物医药涵盖各种各样的任务,数据非常的复杂,而且数据的获得和处理需要很多专业知识。这导致了很多机器学习的研究者只能在少量比较有名但是被研究的很多了的任务上做方法的研究,而忽略了大量的有意义的,但是非常的缺乏机器学习方法研究的任务。为了解决这个问题,我们一帮来自Harvard,MIT,Stanford,CMU,UIUC,Georgia Tech,IQVIA的学生和教授一起发起了Therapeutics Data Commons (TDC),第一个机器学习在生物医药的大规模数据集。TDC目前包含了20+有意义的任务,和70多个高质量数据集,从靶蛋白的发现,药物动力学,安全性,药物生产都有包含到。而且不仅仅是小分子,还有抗体,疫苗,miRNA等。之后也会加入CRISPR,Clinical Trials等等。这些数据都是处理成可以直接输入进机器学习模型的。而且大部分都是新坑!我们也提供了一些leaderboard来提供模型SOTA对比。欢迎大家使用TDC和提供建议!更多信息,请访问网站和GitHub!
网站
https://zitniklab.hms.harvard.edu/TDC/
GitHub
https://github.com/mims-harvard/TDC
背景
近几年来,机器学习(Machine Learning, ML)在生物药医药领域有了非常多的发展和应用,比如最近的AlphaFold2在蛋白质结构预测上的大幅效果提升[1],ML预测出来的强力抗生素Halicin[2]等。但是,获取和处理原始生物医药数据到ML-Ready数据需要很多专业知识,对于机器学习的研究者来说,很难快速精准的处理出来。而且生物医药是个巨大的领域,很多数据集都分散在各个角落,没有一个中心的平台来整理和获取这些数据。因为这些原因,现在的ML研究者在方法研究上只关注非常少的几个任务来在几个小数据集上来增进结果的几个点,然而大量的有意义的任务都还没有被前沿的ML方法所研究。这个极大的降低了ML在生物医药领域的研究进度。
TDC 介绍
TDC Team
为了解决这个问题,我(Kexin)和来自Georgia Tech的Tianfan @futianfan, MIT的Wenhao, CMU的Yue @微调, Stanford的Yusuf,以及我们的导师们Connor, Jure, Jimeng, Danica和Marinka,一起发起了Therapeutics Data Commons(TDC), 第一个大规模的ML在生物医药上面的数据集和基准。

在第一个版本里,我们整理了ML在生物医疗上的20多个非常有意义的任务和70多个数据集,从靶蛋白的发现,药物动力学,安全性,药物生产都有包含到。而且不仅仅是小分子,还有抗体,疫苗,miRNA等。除了数据本身,我们在平时做研究的时候注意到有很多经常用到的数据函数,于是我们也提供了很多函数来支持ML在生物医药的研究。所有ML-ready数据和data functions都只用三行代码就可以获得!
TDC Overview
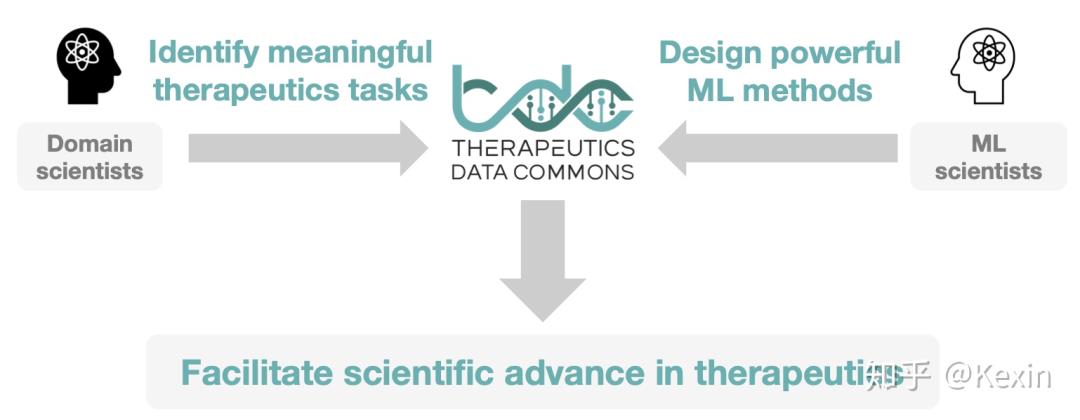
TDC Vision
通过TDC,我们想达到的最终目的是作为一个连接点:生物医药行业的人发现有意义的领域问题,TDC把它变成ML的任务以及处理成ML-ready的数据,然后ML的研究者可以快速的使用TDC来设计前沿的ML方法。通过这样,我们希望可以帮助ML社区的人关注在解决有现实意义和有价值的生物医药问题上。
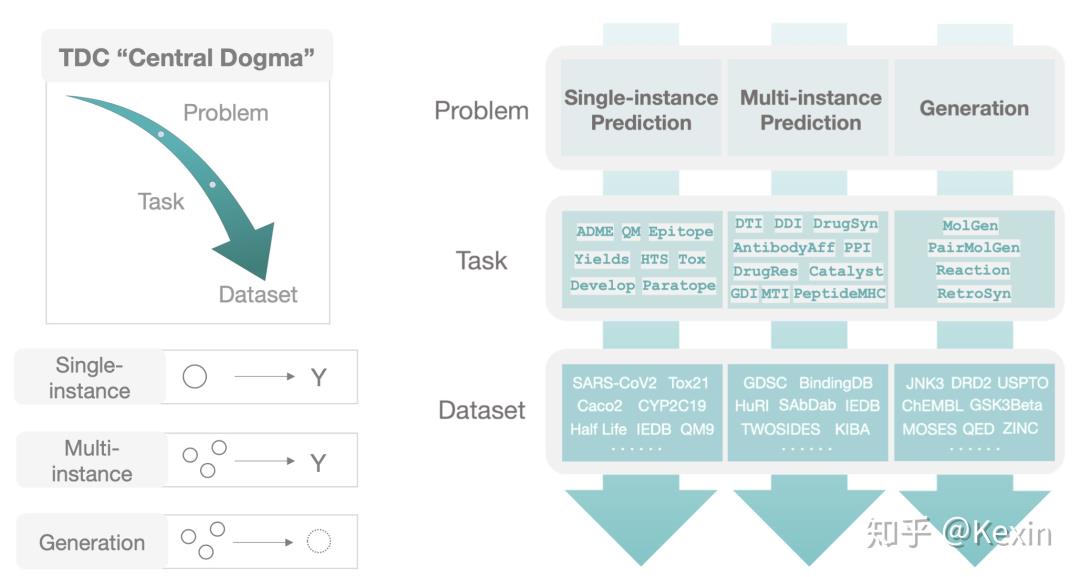
TDC Modular Design
TDC的结构
接下来我来详细的介绍一下TDC。TDC的目的是涵盖多种多样的任务,而每一个任务有不一样的数据结构。因此,我们提出了一个三层阶级式的架构---我们叫它TDC “Central Dogma”(中心法则)。据我们所知,这是系统评估机器学习在整个生物医药领域中的首次尝试。
第一层是问题 Problem,我们把所有的任务归纳成3个大的ML问题:
Single-instance prediction(单实例预测): 预测单个实体(比如分子,蛋白)的某些性质。
Multi-instance prediction(多实例预测): 预测多个实体之间的某些性质(比如反应类型)
Generation(生成): 已知一系列的实体,生成新的拥有某些性质的实体(比如优化后的分子)
第二层是学习任务 Task。每个任务都属于其中一个问题类型。这些任务是从生物医药的角度去定义的。在应用中的改进范围包括设计新的抗体,识别个性化的组合疗法,改善疾病的诊断以及寻找新的治疗新疾病的方法。
最后,在TDC的第三层中,每个任务都通过多个数据集 Dataset 实例化。
总结一下,就是有三个问题,每个问题有很多学习任务,每个学习任务又有很多数据集。这个三层结构让我们很清晰的去整理和使用TDC。
TDC的编程框架
在TDC的编程框架里,我们给每一个Problem设计了一个基础的Data Loader(数据加载)class,然后每一个Task继承了这个base class。所以假设你想要检索问题“ Z”类里的学习任务“ Y”里的数据集“ X”,你只要输:
from tdc.Z import Ydata = Y(name = 'X')splits = data.split()
举个例子,假设你想要获取药物ADME预测任务中的Bioavailability Dataset,你可以直接输
from tdc.single_pred import ADMEdata = ADME(name = 'Bioavailability_Ma')split = data.get_split()
这个split里就包含了三个Pandas DataFrame,每一行是最通用的药物分子输入格式SMILES String(可以理解为药物分子图的字符表达式)以及Bioavailability值。
TDC的数据
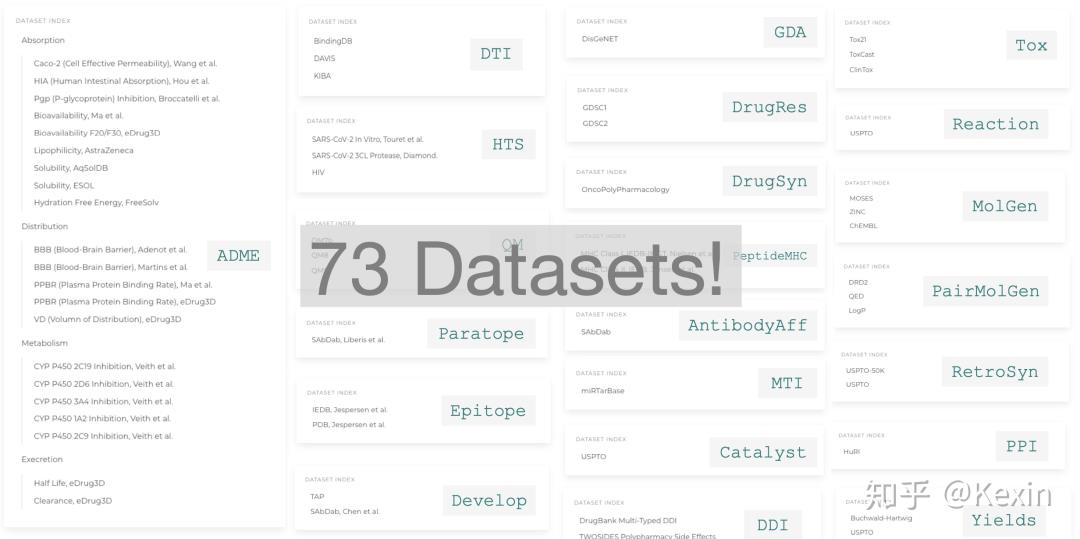
TDC Datasets Snapshot.
就像刚开始说的,你可以就只用这三行代码来获取70多个有意义来自20多个重要的生物医药任务的数据集!很多任务在ML方法研究里都是全新任务或者全新的数据集(未挖掘的新坑)!举几个例子:
ADMET预测:ADMET包含了一系列重要的药物指标来衡量的一个药物分子从口服下去以后能不能安全且有效的达到想要的靶点上。准确的预测这些指标可以节省药企很多资源。之前有很多网页服务器来做ADMET预测,可数据都是非公开的。TDC从很多小数据库和零散的期刊等等来源收集了20多个药厂里都实际在用的重要的指标,所有数据全部开源!
精准组合药物:有两个比较大的趋势是一,同样的药物根据不一样的病人个体,效果会不一样,特别是肿瘤方面的药。因此ML来预测药在不一样的病人的基因表达下的效果很重要。TDC从GDSC[3]里处理出来一个大数据集,里面每一个数据点对应一个药物分子和Cell Line的基因表达,和它们之间的反应效果。另外一个趋势是很多药物分子的组合会比单个药物分子有更好的效果(drug synergy),而且能够大量的节省研发时间。所以如果能够预测出两个药物之间会不会有组合效应,是非常有意义的。TDC处理了两个大数据集(来自Merck[4]以及NCI[5]),每一个数据点包含2个药物分子结构和cell line expressions,以及他们的synergy effect。
生物药(Biologics)。近几年来ML在小分子上有很多不错的工作,可是在大分子生物药上没有很多工作。TDC于是包含了6个生物药方面的任务,比如抗体和抗原的亲和力预测,多肽和MHC的亲和力预测,miRNA和靶点的反应预测等等。
我们最近也在准备包含一些3D的药物分子和蛋白任务,CRISPR基因编辑方面的任务(off-target,repair outcome),和clinical trials方面的任务。如果你有新的有意思的任务,也请联系我们!
TDC Leaderboard
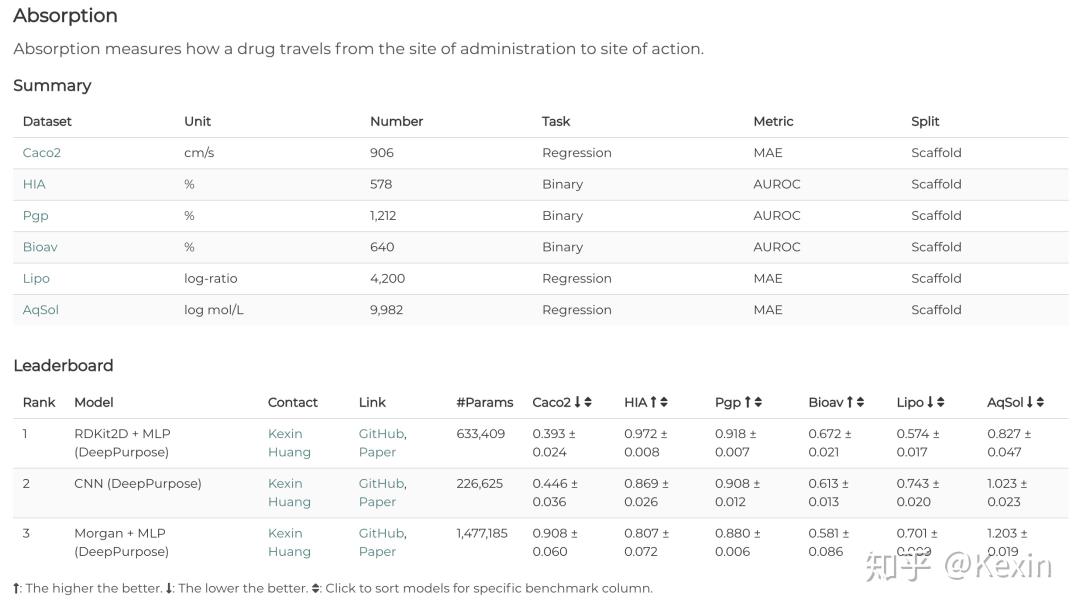
Absorption Category in ADMET Benchmark Group
在这些数据集的基础上,TDC也提供各种各样的Leaderboard来给ML研究者们对比模型预测效果。TDC的每个数据集都可以作为一个基准。但是我们观察到要真正使用一个ML模型在很多生物医药问题上,这个ML模型必须在一系列的数据集和任务上达到好效果。因此,我们合并很多子基准来形成一个基准组合(Benchmark Group)。一个组合里的所有子基准都围绕着一个有意义的生物医疗问题,所有的衡量标准和训练测试分割方式我们都设计过来模拟实际生物医药的应用场景。举个例子,我们第一个基准组合是ADMET性质预测。一个好ML模型不仅仅要对于某一个ADMET的性质预测好,而是要对所有的性质都有好效果。所以在这个组合里,我们有22个ADMET性质预测子基准。我们也用scaffold split去模拟实际药厂的应用场景。
对于一个Benchmark Group,TDC提供了一个编程框架去快速的让ML研究者去搭建和评估模型。比如用ADMET group作为例子,
from tdc import BenchmarkGroupgroup = BenchmarkGroup(name = 'ADMET_Group', path = 'data/')predictions = {}for benchmark in group:name = benchmark['name']train, valid, test = benchmark['train'], benchmark['valid'], benchmark['test']predictions[name] = y_predgroup.evaluate(predictions)
你也可以获取每一个子基准:
benchmark = group.get('Caco2_Wang')predictions = {}name = benchmark['name']train, valid, test = benchmark['train'], benchmark['valid'], benchmark['test']predictions[name] = y_predgroup.evaluate(predictions)
除了提供最标准的一个train/valid/test分割,你也可以获得很多不一样的train/valid splits,来测试模型的鲁棒性:
out = group.get_auxiliary_train_valid_split(seed = 42, benchmark = 'Caco2_Wang')train, valid = out['train'], out['valid']group.evaluate(y_pred_val, y_true_val, benchmark = 'Caco2_Wang')
TDC第一个leaderboard 关于ADMET已经发布!ADMET非常的重要,而且是个非常适合没有任何生物医药背景的ML研究者起步的任务。欢迎大家提交结果!我们网站里有更多的信息。
TDC的数据处理函数
TDC Data Functions
除了核心的数据集以及Leaderboard以外,TDC也包含了各种各样的functions。现在主要有四块:
模型评估:TDC提供了一个只要3行代码的评估函数,来评估TDC里的任务。
数据分割:一些训练和测试集的分割方法,来模拟实际的生物医药场景。比如scaffold split。
数据处理:一些helpers比如可视化,标签的转化,binarization等等。
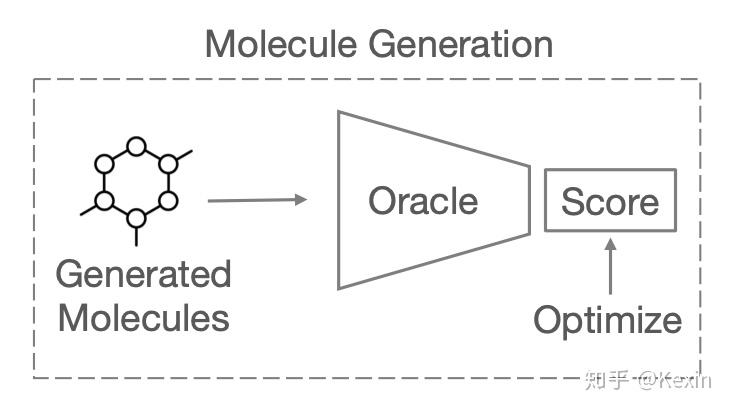
分子生成Oracles:在分子生成任务中,一般的目标是去产生新的药物分子有更好的性质,然后这个性质是通过一个默认gold label函数(oracle)来产生的。所以一个oracle就定义了一个分子生成的任务。TDC收集了20多个有意义的oracles,全部都只要3行代码。举个例子:
from tdc import Oracleoracle = Oracle(name = 'GSK3B')oracle(['CC(C)(C)[C@H]1CCc2c(sc(NC(=O)COc3ccc(Cl)cc3)c2C(N)=O)C1', \'CCNC(=O)c1ccc(NC(=O)N2CC[C@H](C)[C@H](O)C2)c(C)c1', \'C[C@@H]1CCN(C(=O)CCCc2ccccc2)C[C@@H]1O'])# [0.03, 0.0, 0.0]
之后我们会逐步添加更多有意义的data functions,比如各种分子格式之间的转化,更有实际意义的oracles(比如之后会提供docking score,逆合成步数)等等。
安装TDC
TDC的核心数据只有非常少的环境要求,你可以通过pip来安装它:
pip install PyTDC对于一些比较具体的data functions,TDC需要更加多的环境。你可以通过conda-forge安装:
conda install -c conda-forge pytdcTDC是一个开源社区
TDC网站和GitHub
所有的详细信息都在TDC的网站里,
https://zitniklab.hms.harvard.edu/TDC/
也欢迎大家star TDC的GitHub https://github.com/mims-harvard/TDC 来看TDC的最新消息!
有任何问题,欢迎评论或者私聊我!
参考资料
[1] AlphaFold: a solution to a 50-year-old grand challenge in biology. AlphaFold: a solution to a 50-year-old grand challenge in biology
[2] Stokes, Jonathan M., et al. "A deep learning approach to antibiotic discovery."Cell. 180.4 (2020): 688-702.
[3] Yang, Wanjuan, et al. “Genomics of Drug Sensitivity in Cancer (GDSC): a resource for therapeutic biomarker discovery in cancer cells.” Nucleic acids research 41.D1 (2012): D955-D961.
[4] O’Neil, Jennifer, et al. “An unbiased oncology compound screen to identify novel combination strategies.” Molecular cancer therapeutics 15.6 (2016): 1155-1162.
[5] Zagidullin, Bulat, et al. “DrugComb: an integrative cancer drug combination data portal.” Nucleic acids research 47.W1 (2019): W43-W51.
以上是关于TDC | 第一个机器学习在生物医药上的大规模数据集和Leaderboard的主要内容,如果未能解决你的问题,请参考以下文章
机器学习案例三:数据降维与相关性分析(皮尔逊(Pearson),二维相关性分析(TDC),灰色关联分析,最大信息系数(MIC))
弗雷塞斯 从生物学到生物信息学到机器学习 转录组入门:了解fastq测序数据