ISME:南农沈其荣团队基于大数据准确预测土壤的枯萎病发生
Posted 宏基因组
tags:
篇首语:本文由小常识网(cha138.com)小编为大家整理,主要介绍了ISME:南农沈其荣团队基于大数据准确预测土壤的枯萎病发生相关的知识,希望对你有一定的参考价值。
基于大数据整合准确预测土壤的枯萎病发生
Predicting disease occurrence with high accuracy based on soil macroecological patterns of Fusarium wilt
Article,2020-07-17
The ISME Journal, [IF 9.49]
DOI:https://doi.org/10.1038/s41396-020-0720-5
通讯作者 Qirong Shen (沈其荣,shenqirong@njau.edu.cn)
主要单位:南京农业大学(The Key Laboratory of Plant Immunity, Jiangsu Provincial Key Lab for Organic Solid Waste Utilization, National Engineering Research Center for Organic-based Fertilizers, Jiangsu Collaborative Innovation Center for Solid Organic Waste Resource Utilization, Nanjing Agricultural University)
写在前面
副主编(文涛)寄语:从团队的角度来讲:作为南农沈其荣老师旗下土壤微生物团队的成员,我们团队,袁军老师还有我十分感谢在修改文章阶段,刘永鑫老师对文章提出的建议和改进,我们几乎全部采纳了刘老师的建议。

主要结果
1. 世界范围内镰刀菌枯萎病相关微生物组数据的基本统计
我们收集了目前世界上公开发表的大部分有关镰刀菌枯萎病相关的土壤微生物测序研究,并展示了这些研究主要的分布,发现大部分研究取样点都在中国(图1),这于我国的种植模式有关。其次我们分析了这些测序项目的一些基本特征,种植作物,国家,测序区域,测序平台等。发现大部分数据都是使用Illumina的各种平台测定的(图1),表明了数据公开在这几年逐渐被多数人接受。
为了得到目标数据,本研究使用两种策略来获得原始测序数据。1)基于已发表研究,通过关键词(Fusarium wilt community、Fusarium wilt structure或Fusarium wilt microbiome)从谷歌学术查找与镰刀菌枯萎病微生物群落相关的研究论文,得到测序样品登录号,对这些原始数据分析发现,大部分相关研究的原始数据存储在美国生物技术信息中心(NCBI),但也有一部分数据存储在日本核酸数据库(DDBJ)和欧洲核酸数据库(EUI)。2)目前NCBI上存储了大量尚未公开发表文章的数据,往往这些数据的分组及其采样信息被描述的很清楚,我们通过项目号搜索Fusarium wilt community、Fusarium wilt structure或Fusarium wilt microbiome,在NCBI上得到一批目前尚未发表但有原始数据的项目。将以上两种策略得到的相关项目合并,即为最终整合的项目数量。
当得到足够的相关测序项目后,使用相关生物信息学工具下载核酸序列(原始数据);然后以QIIME、USEARCH工具为核心,使用R、Python和Shell等语言为胶水,对原始数据进行分析,得到常见的OTU表格;最后合并不同研究的OTU,得到基于整合后的多个独立研究的合并表格。
图1 微生物组数据样本收集概况
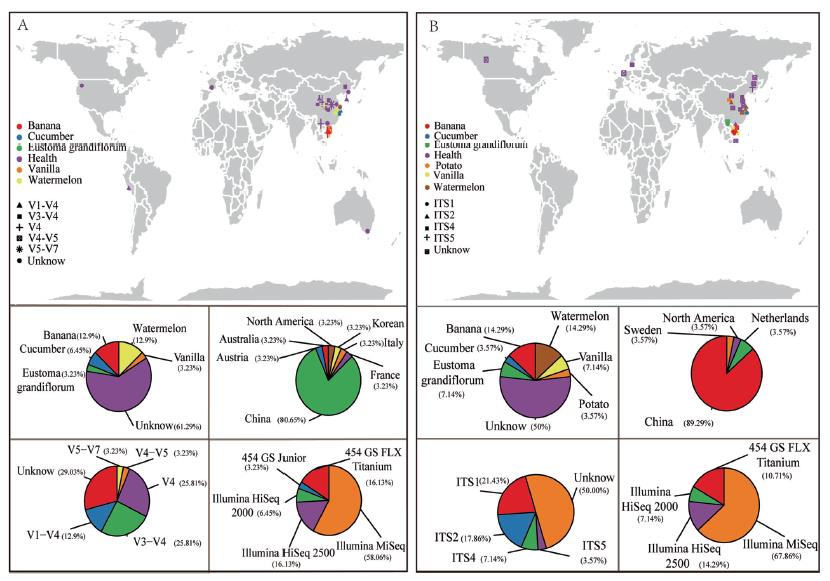
A. 细菌16S,B. 真菌ITS
2. 整合的微生物组数据基本多样性特征
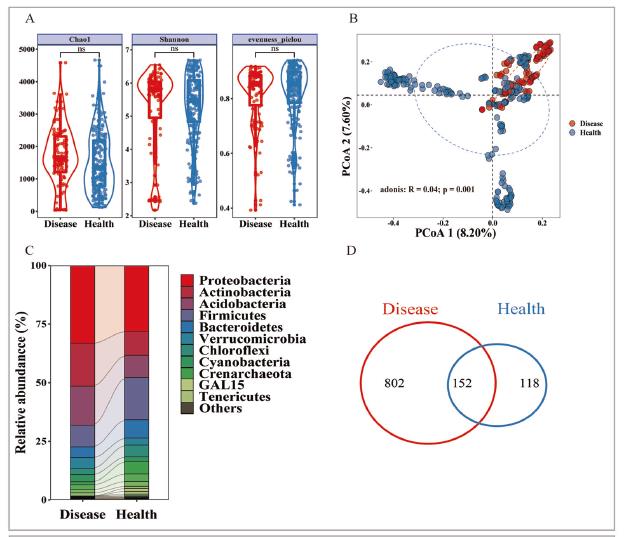
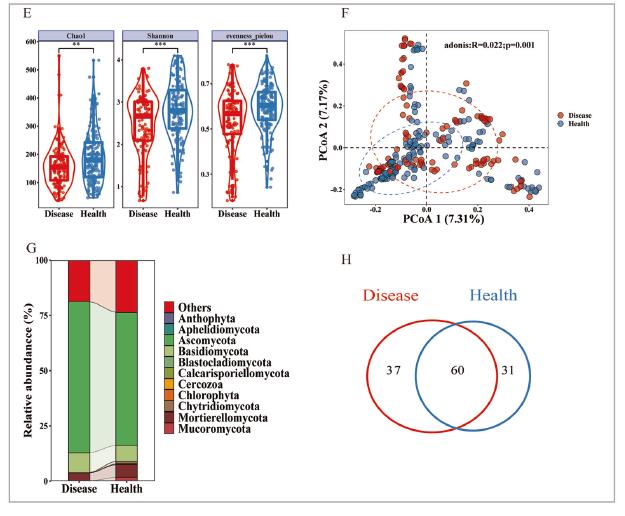
就细菌群落而言,将全部OTU表格合并之后发现群落中OTU的数量已经超过40000个。为了对微生物群落进行一个概括,首先使用传统的多样性分析方法表征基于多研究整合的微生物群落的宏观特征。将OTU表格按照3000条序列数抽平随后计算Chao1、Shannon和evenness_pielou三个指标,但是这三个指标均没有在发病土壤和健康土壤中表现出差异(图2A);其次通过Bray-Curtis举例对群落进行PCoA排序,通过多元置换方差分析进行差异检测后发现发病细菌群落和健康土壤群落之间显著不同(p = 0.001;permutational multivariate analysis of variance (PERMANOVA) by Adonis)(图2B);最后通过堆叠柱状图展示细菌群落在门水平上的物种分布和丰度特征,发现健康细菌群落中变形菌门(Proteobacteria)、放线菌门(Actinobacteria)和酸杆菌门(Acidobacteria)相对丰度较发病土壤高(图2C),但是发病土壤细菌群落中有更多的特有OTU(发病土壤802个,健康土壤118个)。
就真菌群落而言,将全部OTU表格合并之后发现群落中OTU的数量已经超过8000个,这个数量是远远小于细菌群落的,这与土壤中真菌数量少、数据库不够全面有关。我们使用与细菌群落数据处理相同的流程对整合后的真菌群落进行alpha多样性分析发现,与细菌群落表现不同,这些指标在健康土壤中显著高于发病土壤(图2D);同样通过Bray-Curtis距离对群落进行PCoA排序,通过多元置换方差分析进行差异检测,发现真菌微生物群落在发病和健康土壤中显著不同(p-values < 0.05;permutational multivariate analysis of variance (PERMANOVA) by Adonis)(图2E)。在发病土壤中Ascomycota的相对丰度更高,而在健康土壤中Mortierellomycota却更多(图2F)。
虽然PCoA排序可以从总体上确定基于整合分析的发病微生物群落和健康微生物群落之间的不同,但是这远未达到预期目的,为了找出造成发病和健康土壤中微生物群落差异的特征微生物,我们使用了在微生物领域运用很好的机器学习。
图2 枯萎病发病和健康土壤的微生物群落多样性和物种组成
3. 机器学习区分枯萎病发病和健康土壤的微生物群落特征
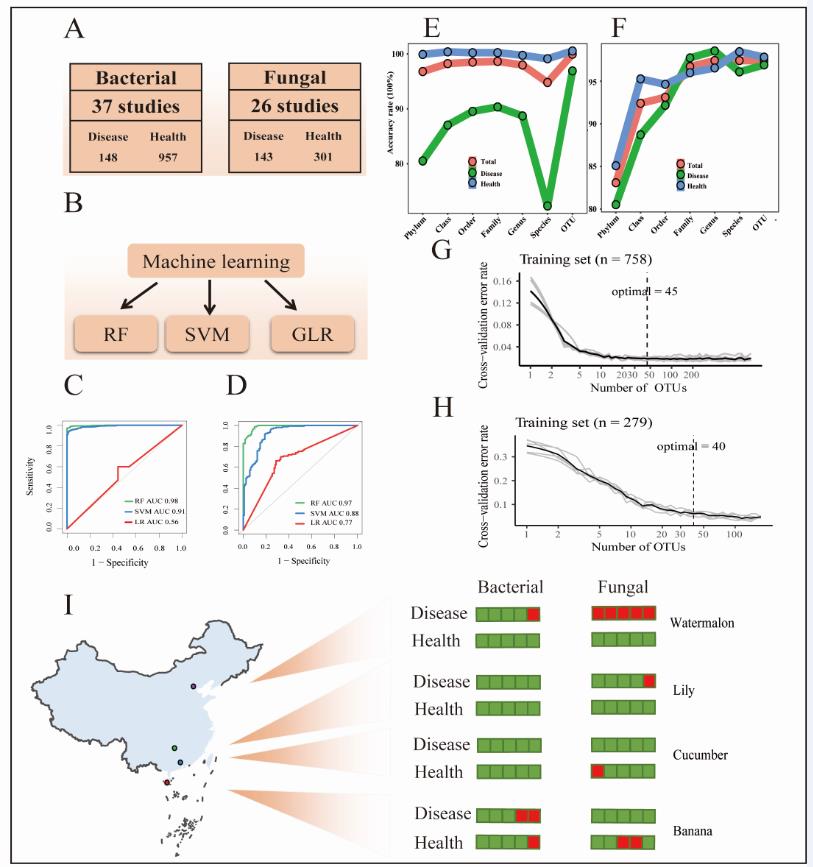
为了更好的区分枯萎病患病土壤和健康土壤的微生物群落,我们选择了三个机器学习算法,即随机森林(RF)、支持向量机(SVM)和逻辑回归(LR),分别对发病土壤和健康土壤的微生物群落进行学习并构建分类器,根据准确度,我们选择了随机森林(一种用于分类和回归分析的稳定机器学习算法)创建了分类模型。由于对不同分类等级微生物进行建模后的效果不同,因此我们将细菌微生物群落分类单元按照门、纲、目、科、属和OTU水平分别进行分类建模,以获得区分枯萎病土壤和健康土壤中最佳的分类器,结果表明,无论是细菌群落还是真菌群落,均在OTU水平上建模效果最佳,准确度分别为98.94%和96.42%(图3A-B)。
为了进一步验证模型的准确度,一方面我们纳入剩余的研究并进行分类预测。通过分析来自NCBI SRA数据库的13个独立的细菌测序数据集(包括26个患病土壤样本和321个健康土壤样本)进行细菌模型验证,发现细菌模型对所有采集样本预测的平均准确率为94.46%,其中,对患病样本的准确率为91.75%,对健康样本的准确率为96.45%)。同时,我们使用来自NCBI SRA数据库的6个独立的真菌测试数据集(包括21个病土样本和144个健康土样本)进行真菌模型验证,发现真菌模型对所有采集样本的平均准确率为93.05%,其中,对患病样本的准确率为91.67%,对健康样本的准确率为95.25%。
另一方面,我们从全国范围内采集了枯萎病发病和健康土壤,具体如下:我们于2019年4月下旬分别从海南、广东、北京和江苏采集了包含香蕉、黄瓜、西瓜和百合在内的4种不同作物的田间土壤样品,采样时选择至少连作5年且遭受镰刀菌枯萎病感染的地块,同时选择了没有发生枯萎病的新开垦农田进行健康土壤收集,为了避免地理因素引起的差异,在病害地附近选择健康地。将样本采集后按照标准的DNA提取、建库和测序流程测定了土壤中的细菌群落和真菌群落。原始数据的处理流程同样采取先前整合数据的分析流程。通过细菌模型对样本分类预测,发现所有样本的平均准确率为90.00%,其中对患病样本的准确率为87.50%,对健康样本的准确率为92.50%(图3I)。使用真菌模型进行分类预测,表明所有样本的平均准确率为80.00%,其中对病土样本的准确率为97.50%,对健康土样本的准确率为62.50%(图3I)。这一结果说明我们通过随机森林建模得到的分类器对枯萎病发病和健康土壤的微生物群落特征进行了良好的区分。
图3 机器学习建模、特征提取和预测
4. 基于机器学习得到的微生物特征解析
随机森林分类器很好的区分了枯萎病发病土壤和健康土壤,更重要的是我们得到了作为区分发病和健康土壤的特征微生物。就细菌群落而言,我们通过交叉检验对参与建模的1339个OTU重要性进行评估,发现其中45个OTU对分类器的确定性至关重要,我们称这45个OTU为特征OTU,也叫生物标记物(图4C)。这些OTU的平均丰度在4.84%~0.16%,对分类影响最大的OTU属于Gemmatimonadetes菌门。这45个OTU中有33个在发病微生物群落中相对丰度较高(FDR adjusted P < 0.05, Wilcoxon rank sum test;),他们主要属于变形菌门(Proteobacteria)和酸杆菌门(Acidobacteria),在更低的分类水平上属于Sphingomonadaceae、Hyphomicrobiaceae、Koribacteraceae和Xanthomonadaceae菌科;但是在健康土壤中相对丰度较高的微生物在更低的分类水平上属于Streptomycetaceae、Bradyrhizobiaceae、Inobacteraceae、Comamonadaceae和Chthoniobacteraceae科。就真菌群落而言,我们通过交叉检验对参与建模的177个OTU重要性进行评估,发现其中40个OTU对分类效果至关重要,其中26个OTU在发病土壤中具有较高的相对丰度,其余14个在健康土壤中丰度较高,这些属于Fusarium和Mycothermus属的OTU在发病土壤中丰度较高,但是属于Aspergillus、Mortierella和Neurospora属的OTU在健康土壤中丰度较高(图4D)。与我们认识一致的是尖孢镰刀菌在发病土壤中的丰度(7.80%)高于健康土壤(4.00%),但是同属于镰刀菌属的Fusarium keratoplasticum的相对丰度却在健康土壤中更高。
图4 特征细菌和真菌丰度展示
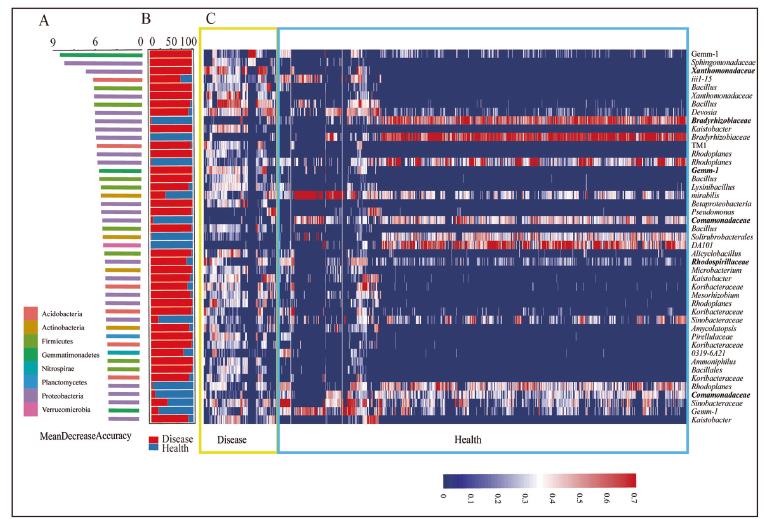
45个细菌OTU特征展示
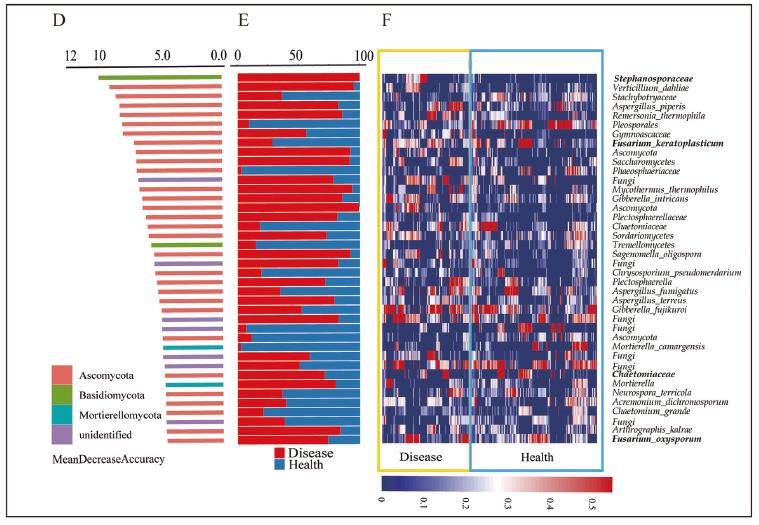
40个真菌OTU特征展示
5. 细菌特征OTU网络分析
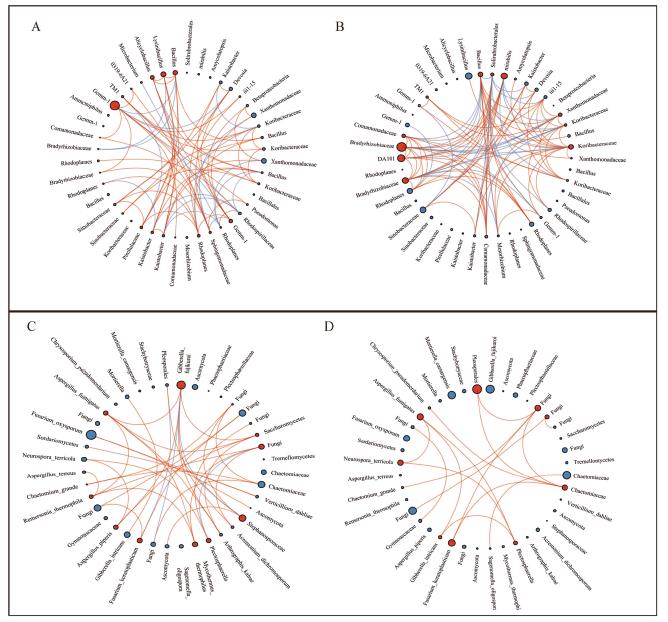
细菌特征OTU网络分析表明,健康网络中包含的节点和连接数较多,并且网络平均度(average degree)和中心紧密度(centralization-closeness)较高。一些被注释为Bacillaceae、Hyphomicrobiaceae、Bradyrhizobiaceae、Comamonadaceae、Streptomycetaceae和Chthoniobacteraceae的OTU在健康网络中连接数更高,同时他们在健康土壤群落中相对丰度更高。另外一些注释为Alicyclobacillaceae和Sphingomonadaceae的OTU在发病网络中连接数较高,同时在发病土壤群落中相对丰度较高。值得注意的是,OTU1111883和549433属于Gemmatimonadetes门,在分类器中的重要性较高,在发病土壤群落中相对丰度及与其他OTU之间的连接数也更多。这些OTU可能在发病微生物群落中具有重要作用(图5A-B)。
真菌特征网络不同于细菌特征网络,发病真菌特征网络包含的节点和连接数更多,平均度(average degree)和中心紧密度(centralization-closeness)也更高。就具体的OTU而言,三个分别属于Aspergillus、Remersonia属和Stephanosporaceae科的OTU在发病真菌网络中具有较高的中心度(center degree)和中介性(betweenness)。在健康真菌特征网络中属于Chaetomiaceae属的OTU具有较高的中心度和中介性(图5C-D)。
图5. 细菌真菌特征OTU网络分析
作者介绍
沈其荣教授

沈其荣简介:南京农业大学学术委员会主任,长期从事土壤微生物、有机肥和生物有机肥研究。提出和建立了土壤微生物区系调控的理论和技术体系,并研发出调控土壤微生物区系的有机(类) 肥料系列产品(有机肥、有机无机复混肥、生物有机肥、全元生物有机肥),相关技术工艺已被全国600 多家企业采用;研发的土壤熏蒸与生物有机肥联用防治土传病害综合技术效果显著,为中国土壤生物肥力提升和有机(类)肥料产业发展做出了重大贡献。以第一完成人获国家技术发明二等奖1项、国家科技进步二等奖1项、国家专利金奖1项、国家专利优秀奖1项、国家教学成果二等奖1项以及省部级一等奖8项。以第一和第二 完成人获中国发明专利60多件、国际PCT专利6件,其中50多件专利在企业 得到转化。发表SCI论文400多篇,H指数50,2014年-2018 年连续五年入选 ELSEVIER中国高被引学者榜单。获全国创新争先奖状、国家教学名师、全国师 德先进个人、全国优秀科技工作者、江苏省劳模和先进工作者、农业部和江苏省突出贡献专家、江苏省十大杰出发明人和江苏省首届发明人奖。已累计培养硕士研究生121 名、博士研究生83 名。目前和曾经兼任:国家973 计划首席科 学家、 国家863 计划现代农业领域主题专家、第六届和第七届国家自然科学基 金委生命学部专家咨询委员会委员、 第七届国务院学位委员会农业资源与环境 学科评议组召集人、中国有机(类)肥料产业技术创新联盟理事长、中国土壤 学会副理事长、中国植物营养学会生物与有机肥专业委员会主任、农业部耕地 质量建设与管理专家组组长、教育部和农业部科技委委员、中国农科院学术委 员会委员、江苏省自然科学基金委农业学科组组长等社会职务。
袁军

袁军,博士,就职于南京农业大学资环学院沈其荣教授团队。研究方向:植物-土壤反馈,土壤微生物群落调控,连作障碍修复,新型肥料研发。目前以第一作者在The ISME Journal, Microbiome, SBB 等国际著名期刊上发表十余篇文章。
文涛

猜你喜欢
10000+:
系列教程:
专业技能:
一文读懂:
必备技能:
文献阅读
扩增子分析:
在线工具:
科研经验:
编程模板:
生物科普:
写在后面
学习16S扩增子、宏基因组科研思路和分析实战,关注“宏基因组”
点击阅读原文,跳转最新文章目录阅读
以上是关于ISME:南农沈其荣团队基于大数据准确预测土壤的枯萎病发生的主要内容,如果未能解决你的问题,请参考以下文章