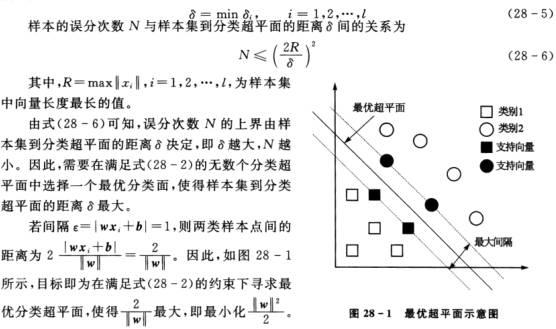
支持向量机SVM—分类
Posted 数模自愿分享交流
tags:
篇首语:本文由小常识网(cha138.com)小编为大家整理,主要介绍了支持向量机SVM—分类相关的知识,希望对你有一定的参考价值。




附录1 |
运行环境:Matlab2011a |
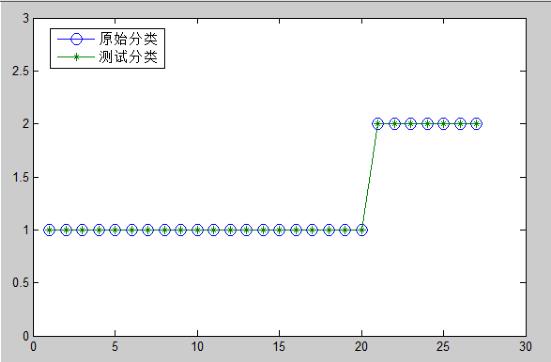
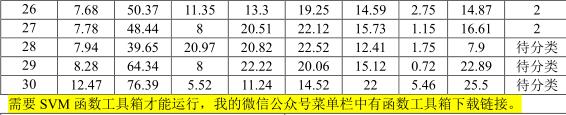
clc, clear a0=load('fenlei.txt'); %把表中x1...x8的所有数据保存在纯文本文件fenlei.txt中 a=a0'; b0=a(:,[1:27]); dd0=a(:,[28:end]); %提取已分类和待分类的数据 [b,ps]=mapstd(b0); %已分类数据的标准化 %mapstd按行逐行地对数据进行标准化处理, %将每一行数据分别标准化为均值为ymean(默认为0)、 %标准差为ystd(默认为1)的标准化数据,其计算公式是:y = (x-xmean)*(ystd/xstd) + ymean。 %如果设置的ystd=0,或某行的数据全部相同(此时xstd =0), %存在除数为0的情况,则Matlab内部将此变换变为y = ymean。 dd=mapstd('apply',dd0,ps); %待分类数据的标准化 group=[ones(20,1); 2*ones(7,1)]; %已知样本点的类别标号,即设置分类, %本程序设置前20个为第一类,21-27为2类 s=svmtrain(b',group) %训练支持向量机分类器 sv_index=s.SupportVectorIndices %返回支持向量的标号(分类器) beta=s.Alpha %返回分类函数的权系数(分类器) bb=s.Bias %返回分类函数的常数项(分类器) mean_and_std_trans=s.ScaleData %第1行返回的是已知样本点均值向量的相反数, %第2行返回的是标准差向量的倒数(分类器) check=svmclassify(s,b') %验证已知样本点 %将检验图画出,更直观些 x=1:27;%样本数据有27个 a=group'; b=check'; axis([0,28,0,3]);%设置坐标轴范围 plot(x,a,'-o',x,b,'-*') err_rate=1-sum(group==check)/length(group) %计算已知样本点的错判率 solution=svmclassify(s,dd') %对待判样本点进行分类 |
|
检验图 |
|
|
|
附录2 |
运行环境:Matlab2011a |
clc,clear a=load('cancerdata2.txt'); a(:,1)=[]; %删除第一列病例号 gind=find(a(:,1)==1); %读出良性肿瘤的序号 bind=find(a(:,1)==-1); %读出恶性肿瘤的序号 training=a([1:500],[2:end]); %提出已知样本点的数据 training=training'; [train,ps]=mapstd(training); %已分类数据标准化 group(gind)=1; group(bind)=-1; %已知样本点的类别标号 group=group'; %转换成列向量 xa0=a([501:569],[2:end]); %提出待分类数据 xa=xa0'; xa=mapstd('apply',xa,ps); %待分类数据标准化 s=svmtrain(train',group, 'Method','SMO', 'Kernel_Function','quadratic') %使用序列最小化方法训练支持向量机的分类器, %如果使用二次规划的方法训练支持向量机则无法求解 sv_index=s.SupportVectorIndices' %返回支持向量的标号 beta=s.Alpha' %返回分类函数的权系数 b=s.Bias %返回分类函数的常数项 mean_and_std_trans=s.ScaleData %第1行返回的是已知样本点均值向量的相反数, %第2行返回的是标准差向量的倒数 check=svmclassify(s,train'); %验证已知样本点 err_rate=1-sum(group==check)/length(group) %计算错判率 solution=svmclassify(s,xa'); %进行待判样本点分类 solution=solution' sg=find(solution==1) %求待判样本点中的良性编号 sb=find(solution==-1) %求待判样本点中的恶性编号 |
|
小编建了个群:139170798(国赛备战群),这里有海量数模资料汇总,自愿分享,加的时候请备注学校简称+小名。
以上是关于支持向量机SVM—分类的主要内容,如果未能解决你的问题,请参考以下文章