基于R语言pec包对Cox回归模型进行外部验证与评价
Posted 临床研究与医学统计
tags:
篇首语:本文由小常识网(cha138.com)小编为大家整理,主要介绍了基于R语言pec包对Cox回归模型进行外部验证与评价相关的知识,希望对你有一定的参考价值。
题记:这是我们临床预测模型构建的第12篇文章。感谢王子威贤弟赐稿,三人行必有我师!
1. 背景知识
在Logistic回归中我们可以由回归方程,根据患者的自变量取值预测其发生结局的概率,读者可以参考前述章节。在Cox回归中,很多人可能和笔者有相同的困惑,如何利用已经构建好的生存资料预测模型预测单个患者的生存概率?R中pec包中predictSurvProb()函数可以利用rms包cph()函数,survival包coxph()函数,survial包survfit()函数拟合的模型计算患者的生存概率。
predictSurvProb()函数的用法为predictSurvProb(object,newdata,times,…)
其中,object是survival::coxph()函数或rms::cph()函数已经拟合好的模型,newedata是一个data.frame(数据框结构)的数据集,其中行代表观测,列为预测模型需要用到的变量,times是一个向量,包含了想要预测的时间点。
2. 案例分析
下面我们利用TCGA数据库下载的232例肾透明细胞癌的临床数据进行实战操作。本数据集中共8个变量,death为结局变量,os为生存时间,age, gender, tnm, ssign, fuhrman为自变量。
3. R语言代码及结果解读
首先加载所需pec包及必要辅助的程序包,并读取数据Case_in_TCGA.csv。
library(dplyr)
library(rms)
## Warning: package 'Hmisc' was built under R version 3.5.2library(survival)
library(pec)
## Warning: package 'pec' was built under R version 3.5.2data <- read.csv("Case_in_TCGA.csv")将232例病人随机划分为训练集与验证集,其中data.1为训练集,data.2为验证集。
set.seed(1450)
x <- nrow(data) %>% runif()
data <- transform(data,sample=order(x)) %>% arrange(sample)
data.1 <- data[1:(nrow(data)/2),]
data.2 <- data[((nrow(data)/2)+1):nrow(data),]使用训练集data.1的116例拟合Cox回归模型。
cox1 <- cph(Surv(os,death)~age+gender+tnm+fuhrman+ssign, data=data.1, surv=TRUE)
cox2 <- cph(Surv(os,death)~tnm+ssign, data=data.1, surv=TRUE)需要设置预测生存概率的生存时间点,这里根据训练集所构建模型预测验证集每个患者第1、3、5年的生存概率。
t <- c(1,3,5)
survprob <- predictSurvProb(cox1,newdata=data.2,times=t)
head(survprob)## 1 3 5
## 117 0.9128363 0.7447740 0.6346714
## 118 0.8907077 0.6879995 0.5615877
## 119 0.9077517 0.7314529 0.6172421
## 120 0.9121396 0.7429388 0.6322600
## 121 0.8366330 0.5619540 0.4109773
## 122 0.8577605 0.6091122 0.4653860这个包的优秀之处除了其可以根据训练集构建的预测模型计算验证集中每个对象的生存概率,其还可以进一步在验证集中计算C-Index。前述章节我们介绍过C-Index的含义。此处简要罗列如下:C-Index,全称Concordance Index,主要用于反映预测模型的区分能力,考察模型是否可以进行准确预测区分。C-Index的定义很是简单,C-Index = 一致的对子数/有用的对子数。想象一下将所有研究对象随机的两两配对,N个研究对象,会产生N*(N-1)/2个对子数,若样本量N很大,则计算量巨大,必须借助计算机软件才能完成。我们首先找到其中一致的对子数作为分子。何谓一致对子数?以生存分析Cox回归分析为例,若实际生存时间较长的其预测的生存概率也较大,或者生存时间较短的其预测的生存概率也较小,即称为预测结果和实际结果一致,反之,则为不一致。然后我们在这么多对子中找到有用的对子数作为分母。何谓有用对子数?还是以生存分析Cox回归分析为例,所谓有用的对子数要求配对的两人中至少有一人发生了目标终点事件。也就是说,如果配对的两人在整个观察期内均未出现终点事件,则不计入分母。另外,还有两种情况也需要排除:
(1).若配对的两人中有一人发生了终点事件,另外一人因失访导致无法比较二人的生存时间,这种情况应该排除。
(2).配对的两人在同一时间死亡,也应该排除。找到了有用的对子数作为分母,那么如何确定分子呢?
pec包cindex()函数可以计算预测模型的C-Index并可通过交叉验证的方式评估各种回归建模策略对同一组数据区分度的高低。
使用在data.2中对模型的区分度C-Index进行验证。
c_index <- cindex(list("Cox(5 variables)"=cox1, "Cox(2 variables)"=cox2),
formula=Surv(os,death)~age+gender+tnm+fuhrman+ssign,
data=data.2,
eval.times=seq(1,5,0.1))
plot(c_index,xlim = c(0,5))上图表明Cox(2 variables)回归模型区分度略好于Cox(5 variables),但这个结果未进行交叉验证,可能不稳定,我们可进一步采用交叉验证的方法比较两个回归模型的区分度。
在data.2中对模型的区分度进行验证,同时使用bootstrap重抽样法进行交叉验证。
c_index <- cindex(list("Cox(5 variables)"=cox1, "Cox(2 variables)"=cox2),
formula=Surv(os,death)~fuhrman+ssign,
data=data,
eval.times=seq(1,5,0.1),
splitMethod="bootcv",
B=1000)
plot(c_index,xlim = c(0,5))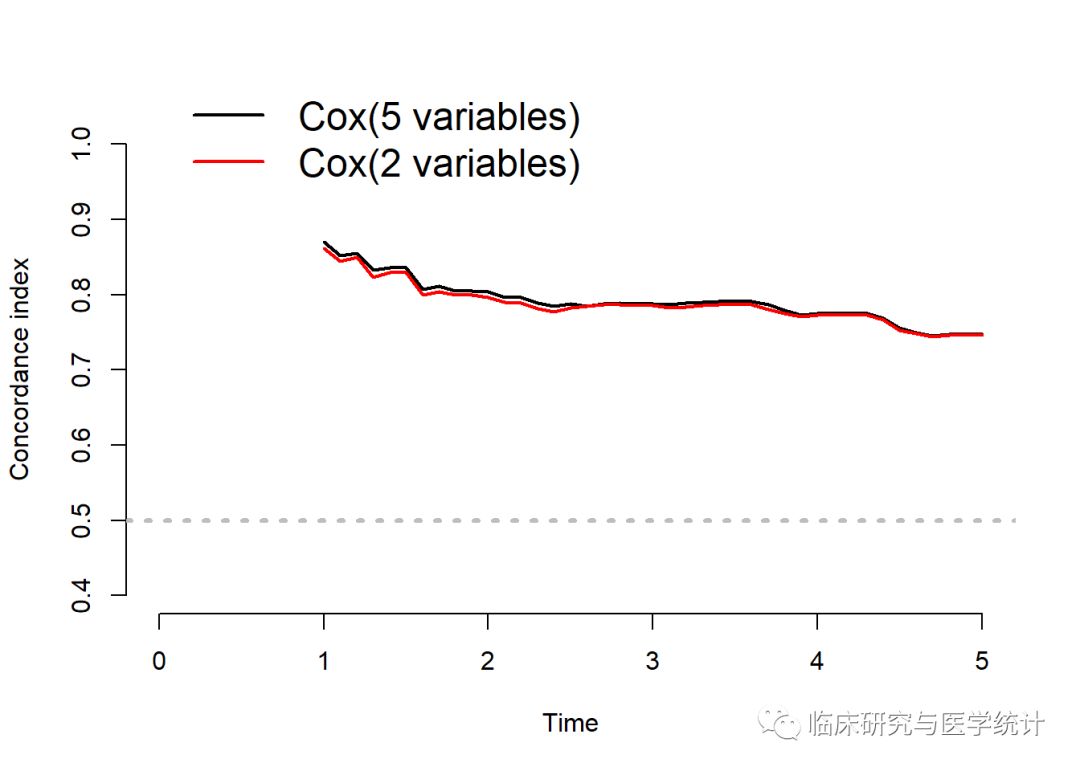
上图表明,两个Cox回归模型区分度差别并不大,但仅纳入2个变量的模型显然比纳入5个变量的模型更为简便。从实用性角度讲,当然模型越简便越好。
这个包的除了可以在验证集中计算每个对象的生存概率、计算C-Index,还可以在验证集中画校准曲线(Calibration plot)。以上几点在rms包中实现都很困难,pec包很好的解决了这些问题。前述章节我们介绍过Calibration plot的含义。此处简要罗列如下:Calibration指结局实际发生的概率和预测的概率的一致性。看起来有点让人费解,我们还是用本章开篇的这个例子,我们预测的100个研究对象的患病情况,不是指我们真用模型预测出来有病/无病,模型只给我们有病的概率,根据概率大于某个截断值(比如说0.5)来判断有病/无病。100个人,我们最终通过模型得到了100个概率,也就是100个0-1之间的数,我们将这100个数,按照从小到大排列,再依次将这100个人分成10组,每组10个人,实际的概率其实就是这10个人中发生疾病的比例,预测的概率就是每组预测得到的10个比例的平均值,然后比较这两个数,一个作为横坐标,一个作为纵坐标,就得到了一致性曲线图(Calibration Plot),也可计算这个图的95%区间范围。
下面我们使用pec包calPlot()函数演示如何在验证集中画Calibration Plot。而且我们可以把两个模型的校准曲线画在一个坐标系里,不得不再次夸以下这个包,真的很好用,想笔者之所想啊。
calPlot(list("Cox(5 variables)"=cox1,"Cox(2 variables)"=cox2),
time=3,#设置想要观察的时间点
data=data.2)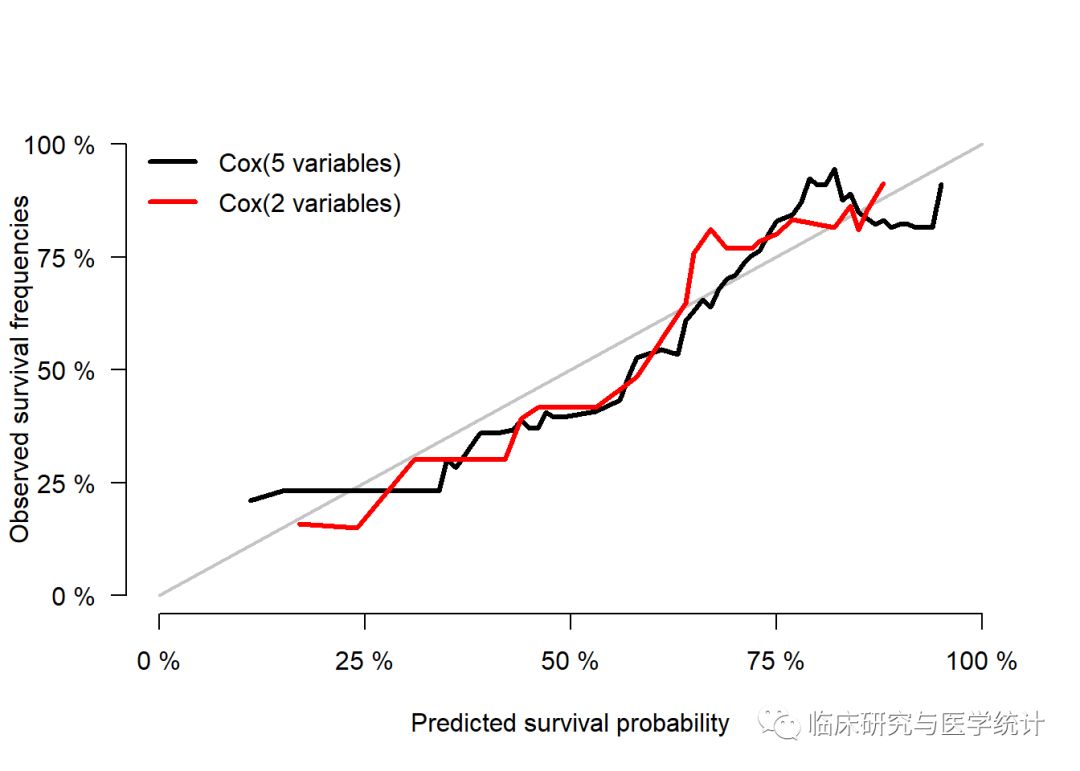
同样,我们可以对232例总体使用bootstrap法重抽样,对模型进行交叉验证。
calPlot(list("Cox(5 variables)"=cox1,"Cox(2 variables)"=cox2),
time=3,#设置想要观察的时间点
data=data,
splitMethod = "BootCv",
B=1000)4. 参考文献
[1]. https://cran.r-project.org/web/packages/pec/index.html
作者简介:王子威,海军军医大学附属长海医院泌尿外科研究生在读,R语言爱好者。
*扫描下图二维码或点击“阅读原文”查看周老师全部课程
以上是关于基于R语言pec包对Cox回归模型进行外部验证与评价的主要内容,如果未能解决你的问题,请参考以下文章
R语言survival包的coxph函数构建cox回归模型ggrisk包的ggrisk函数可视化Cox回归的风险评分图基于业务经验指定经验cutoff值(基于LIRI基因数据集)
R语言使用survival包的coxph函数构建cox回归模型使用ggrisk包的ggrisk函数可视化Cox回归的风险评分图(风险得分图)并解读风险评分图基于LIRI数据集(基因数据集)
R语言survival包coxph函数构建cox回归模型ggrisk包ggrisk函数可视化Cox回归的风险评分图使用cutoff包基于最小p值法方法计算最佳截断值(基于LIRI基因数据集)
R语言survival包的coxph函数构建cox回归模型ggrisk包的ggrisk函数可视化Cox回归的风险评分图使用title参数自定义图例轴标签的标题信息(基于LIRI基因数据集)
R语言使用survival包的coxph函数构建cox回归模型使用ggrisk包的ggrisk函数可视化Cox回归的风险评分图(风险得分图)使用风险得分的roc曲线的约登值(基于LIRI基因数据集