R语言 | 密度图
Posted 生物空间站
tags:
篇首语:本文由小常识网(cha138.com)小编为大家整理,主要介绍了R语言 | 密度图相关的知识,希望对你有一定的参考价值。
密度图
密度图是指使用不同颜色表示样本在绘图区域中分布情况的图像。
通常情况下,会根据两个变量将平面绘图区域分为非常多的子区域,之后以不同颜色表示落在该区域上样本的数目,以此来评估样本的分布情况。
下面会介绍几种在R中实现密度图的方法。
smoothScatter
smoothScatter是R自带的一个专门用于绘制平滑颜色密度图的函数。
首先还是先介绍该函数的绘图参数。
smoothScatter(x, y = NULL, nbin = 128, bandwidth,
colramp = colorRampPalette(c("white", blues9)),
nrpoints = 100, ret.selection = FALSE,
pch = ".", cex = 1, col = "black",
transformation = function(x) x^.25,
postPlotHook = box,
xlab = NULL, ylab = NULL, xlim, ylim,
xaxs = par("xaxs"), yaxs = par("yaxs"), ...)各参数意义:
x和y定义绘图的x和y轴坐标;
nbin定义对绘图区氛围多少个子区域;
bandwidth定义绘图向量的带宽,一般为默认
colramp定义密度图的绘图颜色;
nrpoints定义图像中绘制点的个数,如果需要绘制所有点,则设为Inf;
pch、cex和col定义图像中点的形状、大小和颜色;
transformation为密度尺度和颜色尺度的转换公式
postPlotHook定义绘图后的调整,默认是添加外框;
xlab和ylab为两个坐标轴的标签;
xlim和ylim为两个坐标轴的范围;
xaxs和yaxs的意义与par中函数意义相同。
绘制密度图需要两个同等元素数量并且互相对应的向量来定义x和y轴,也可以使用数据框中的两列,首先建立一个随机的绘图数据。
x <- c(rnorm(500,mean = 1,sd = 3))
y <- c(rnorm(500,mean = 2,sd = 0.8))
data <- data.frame(x,y)接下来绘制密度图。
smoothScatter(data$x,data$y,main = "Scatterplot colored by smoothed densities",
xlab = "",ylab = "")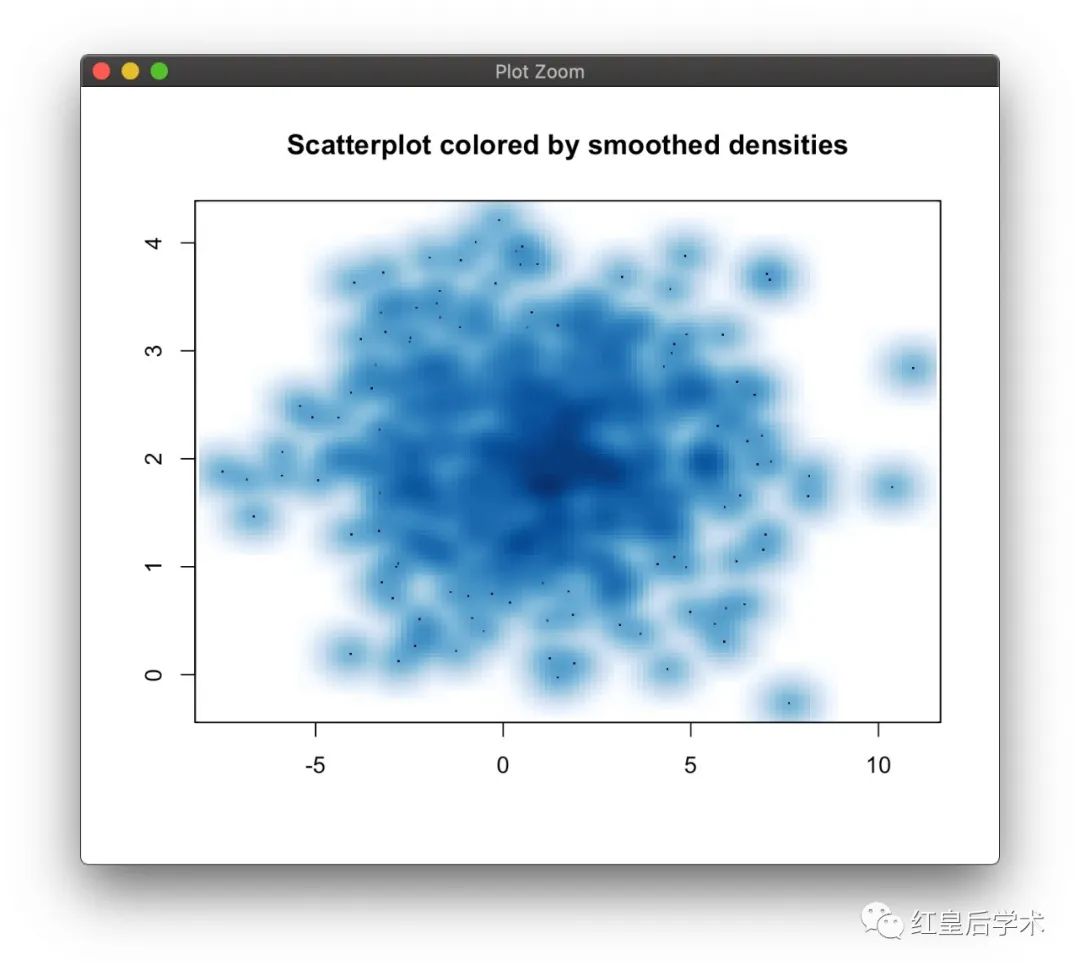
hexbin包
第二种方式是使用hexbin包,该包的主要绘图函数就是hexbin函数。
其特点是将绘图区域分成正六边形的形式,并且提供图例。
hexbin(x, y, xbins = 30, shape = 1,
xbnds = range(x), ybnds = range(y),
xlab = NULL, ylab = NULL, IDs = FALSE)各参数意义:
x和y为绘图的x和y轴坐标;
xbins定义将数据分箱的个数;
shape为绘图区域的比例,数值为yheight/xwidth;
xbands和ybands定义x和y轴的范围;
xlab和ylab为x和y轴的标签;
IDs定义是否返回单独的每个小格子的ID。
还是使用刚才生成的随机数据进行绘图。
install.packages("hexbin")library(hexbin)
plot(hexbin(data$x,data$y,xbins = 50,xlab = "",ylab = ""),
main = "Scatterplot colored by smoothed densities")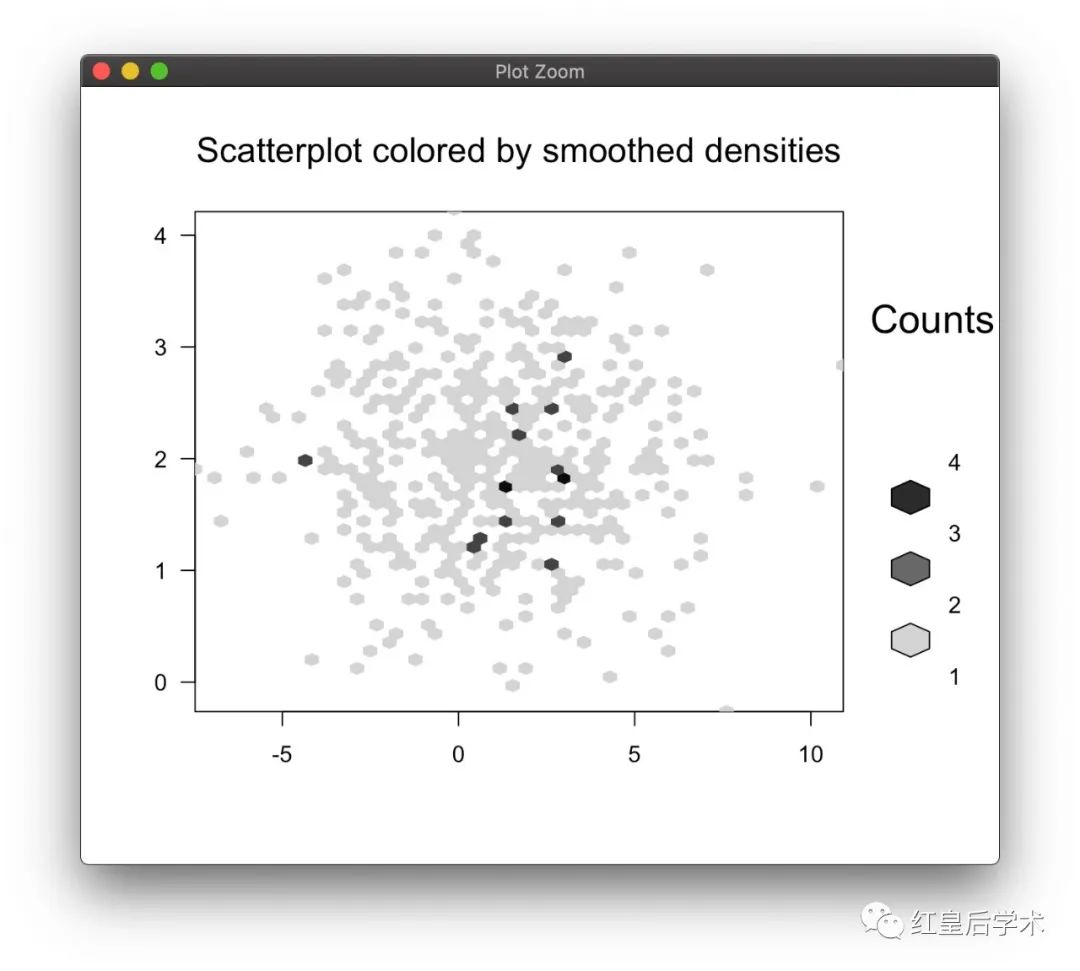
⚠️hexbin函数只是生成一个用于绘图的文件,还需要结合plot函数才能生成图像。
iplot函数
第三种方式是使用IDPmics包中的iplot函数。
iplot(x, y = NULL,
pixs = 1, zmax = NULL, ztransf = function(x){x},
colramp = IDPcolorRamp, cex = par("cex"),
main = NULL, d.main = 1, cex.main = par("cex.main"),
xlab = NULL, ylab = NULL, cex.lab = 1,
legend = TRUE, d.legend = 1,
cex.axis = par("cex.axis"), nlab.xaxis = 5, nlab.yaxis = 5,
minL.axis = 3, las = 1, border = FALSE,
oma = c(5,4,1,0)+0.1, mgp = c(2,0.5,0)*cex.axis, tcl = -0.3, ...
)各参数意义:
x和y定义绘图数据的x和y轴坐标,如果x为数据框或矩阵,则其前两列为x和y坐标,此时y要设置为NULL,x和y可以是数值型或因子向量;
pixs定义图像像素的大小,单位为mm;
zmax定义每个像素中可以绘制的最大数值;
ztransf定义数据尺度和颜色尺度的转换函数;
colramp定义绘图颜色;
cex定义图中文字的大小;
main、d.main和cex.main分别定义标题内容、标题和图像顶端的距离以及标题的大小;
xlab和ylab定义x和y轴的标签;
cex.lab定义标签的大小;
legend和d.legend定义是否显示图例和图例与图像右侧的距离;
cex.axis定义坐标轴上注释标签的大小;
nlab.xaxis和nlab.yaxis定义图像x和y轴标签的个数;
minL.axis定义因子变量水平的最小缩写长度;
las定义轴标签的方向;
border是否为图例中每一个单独的颜色绘制边框;
oma图像的外边界大小;
mgp定义轴标题、轴标签和轴线的距离;
tcl定义轴上刻度线的长度。
同样使用刚才生成的随机数据进行绘图。
install.packages("IDPmisc")library(IDPmisc)
iplot(data$x,data$y,main = "Scatterplot colored by smoothed densities",
xlab = "",ylab = "")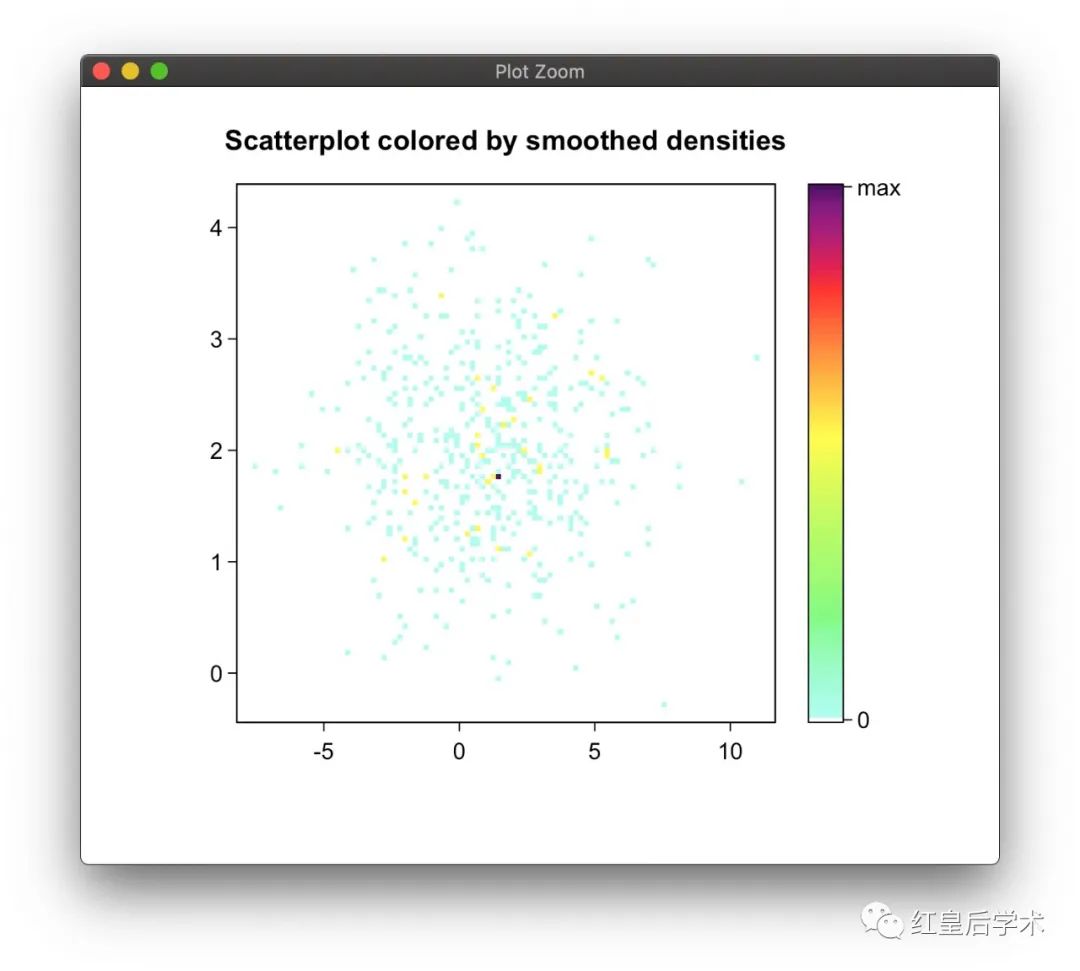
s.kde2d
第四种方法是ade4包的s.ked2d函数。
s.kde2d(dfxy, xax = 1, yax = 2, pch = 20, cpoint = 1, neig = NULL, cneig = 2,
xlim = NULL, ylim = NULL, grid = TRUE, addaxes = TRUE, cgrid = 1,
include.origin = TRUE, origin = c(0, 0), sub = "", csub = 1.25,
possub = "bottomleft", pixmap = NULL, contour = NULL,
area = NULL, add.plot = FALSE)各参数意义:
dfxy,绘图的数据框,至少含有两列;
xax,定义哪列数据为x轴;
yax,定义哪列数据为y轴;
pch,图中点的形状;
cpoint,图中点的大小,类似于cex;
neig,是否绘制相邻图;
cneig,相邻图的线宽;
xlim、ylim,x和y轴的范围;
grid,是否绘制背景网格;
addaxes,是否绘制坐标轴;
cgrid,网格线的线宽;
include.origin,是否绘制原点;
origin,原点的坐标;
sub,图例的文字
csub,图例文字的大小;
possub,图例的位置;
contour,通过一个4列的数据绘制等值线;
area,用于绘制等值线表面的数据;
add.plot,是否添加到现有图像中。
绘图示例。
library(ade4)if(!adegraphicsLoaded()) {
data(rpjdl)
coa1 <- dudi.coa(rpjdl$fau, scannf = FALSE, nf = 3)
s.kde2d(coa1$li)
}⚠️s.kde2d绘制密度图的目的与之前的3个方法不同,根据自身的需求合理选择
以上是关于R语言 | 密度图的主要内容,如果未能解决你的问题,请参考以下文章