师兄绝招 | 手把手教你R语言轻松搞定临床三线表
Posted 医科研
tags:
篇首语:本文由小常识网(cha138.com)小编为大家整理,主要介绍了师兄绝招 | 手把手教你R语言轻松搞定临床三线表相关的知识,希望对你有一定的参考价值。
按照流行病学和相关领域的标准做法是,期刊文章的第一张表格,我们通常称为“表1”,是一份表,列出按暴露程度分层的研究人群基线特征的描述性统计数据。这个包使得使用R生成这样一个表相当简单。输出格式是html(它的优点是很容易复制到Word文档中;Chrome浏览器工作得很好)。
实际上这个包还能够对表格有更多定制的功能,但这样用起来爽是需要代价的,更多的代码与CSS知识
官方文档参考资料(https://benjaminrich.github.io/table1/vignettes/table1-examples.html)
R包table1
if(!require(table1)) install.packages("table1",ask=F,update=F)
## Loading required package: table1
## Warning: package 'table1' was built under R version 3.6.1
##
## Attaching package: 'table1'
## The following objects are masked from 'package:base':
##
## units, units<-
require(table1)
Example1
使用boot包中的自带数据
melanoma数据集
library(boot)
## Warning: package 'boot' was built under R version 3.6.1
melanoma2 <- melanoma
head(melanoma)
## time status sex age year thickness ulcer
## 1 10 3 1 76 1972 6.76 1
## 2 30 3 1 56 1968 0.65 0
## 3 35 2 1 41 1977 1.34 0
## 4 99 3 0 71 1968 2.90 0
## 5 185 1 1 52 1965 12.08 1
## 6 204 1 1 28 1971 4.84 1
dim(melanoma)
## [1] 205 7
## input melanoma是一个数据框
## 对我们感兴趣的变量因子化
melanoma2$status <-
factor(melanoma2$status,
levels=c(2,1,3),
labels=c("Alive", # 第一个作为参考组
"Melanoma death",
"Non-melanoma death"))
可以来绘制一个表格试试,意思就是以感兴趣的因子作为分类status,同理这个status可以是任意大家感兴趣的变量。
格式为~感兴趣的基线变量|感兴趣的分类变量,data
table1(~ factor(sex) + age + factor(ulcer) + thickness | status, data=melanoma2)
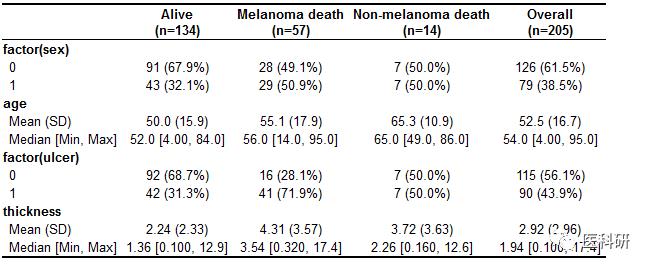
这样的表格已经可以了,但仍然可以改进
基线分类变量sex,ulcer没有很好的label,基线的连续型变量可以指定单位units,下面来improve
## 给分类变量sex指定标签
melanoma2$sex <-
factor(melanoma2$sex, levels=c(1,0),
labels=c("Male",
"Female"))
## 给分类变量ulcer指定标签
melanoma2$ulcer <-
factor(melanoma2$ulcer, levels=c(0,1),
labels=c("Absent",
"Present"))
## 给变量名指定标签
label(melanoma2$sex) <- "Sex"
label(melanoma2$age) <- "Age"
label(melanoma2$ulcer) <- "Ulceration"
label(melanoma2$thickness) <- "Thickness"
## 给连续型变量指定单位
units(melanoma2$age) <- "years"
units(melanoma2$thickness) <- "mm"
## 再增加overall统计量
table1(~ sex + age + ulcer + thickness | status, data=melanoma2, overall="Total")
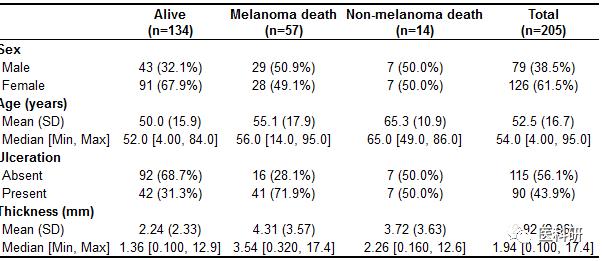
细节控制
labels <- list(
variables=list(sex="Sex",
age="Age (years)",
ulcer="Ulceration",
thickness="Thickness (mm)"),
groups=list("", "", "Death"))##表格上的第一级Death
# 重新给status命名标签,death放到上面去
levels(melanoma2$status) <- c("Alive", "Melanoma", "Non-melanoma")
#按想要的顺序顺序设置分组或列,
#Total放第一列,split分开status
strata <- c(list(Total=melanoma2), split(melanoma2, melanoma2$status))
# 添加渲染风格-连续型变量与分类变量展示不同
# 连续型渲染风格函数
my.render.cont <- function(x) {
with(stats.apply.rounding(stats.default(x), digits=2), c("",
"Mean (SD)"=sprintf("%s (± %s)", MEAN, SD)))
}
# 分类变量渲染风格
my.render.cat <- function(x) {
c("", sapply(stats.default(x), function(y) with(y,
sprintf("%d (%0.0f %%)", FREQ, PCT))))
}
## 结果
## groupsapn为分组的个数,1为Total, 1为Alive,以及2为Death
## 增加了Death的亚组
table1(strata, labels, groupspan=c(1, 1, 2),
render.continuous=my.render.cont, render.categorical=my.render.cat)
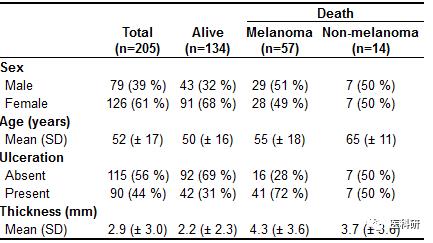
Example2
自己模拟一个数据
函数式编程
…把其余参数全部传递,…的参数传递到了sample函数
f <- function(x, n, ...) factor(sample(x, n, replace=T, ...), levels=x)
set.seed(427)
## 构造数据框
n <- 146
dat <- data.frame(id=1:n)
dat$treat <- f(c("Placebo", "Treated"), n, prob=c(1, 2)) # 2:1 randomization
dat$age <- sample(18:65, n, replace=TRUE)
dat$sex <- f(c("Female", "Male"), n, prob=c(.6, .4)) # 60% female
dat$wt <- round(exp(rnorm(n, log(70), 0.23)), 1)
dat$wt[sample.int(n, 5)] <- NA## 加入一些缺失值
head(dat)
## id treat age sex wt
## 1 1 Treated 18 Female 62.6
## 2 2 Treated 50 Male 57.4
## 3 3 Treated 37 Male 104.6
## 4 4 Treated 25 Female 55.5
## 5 5 Placebo 60 Female 58.4
## 6 6 Treated 44 Female 41.9
标签命名
单位
## 分类变量
label(dat$age) <- "Age"
label(dat$sex) <- "Sex"
label(dat$wt) <- "Weight"
label(dat$treat) <- "Treatment Group"
## 连续型变量
units(dat$age) <- "years"
units(dat$wt) <- "kg"
## 绘制默认表格
table1(~ age + sex + wt | treat, data=dat)
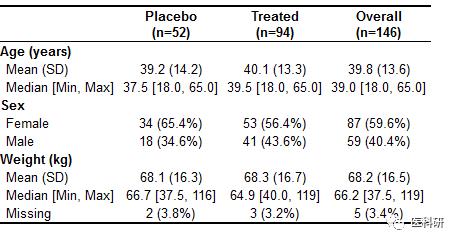
删除overall
table1(~ age + sex + wt | treat, data=dat, overall=F)
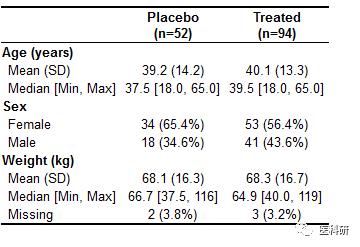
轻松进行多个变量分类,一键定制
table1(~ age + wt | treat*sex, data=dat)
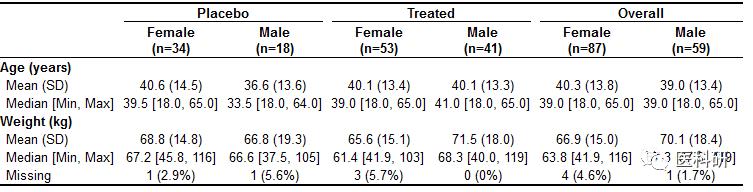
改变顺序
table1(~ age + wt | treat*sex, data=dat)
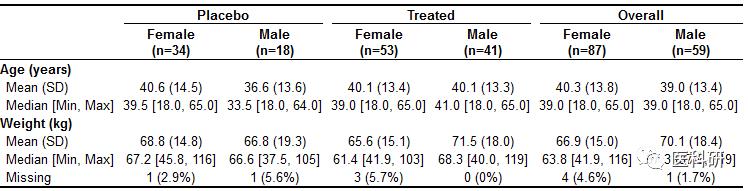
无感兴趣变量统计表格
table1(~ treat + age + sex + wt, data=dat)
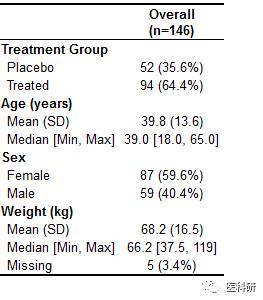
更复杂的定制
## 给原数据增加一个dose列
dat$dose <- (dat$treat != "Placebo")*sample(1:2, n, replace=T)
## 给dose加标签
dat$dose <- factor(dat$dose, labels=c("Placebo", "5 mg", "10 mg"))
## strata定制
## split指定按dose分亚组
strata <- c(split(dat, dat$dose), ##dose分组
list("All treated"=subset(dat, treat=="Treated")), ## all treated组
list(Overall=dat))## overall
labels <- list(
variables=list(age=render.varlabel(dat$age),
sex=render.varlabel(dat$sex),
wt=render.varlabel(dat$wt)),
groups=list("", "Treated", ""))## 一级分组标签
## groupspan二级分组告诉你标题栏的线包括几个变量
## 对应groups
table1(strata, labels, groupspan=c(1, 3, 1))
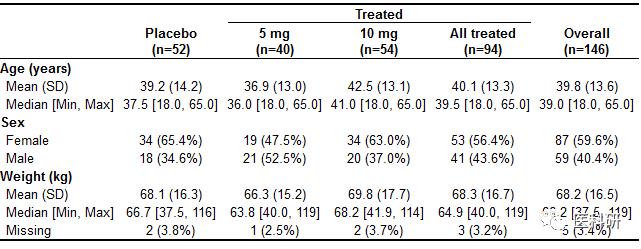
显示不同变量的不同统计数据
例如下面的渲染风格
age展示Median
wt展示mean
rndr <- function(x, name, ...) {
if (!is.numeric(x)) return(render.categorical.default(x))
what <- switch(name,
age = "Median [Min, Max]",
wt = "Mean (SD)")
parse.abbrev.render.code(c("", what))(x)
}
table1(~ age + sex + wt | treat, data=dat,
render=rndr)
改变表格的样式
内置了多个渲染风格
zebra: alternating shaded and unshaded rows (zebra stripes)
grid: show all grid lines
shade: shade the header row(s) in gray
times: use a serif font
center: center all columns, including the first which contains the row labels
## 更换表格风格,用topclass参数设置
## zebra似乎不错
table1(~ age + sex + wt | treat, data=dat, topclass="Rtable1-zebra")
增加一列pvalue
实际上这个功能本身是没有的,但是可以通过添加一个渲染来实现
library(MatchIt)
## Warning: package 'MatchIt' was built under R version 3.6.1
data(lalonde)
head(lalonde)
## treat age educ black hispan married nodegree re74 re75 re78
## NSW1 1 37 11 1 0 1 1 0 0 9930.0460
## NSW2 1 22 9 0 1 0 1 0 0 3595.8940
## NSW3 1 30 12 1 0 0 0 0 0 24909.4500
## NSW4 1 27 11 1 0 0 1 0 0 7506.1460
## NSW5 1 33 8 1 0 0 1 0 0 289.7899
## NSW6 1 22 9 1 0 0 1 0 0 4056.4940
## 分类变量
lalonde$treat <- factor(lalonde$treat, levels=c(0, 1, 2), labels=c("Control", "Treatment", "P-value"))
lalonde$black <- factor(lalonde$black)
lalonde$hispan <- factor(lalonde$hispan)
lalonde$married <- factor(lalonde$married)
lalonde$nodegree <- factor(lalonde$nodegree)
lalonde$black <- as.logical(lalonde$black == 1)
lalonde$hispan <- as.logical(lalonde$hispan == 1)
lalonde$married <- as.logical(lalonde$married == 1)
lalonde$nodegree <- as.logical(lalonde$nodegree == 1)
##连续变量
label(lalonde$black) <- "Black"
label(lalonde$hispan) <- "Hispanic"
label(lalonde$married) <- "Married"
label(lalonde$nodegree) <- "No high school diploma"
label(lalonde$age) <- "Age"
label(lalonde$re74) <- "1974 Income"
label(lalonde$re75) <- "1975 Income"
label(lalonde$re78) <- "1978 Income"
units(lalonde$age) <- "years"
rndr <- function(x, name, ...) {
if (length(x) == 0) {
y <- lalonde[[name]]
s <- rep("", length(render.default(x=y, name=name, ...)))
if (is.numeric(y)) {
p <- t.test(y ~ lalonde$treat)$p.value
} else {
p <- chisq.test(table(y, droplevels(lalonde$treat)))$p.value
}
s[2] <- sub("<", "<", format.pval(p, digits=3, eps=0.001))
s
} else {
render.default(x=x, name=name, ...)
}
}
rndr.strat <- function(label, n, ...) {
ifelse(n==0, label, render.strat.default(label, n, ...))
}
## 绘图
table1(~ age + black + hispan + married + nodegree + re74 + re75 + re78 | treat,
data=lalonde, droplevels=F, render=rndr, render.strat=rndr.strat, overall=F)
往期精彩合集
| | |
| |
以上是关于师兄绝招 | 手把手教你R语言轻松搞定临床三线表的主要内容,如果未能解决你的问题,请参考以下文章