Perl引用介绍
Posted 百迈客医学
tags:
篇首语:本文由小常识网(cha138.com)小编为大家整理,主要介绍了Perl引用介绍相关的知识,希望对你有一定的参考价值。
为什么使用引用?
引用是处理复杂数据结构、面向对象编程和精美子例程的基础。
perl的标量变量只能保存单个数值;数组是标量的有序集合;而哈希是标量的无序集合,字符串作为键。尽管标量可以是任意字符串,但是该字符串允许我们在数组或者数列中对复杂的数据进行编码,这三种数据类型都不适合用于表示复杂的数据关系。此时,面对复杂的数据关系,我们就要用到引用。在平时的工作中常用的引用是数组和哈希的引用,有了这俩,我们可以处理复杂的数据关系。下面,小编就依次讲解这两种引用。
数组引用
perl中反斜杠“”有很多含义,当放在数组前面的时候就表示对数组的取引用,比如@fruits,表示对数组@fruits的引用。如下:
1.my @fruits=(apple,cherry,banana,orange,grape); #数组
2.my $ref_fruits=@fruits; #标量表示对数组的引用
左右滑动查看
那么我们如何取得引用里的元素呢?仔细观察@fruits数组,我们可以发现该数组包含两部分:@符合和数组名称。同样的道理,fruits[0]是由和数组名称fruits以及元素所在的小标构成。我们可以将数组的任意引用放入大括号中,用于替换数组名称,最后以访问原始数组的方法作为结束。例如,下面两行都指向同一个数组:
1.@fruits
2.@{$ref_fruits}
而以下两种方式都指向数组的第一个元素
1.$fruits[0] #apple
2.${$ref_fruits}[0] #apple
哈希引用
我们也可以使用反斜杠“”对哈希进行引用,例如:
1.my %Amy_info=(
2.name => 'Amy',
3.hat => 'white',
4.shirt =>'red',
5.positoion =>'home',
6.); #定义哈希
7.my $ref_Amy_info=\%Amy_info;#对哈希的引用
左右滑动查看
我们也能够对哈希引用进行解引用操作以获取初始数据,操作策略和对数组解引用一致。我们按照没有使用引用的方式编写读取哈希中数据的语法,然后用一对大括号包围着的引用名称替换哈希名称。如下,都表示提取name键对应的值:
1.my $name=$Amy_info{'name'};
2.my $name=${$ref_Amy_info}{'name'};
匿名数组哈希引用
在处理数据的过程中,有时我们会创建临时的数组和哈希的名称,但当数据较复杂的时候,继续创建这些哈希和数组的名称就比较困难,而且我们必须记住这些变量的名称。为了避免这种麻烦,我们在读取数据创建数组和哈希的时候,可以创建匿名数组和哈希,直接对这些匿名数组和哈希进行引用就好。
我们可以使用一个方括号创建匿名数组,上文中对数组fruits的引用,我们可以省略命名一个fruits数组,可以创建一个匿名数组直接对其进行引用:
1.my $ref_fruits=[apple,cherry,banana,orange,grape)];
左右滑动查看
同样,我们可以使用大括号直接构造一个匿名哈希,从而省去中间临时变量的名字。例如下面的代码,我们可以省去中间变量%Amy_info,直接对匿名哈希进行引用。
1.my $ref_Amy_info={
2.name => 'Amy',
3.hat => 'white',
4.shirt =>'red',
5.positoion =>'home',
6.};
小试牛刀

自动带入和匿名数组引用
说了这么多,下面小编将带领大家进行实战练习。我们构造一个文件:第一列是miRNA,第二列是预测到的靶基因,三四列代表两款软件是否预测到该靶向关系。现在我们需要将每个miRNA对应的靶基因放在一起,此时我们就需要用到匿名数组。示例文件如下:
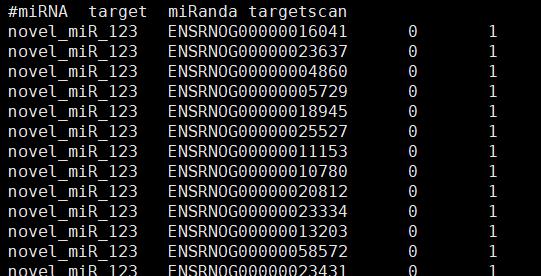
匿名数组的代码如下:
1.#/user/bin/perl
2.my($file,$out)=@ARGV;
3.open(IN,"$file"); #读取靶向关系预测文件
4.open(OUT,">$out"); #重定向输出文件
5.print OUT"miRNA target_gene "; #打印输出文件的标题行
6.my %hash;
7.while(<IN>){
8.chomp;
9.next if(/^#/);#跳过首行
10.my($miRNA,$gene)=(split/ /)[0,1];提取miRNA和靶基因ID
11.$hash{$miRNA}=[] unless $hash{$miRNA};#哈希嵌套匿名数组
12.push @{$hash{$miRNA}},$gene; #向匿名数组中添加元素
13.};
14.foreach my $miRNA(sort keys %hash){读取哈希的键值miRNA
15.print OUT "$miRNA ",join(",",@{$hash{$miRNA}})," "; #输出匿名数组
16.}
17.close IN;
18.close OUT;
左右滑动查看
通过以上代码,实现了我们的目的,输出文件如下:
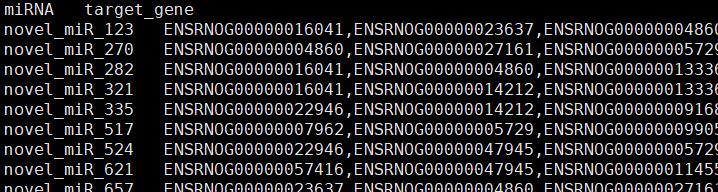
自动带入和匿名哈希引用
还是以上述的示例文件为例,输出两款软件都预测到的靶向关系对,代码如下:
1.#/user/bin/perl
2.my($file,$out)=@ARGV;
3.open(IN,"$file"); #读取靶基因预测文件
4.open(OUT,">$out"); #重定向输出文件
5.print OUT"miRNA target_gene "; #输出文件标题行打印
6.my $hash; #定义哈希的标量引用
7.while(<IN>){
8.chomp;
9.next if(/^#/); #跳过输入文件第一行
10.my@a=split/ /; #以 分割文件
11.$hash->{$a[0]}{$a[1]}=$a[2]+$a[3];#建立$hash对二层匿名哈希的引用
12.};
13.foreach my $miRNA(sort keys%{$hash}){ #第一层匿名哈希的解引用
14.foreach my $gene (sort keys %{$hash->{$miRNA}}){ #二层匿名哈希的解引用
15.if($hash->{$miRNA}{$gene}==2){#判断关系对值是否等于2
16.print OUT"$miRNA $gene "; #等于2则输出关系对
17.}
18.}
19.}
20.
21.close IN; #关闭文件句柄IN
22.close OUT; #关闭文件句柄OUT
左右滑动查看
下图就是输出两款软件都预测到的靶向关系预测结果。
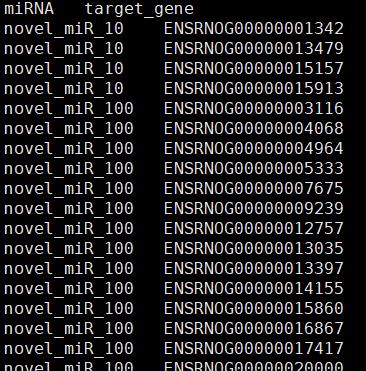
上述哈希匿名引用的代码,是小编为了凑匿名哈希故意这样写代码,以上的代码可以优化,大家可以思考一下在此代码的基础上如何优化,引入匿名数组,输出miRNA及其符合条件的靶向基因的集合。
好啦,关于perl的引用小编就介绍到这,不知大家学会了没有,希望此篇文章能对大家有所帮助。
以上是关于Perl引用介绍的主要内容,如果未能解决你的问题,请参考以下文章