自然语言处理加速ACMG解读:以顾卫红老师的帖子“PLA2G6基因的2个变异”为例
Posted 生物信息学与机器学习
tags:
篇首语:本文由小常识网(cha138.com)小编为大家整理,主要介绍了自然语言处理加速ACMG解读:以顾卫红老师的帖子“PLA2G6基因的2个变异”为例相关的知识,希望对你有一定的参考价值。
个人介绍:懂点遗传、生信、机器学习,开发了明码生物的“康码”和云健康基因的“GenoMe”两款全基因组检测产品,熟悉WGS/肿瘤/ctDNA/甲基化分析,开发了基于Bayes原理的GWAS风险评估模型和基于xgboost框架的致病性评估工具。喜欢技术,喜欢探索!
背景:
上次写的解读“PLA2G6基因的2个变异”的文章,有朋友说ACMG解读不够详细,这次补充了容易排除的部分,便于大家理解,小节部分增加了类似分析的流程。顾卫红老师的帖子截图如下:
生信分析
看到这个帖子和大家激烈的讨论,笔者也做了一点分析:
首先,通过查阅OMIM和GHR数据库,得知PLA2G6基因与“Neurodegeneration ”相关,在患有婴儿神经轴索营养不良症的人群中,至少发现了50个PLA2G6基因突变,这是一种导致智力残疾和运动问题的进行性神经系统疾病。 PLA2G6基因中的突变严重损害PLA2组VI酶的功能。 PLA2组VI酶功能受损可破坏细胞膜并促使轴突中称为球体的结构发生肿胀,所述的轴突是从神经元延伸并将冲动传递到肌肉和其他神经元的纤维。尽管目前还不清楚这种酶的功能如何变化引发婴儿神经轴索营养不良症的体征和症状,但是在这种疾病以及称为泛酸激酶相关性神经退行性疾病的类似疾病中都见到了磷脂代谢问题。这些疾病以及更常见的阿尔茨海默病和帕金森病也与脑铁代谢的变化有关。研究人员正在研究磷脂缺陷,脑铁和神经细胞损伤之间的联系,但尚未确定一些具有婴儿神经轴索营养不良的个体如何发生铁蓄积可能导致这种疾病的特征。
然后做了主要的数据库和评估软件的注释,结果如下:
| gene | cdna.variant | _id | HGMD | clinvar.omim | dbnsfp.clinvar.clinsig | dbnsfp.clinvar.rs | dbnsfp.clinvar.trait | dbnsfp.interpro_domain | dbsnp.gmaf | gnomad_exome.af.af | gnomad_exome.af.af_eas | snpeff.ann | cadd.phred | dbnsfp.dann.rankscore | dbnsfp.revel.rankscore | dbnsfp.m_cap_score.pred |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| chr22:g.38541632C>A | 0.0002 | MODERATE | 24.5 | 0.71035 | 0.65703 | D | ||||||||||
| PLA2G6 | c.238G>A | chr22:g.38541632C>T | DM | 603604.0008 | 5 | rs121908685 | Neurodegeneration with brain iron accumulation 2b | 0.0002 | 8.20E-06 | 5.83E-05 | MODERATE | 25.2 | 0.95778 | 0.74609 | D | |
| PLA2G6 | c.1634A>G | chr22:g.38516874T>C | DM | 4 | rs121908681 | Iron accumulation in brain | Acyl transferase/acyl hydrolase/lysophospholipase|Patatin-like phospholipase domain | 2.44E-05 | 0.000348 | MODERATE | 27.5 | 0.83868 | 0.91658 | D | ||
| chr22:g.38516874T>G | DM | 603604.0002 | 5 | rs121908681 | Neurodegeneration with brain iron accumulation 2b | Acyl transferase/acyl hydrolase/lysophospholipase|Patatin-like phospholipase domain | MODERATE | 29.2 | 0.81622 | 0.97402 | D |
由表可以看出,这2个位点,每个都是复等位基因变异,我们要研究的是中间2行,先看第二行c.238G>A,该变异在OMIM中有收录,增强了致病的可信度;clinvar描述与“Neurodegeneration with brain iron accumulation 2b”相关;人群频率有3列,都很低,万分之一级别;snpeff评估'MODERATE',为中等影响,4个集成软件CADD/DANN/MCAP/REVEL评估都偏向致病性。
第三行c.1634A>G变异,该变异在OMIM中没有收录,不过该位点的另外一个变异(第4行)有收录;除了蛋白结构域受到影响外,其他与第二行c.238G>A变异相似。
我们查看OMIM数据库,得知PLA2G6基因为隐性遗传,2个杂合变异有可能导致发病,跟患者表型似乎也吻合。
自然语言处理挖掘文献
对于基因的临床解读,我们要慎之又慎,于是乎又做了下基于自然语言处理的文献挖掘,对pubmed的所有文献,通过机器学习找到了这2个变异的文章,分别找到2篇和3篇,说明该变异确实研究不多,截图如下:
对于第一个变异c.238G>A,第一篇文章在1个家庭中发现1个纯合变异患者,第二篇文章发现一个该变异的患者,但没说明纯合变异还是杂合变异:

对于第二个变异c.1634A>G,第一篇文章指出该变异是之前新发现的没有解读的变异再次在"Neurodegeneration患者”中被发现,这次不同的是,与另外一个罕见变异c.1077G>A一起引起了新的移码变异c.1074_1077del.GTCG,该移码变异引起了可变剪切的发生,这或许才是真正的致病原因。
后2篇文章则说该位点纯合变异c.1634A>G在巴基斯坦很常见,由于突变异质性,没有发现该变异是突变热点的证据,不能说明该变异具有直接的基因型-表型关系。
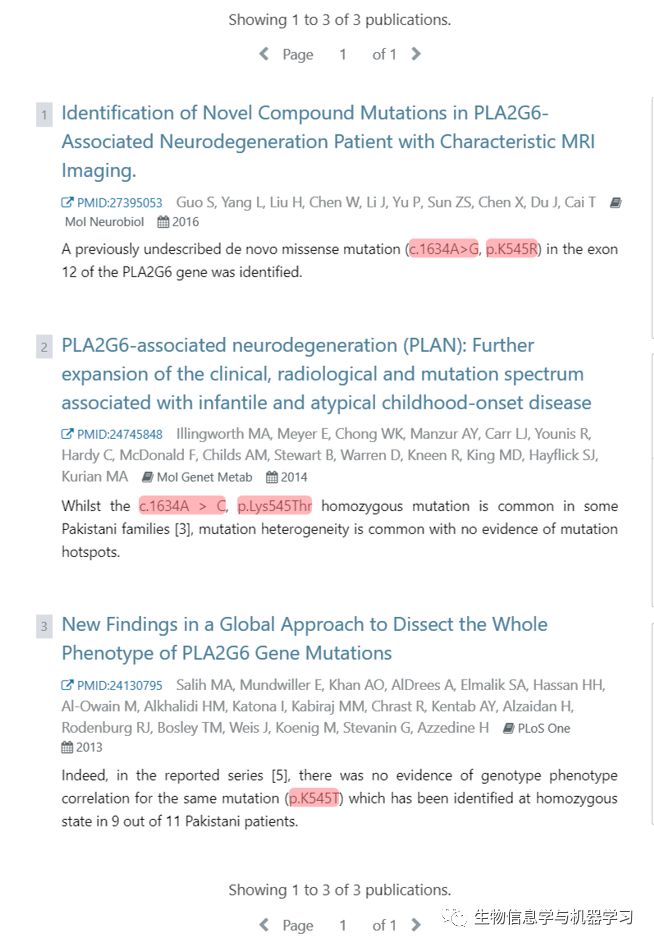
关于文献挖掘的几篇文章,读者如有兴趣,可以下载看下全文。
ACMG分级标准分析
为什么最后才做ACMG分析呢?因为ACMG指南不具有可操作性,只有上面的生信分析和文献挖掘做完后,才能对应到ACMG的致病分级。下面说下ACMG怎么应用到这2个变异的分析,我们按分级高低逐个过下:
PVS1:要求为在一个已知机制的疾病中,关键基因发生丧失功能的变异,包括无义突变、移码突变、剪接位点附近1-2位核苷酸的变异、起始密码子变异、单个或多个外显子缺失。PLA2G6基因机制未明,不符合PVS1。
PS1:要求为已知致病相关的非同义突变。由前面文献挖掘可以看出,第一个变异c.238G>A是非同义突变,并且在2个家庭中各发现了一名患者,可能符合PS1标准;再加上HGMD/OMIM/CLINVAR三个数据库均指出为致病变异,就更增加了可信度,要知道HGMD/OMIM可都是专家校正过的数据库;人群频率极低,4大集成软件评估致病,又增加了可信度。所以变异c.238G>A很可能是致病变异。
对于第二个变异c.1634A>G,文献挖掘不支持该变异与疾病相关,HGMD指出该变异为致病变异,参考的文献正是该变异的第一篇文献,或许HGMD的整理人员疏忽了。OMIM没有收录该变异,clinvar收录的是该变异与脑铁累积相关,也不是直接致病;蛋白结构功能显示该变异与酰基转移酶/酰基水解酶/溶血磷脂酶-PATP样磷脂酶结构域变化相关,磷脂代谢问题与神经退行性疾病相关,不过机理还在研究之中。
PS2:要求患者的新发变异,且无家族史。该变异在多个非患者家庭发现,显然不符合标准。
PS3:已有明确体内体外功能实验明确会导致基因功能受损的变,功能实验需要是可验证的,可重复的。显然也不符合。
PS4:要求变异出现在患病群体中的频率显著高于对照群体。该变异在多个非患者家庭发现,显然也不符合标准。
PM1:要求位于突变热点,第二个变异c.1634A>G文献挖掘不在突变热点,也不符合PM1。
PM2:要求在正常对照人群中未发现的位点(或者是隐性遗传病检测中发现的极低频位点),显然也不符合标准。
PM3:要求在隐性遗传病中,在另一条染色体上又发现了同一基因的一个已知致病突变,这种情况必须通过患者父母或后代验证。这条需要家系分析了,建议对患者父母检测PLA2G6基因。(这里有个疑问,患者本身携带该基因的另外一个致病位点c.238G>A,算不算符合PM3?)
PM4:非移码插入缺失或终止密码子丧失突变导致蛋白质长度变化。变异c.1634A>G没有引起蛋白长度变化,显然也不符合这条标准。
PM5:要求新的错义突变导致一个致病的氨基酸位置变异,但突变成另一个未报道过的形式。鉴于该位点的2种变异都报道过,也不符合。
PM6:要求缺乏父母样本验证的新变异。文献挖掘的后2篇文章有家系是父母验证的正常人群,显然也不符合。
PP1:要求已知致病基因在家系患病成员中共分离的变异位点,并与患者症状相符。文献挖掘支持这一点,加上蛋白结构功能域确实受到影响,所以该变异符合PP1,即支持性变异类型。
备注:
对于第一个变异c.238G>A,clingen数据库里则显示该变异还有可能位于可变剪切区域ENST00000430886.5:c.210-2337G>T,该变异解读:位于下游最近的外显子的边界位置为210,变异位点在内含子3’端开始的第2337个碱基,不过这种变异类型不常见,在该位点的所有变异类型中只占5%,截图展示了该变异的所有类型(VEP注释):
小结
经过上面的生信分析、文献挖掘和ACMG指南分析,我们总结出遗传病诊断的思路是:
1、先研究基因,可以查阅OMIM/GHR数据库,确定显隐性遗传模式和致病机理。
2、再做生信分析:对变异进行HGMD/OMIM/clinvar/uniprotKB四大知识库注释,再注释interpro_domain蛋白结构域,最后注释到3大频率数据库1KG/gnomad/Exome和5大功能预测软件snpeff/CADD/DANN/MCAP/REVEL。
3、接着通过自然语言处理挖掘文献:通过自然语言处理精准定位变异位置,快速理解文献。
4、最后ACMG指南分析:结合生信分析和文献挖掘,按照致病性强弱的顺序,逐个排查。
结论
推测1:发现第一个变异c.238G>A致病,第二个变异c.1634A>G为支持性,鉴于PLA2G6基因隐性遗传,两个杂合变异或许真的会导致发病。
推测2:受第二个变异的第一篇文献启发,两个杂合变异,或许会引起可变剪切,进而导致发病。
以上是关于自然语言处理加速ACMG解读:以顾卫红老师的帖子“PLA2G6基因的2个变异”为例的主要内容,如果未能解决你的问题,请参考以下文章