PyOD主要算法(KNN、IForest 和 MCD)的原理及使用
Posted
tags:
篇首语:本文由小常识网(cha138.com)小编为大家整理,主要介绍了PyOD主要算法(KNN、IForest 和 MCD)的原理及使用相关的知识,希望对你有一定的参考价值。
参考技术Ahttps://pyod.readthedocs.io/en/latest/pyod.models.html
Python Outlier Detection(PyOD)是当下最流行的Python异常检测工具库(toolkit)。该工具库的主要亮点包括:
对于特征空间中的一个样本,如果与之最相似的(即特征空间中距离最近的)k个样本中的大多数都属于某一类别,则该样本的分类结果也是这个类别。
https://www.cnblogs.com/lesleysbw/p/6074662.html
① 什么叫做KD_tree
K:K邻近查询中的k;D:空间是D维空间(Demension)tree:二叉树
② 建树过程
K-D tree的建立就是分裂空间的过程
首先,我们对整个区间 [1 , 15] 建树:先计算区间中所有点在第一维(也就是 x 坐标)上的方差:
平均值 : ave_1 =5.4
方差 : varance_1 =9.04
再计算区间中所有点在第二维(也就是 y 坐标)上的方差:
平均值:ave_2 =6.8
方差:varance_2 =10.96
明显看见,varance_2 > varance_1 ,那么我们在本次建树中, 分裂方式 :split_method =2 , 再将所有的点按照第2维的大小 从小到大排序 ,得到了新的点的一个排列:
(4,2) (1,4) (5,8) (7,9) (10,11)
取中间的点作为分裂点 sorted_mid =(5,8)作为根节点,再把区间 [1 , 2] 建成左子树 , [4 , 5] 建成右子树,此时,直线 : y = 8 将平面分裂成了两半,前面一半给左儿子,后面一半给了右儿子,如图:
建左子树 [1, 3] 的时候可以发现,这时候 第一维的方差大 ,分裂方式就是1 ,把区间 [ 1, 2 ] 中的点按照 第一维 的大小,从小到大排序 ,取 中间点(1,4) 根节点,再以区间 [ 2, 2] 建立右子树 得到节点 (4,2)
建右子树 [4 , 5] 的时候可以发现,这时还是第一维的方差大, 于是,我们便得到了这样的一颗二叉树 也就是 K-D tree,它把平面分成了如下的小平面, 使得每个小平面中最多有一个点 :
③ 查询过程:
查询,其实相当于我们要将一个点“添加”到已经建好的 K-D tree 中,但并不是真的添加进去,只是找到他应该 处于的子空间 即可,所以查询就显得简单的。
每次在一个区间中查询的时候,先看这个区间的 分裂方式 是什么,也就是说,先看这个区间是按照哪一维来分裂的,这样如果这个点对应的那一维上面的值比根节点的小,就在根节点的左子树上进行查询操作,如果是大的话,就在右子树上进查询操作。
每次回溯到了根节点(也就是说,对他的一个子树的查找已经完成了)的时候,判断一下,以该点为圆心,目前 找到的最小距离为半径 ,看是否和分裂区间的那一维所构成的平面相交,要是相交的话,最近点可能还在另一个子树上,所以还要再查询另一个子树,同时,还要看能否用根节点到该点的距离来更新我们的最近距离。为什么是这样的,我们可以用一幅图来说明:
https://github.com/YinghongZhang/BallTree-MIPS
① 原理
为了改进KDtree的二叉树树形结构,并且沿着笛卡尔坐标进行划分的低效率,ball tree将在一系列嵌套的超球体上分割数据。也就是说: 使用超球面而不是超矩形划分区域 。虽然在构建数据结构的花费上大过于KDtree,但是在 高维 甚至很高维的数据上都表现的很高效。
球树递归地将数据划分为 由质心C和半径r定义的节点 ,使得节点中的每个点都位于由r和C定义的超球内。通过使用三角不等式来减少邻居搜索的候选点数量。
② 建树过程
选择一个距离当前圆心最远的观测点A,和距离A最远的观测点B,将圆中所有离这两个点最近的观测点都赋给这两个簇的中心,然后计算每一个簇的中心点和包含所有其所属观测点的最小半径。对包含n个观测点的超圆进行分割,只需要线性的时间。
③ 查询
使用ball tree时,先自上而下找到包含target的叶子结点(c, r),从此结点中找到离它最近的观测点。这个距离就是 最近邻的距离的上界 。检查它的 兄弟结点 中是否包含比这个上界更小的观测点。方法是: 如果目标点距离兄弟结点的圆心的距离d > 兄弟节点所在的圆半径R + 前面的上界r,则这个兄弟结点不可能包含所要的观测点 。否则,检查这个兄弟结点是否包含符合条件的观测点。
用一个随机超平面来切割数据空间, 直到每个子空间里面只有一个数据点为止。切割次数的多少可用来区分异常。
https://www.jianshu.com/p/5af3c66e0410
iForest 由t个iTree孤立树组成,每个iTree是一个二叉树,其实现步骤如下:
可以看到d最有可能是异常,因为其最早就被孤立(isolated)了。
获得t个iTree之后,iForest 训练就结束,然后我们可以用生成的iForest来评估测试数据了。对于一个训练数据x,我们令其遍历每一棵iTree,然后计算x最终落在每个树第几层(x在树的高度),得到x在每棵树的高度平均值。获得每个测试数据的average path length后,我们可以设置一个阈值,低于此阈值的测试数据即为异常。
IForest具有线性时间复杂度。
IForest不适用于特别高维的数据。
最小协方差行列式(Minimum Covariance Determinant)
https://max.book118.com/html/2017/1217/144650709.shtm
论文《Minimum covariance determinant and extensions》中有更详细描述。
论文《A Fast Algorithm for the Minimum Covariance Determinant Estimator》有更详细描述。
孤立森林(IForest)代码实现及与PyOD对比
孤立森林(Isolation Forest)是经典的异常检测算法(论文网址)。本文用python对其进行实现,以及与常用的异常检测包PyOD进行效果对比。
简单来说,孤立森林(IForest)中包含若干孤立树(ITree),每颗树的创建是独立的,与其它树无关。假设数据集包含$n$个样本,每个样本都包含$m$个实数特征。在创建每颗孤立树时,根节点首先包含所有$n$个样本。对于每个节点,随机抽取一个特征,在该特征的最大与最小值之间随机取一数$p$,将小于$p$的样本划分在左子节点,将大于$p$的样本划分在右子节点。划分直到叶节点只包含一个样本,或达到树高为止,文中树高定义为$\\textceil(\\log_2n)$。构建好IForest后的测试阶段,就是计算样本在每颗孤立树上被划分到叶节点的平均路径长度,作为计算异常分数的依据。显然,划分路径越短,异常的可能性越高。
实现代码如下:
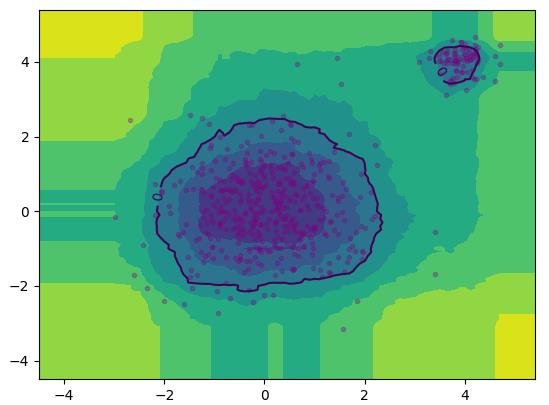
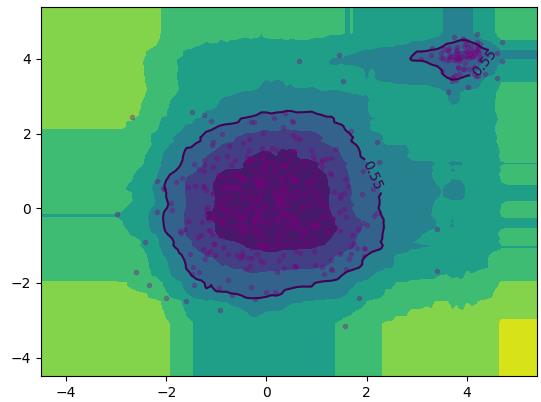
#%% 函数定义 import torch import numpy as np import matplotlib.pyplot as plt def iTree(X:torch.Tensor, e, l): # X数据集,e当前路径长,l树高最大值 if e >= l or len(X) <= 1: return [0, len(X)] # 0 非叶子节点 q = np.random.randint(0, len(X[0])) M, m = X[:, q].max(), X[:, q].min() p = np.random.rand()*(M - m) + m lchild = iTree(X[X[:,q] < p,:], e+1, l) rchild = iTree(X[X[:,q] >= p,:], e+1, l) return [1, lchild, rchild, q, p] def c(n): c = 0 if n == 1 else 2*(np.log(n-1)+0.5772156649) - (2*(n-1)/n) return c def PathLength(x, T, e): # x样本,T树,e当前路径长 if T[0] == 0: return e + c(T[1]) if x[T[3]] < T[4]: return PathLength(x, T[1], e+1) return PathLength(x, T[2], e+1) def myIForest(X, t, psi): # X训练集,t树数量,psi子采样 Ts = [] l = np.ceil(np.log(psi)) for i in range(t): x_i = np.random.choice(range(len(X)), [psi], replace=False) Ts .append(iTree(X[x_i], 0, l)) return Ts def anomalyScore(x, Ts, psi): length = 0 for T in Ts: length += PathLength(x, T, 0) length /= len(Ts) s = 2**(-length/c(psi)) return s #%% 定义正常分布、超参数、绘图矩阵 torch.manual_seed(0) np.random.seed(0) points = torch.randn([512, 2]) points[-80:] = torch.randn([80, 2])/3+4 t, psi = 100, 256 x, y = np.arange(-4.5, 5.5, 0.1), np.arange(-4.5, 5.5, 0.1) X, Y = np.meshgrid(x, y) XY = np.stack([X,Y], -1) Z = np.zeros_like(X) #%% 自定义孤立森林、异常值可视化、决策边界 myTs = myIForest(points, t, psi) for i in range(XY.shape[0]): for j in range(XY.shape[1]): Z[i,j] = anomalyScore(XY[i, j], myTs, psi) plt.plot(points[:,0],points[:,1], \'.\', c = "purple", alpha = 0.3) plt.contourf(X,Y,Z) cont = plt.contour(X,Y,Z, levels=[0.55]) plt.clabel(cont, inline=True, fontsize=10) plt.show() #%% pyOD孤立森林、异常值可视化、决策边界 from pyod.models.iforest import IForest ifor = IForest(t, psi, 0.1, random_state=0) ifor.fit(points) h, w = XY.shape[0], XY.shape[1] XY = XY.reshape(-1, 2) Z = Z.reshape(-1) Z = ifor.decision_function(XY) Z = Z.reshape(h, w) XY = XY.reshape(h,w,2) plt.plot(points[:,0],points[:,1], \'.\', c = "purple", alpha = 0.3) plt.contourf(X,Y,Z) cont = plt.contour(X,Y,Z, levels=[0]) #决策边界为0 plt.clabel(cont, inline=True, fontsize=10) plt.show()
自定义孤立森林和PyOD定义的孤立森林可视化结果分别如下左右图所示:
 |
|
效果相似。其中自定义代码完全按照论文伪代码实现,使用二叉搜索树的平均失败搜索长度进行归一化,异常分数取值$(0,1)$。PyOD的异常分数取值似乎是$(-1,1)$,以0为区分阈值,即把自定义比例的正常样本的异常分数设置为小于0,大于0则为异常样本。此处设置10%为异常,90%正常。另外,由于自定义代码没有使用并行策略,运行时间会比PyOD长得多。
以上是关于PyOD主要算法(KNN、IForest 和 MCD)的原理及使用的主要内容,如果未能解决你的问题,请参考以下文章