如何消除Go的编译特征.md
Posted Hacking就是好玩
tags:
篇首语:本文由小常识网(cha138.com)小编为大家整理,主要介绍了如何消除Go的编译特征.md相关的知识,希望对你有一定的参考价值。
Go默认编译会自带一堆信息,通过这些信息基本可以还原Go的源码架构,
本文就是研究如何消除或者混淆这些信息,记录了这个研究过程,如果不想看可以直接跳到文章末尾,文章末尾提供了一款工具,可以一键消除Go二进制中的这些敏感信息。
但还是推荐看看研究过程,可以明白这个工具的运行原理。
从逆向Go开始先写一个简单的程序
我的go版本是
version gowindows/amdmain
(
main
fmt.Println(i:=build main.build -ldflags main.和 模块,还有独特的 机制,都需要用到这些信息。来自 这篇文章就能知道,通过解析Go二进制中这些内置的数据结构,就可以还原出符号信息。
有安全研究员发现除了可以从 结构中解析、恢复函数符号,Go 二进制文件中还有大量的类型、方法定义的信息,也可以解析出来。这样就可以大大方便对 Go 二进制文件中复杂数据结构的逆向分析。
基于这种方式,已经有人写好了ida的脚本来恢复
-pkg -std -filepath -interface buildInfoMagic = []buildInfoMagic = []findVers
err !=
; !bytes.HasPrefix(data, buildInfoMagic); data = data[
bo binary.ByteOrder
bigEndian
bo = binary.BigEndian
bo = binary.LittleEndian
readPtr uint64
ptrSize ==
readPtr = uint64
readPtr = bo.Uint64
vers = readString(x, ptrSize, readPtr, readPtr(data[vers ==
&& mod[
:
mod = readStringuint64, addr uint64) string
hdr, err := x.ReadData(addr, err != || err != || version gowindows/amdmain
(
main
fmt.Println(build . 编译
编译后运行会输出GOROOT路径
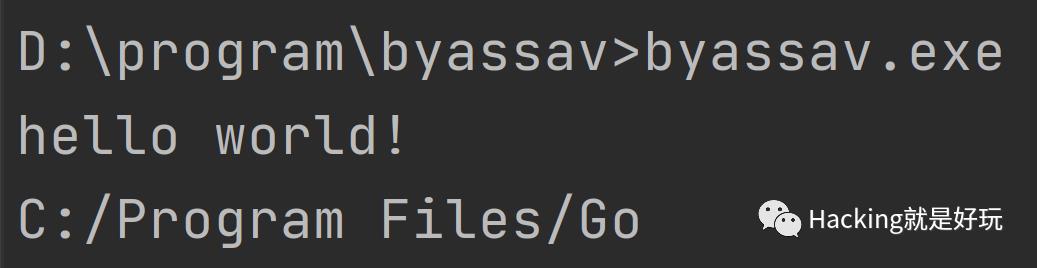
用IDA搜索这个字符串 C:/Program Files/Go,但是并没有搜到。于是转到Main函数,看到了符号信息。
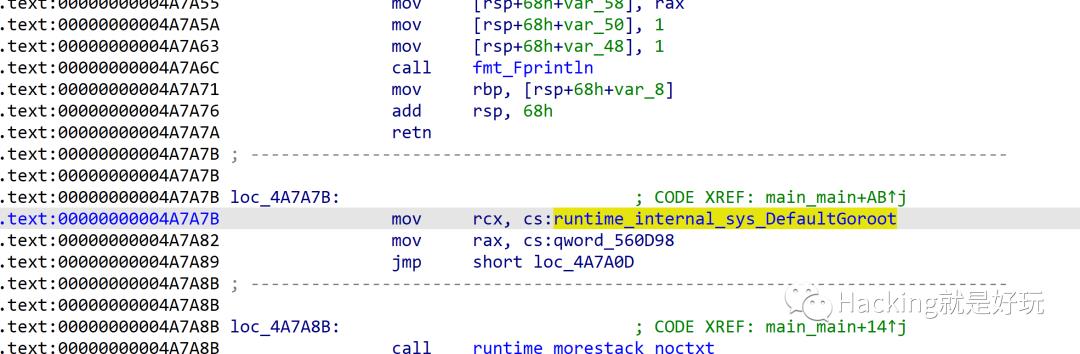
原来它是C:\\\\Program Files\\\\Go字符串,输出的时候将它改变了。

交叉引用查看
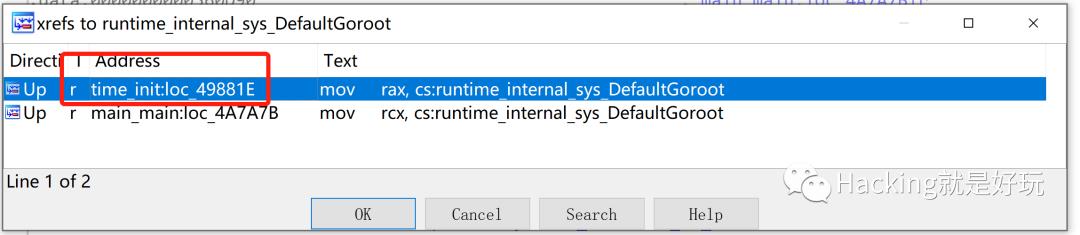
有两个地方,一个是main函数我们调用的地方,一个是time_init,这个是内部函数的实现。
我们就可以通过这个函数来定位到它。

刚刚新学了反汇编寻找地址方式,现在正好派上了用场,程序先解析pclntab获取函数time_init的地址范围,从这个地址开始反汇编,寻找mov rax,立即数指令。
因为这个赋值的汇编指令是mov,写代码的时候还要注意32位和64位寻址的不同。
tryFromTimeInit
f.FileInfo.Arch != Arch386 && f.FileInfo.Arch != ArchAMD64
f.FileInfo.Arch == Arch386
is32 = fcn *Function
std, err := f.GetSTDLib()
err !=
_, v := std
v.Name !=
_, vv := v.Functions
vv.Name !=
pkgLoop
fcn ==
err !=
s < err !=
inst.Op != x86asm.MOV
inst.Args[kindof.String() !=
arg.Base == x86asm.EIP || arg.Base == x86asm.RIP
addr = addr + arg.Base == && arg.Disp >
b ==
err !=
err !=
ver := !IsASCII(ver)
ver, main
(
main
fmt.Println(i:=build -ldflags main.go
使用程序消除信息
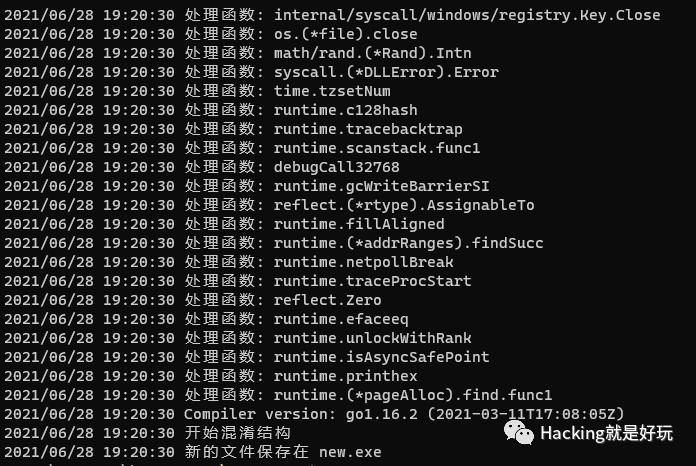
运行新的程序

运行没有问题,之前含有的文件信息都用随机字符串填充了。
用之前的IDA脚本查看
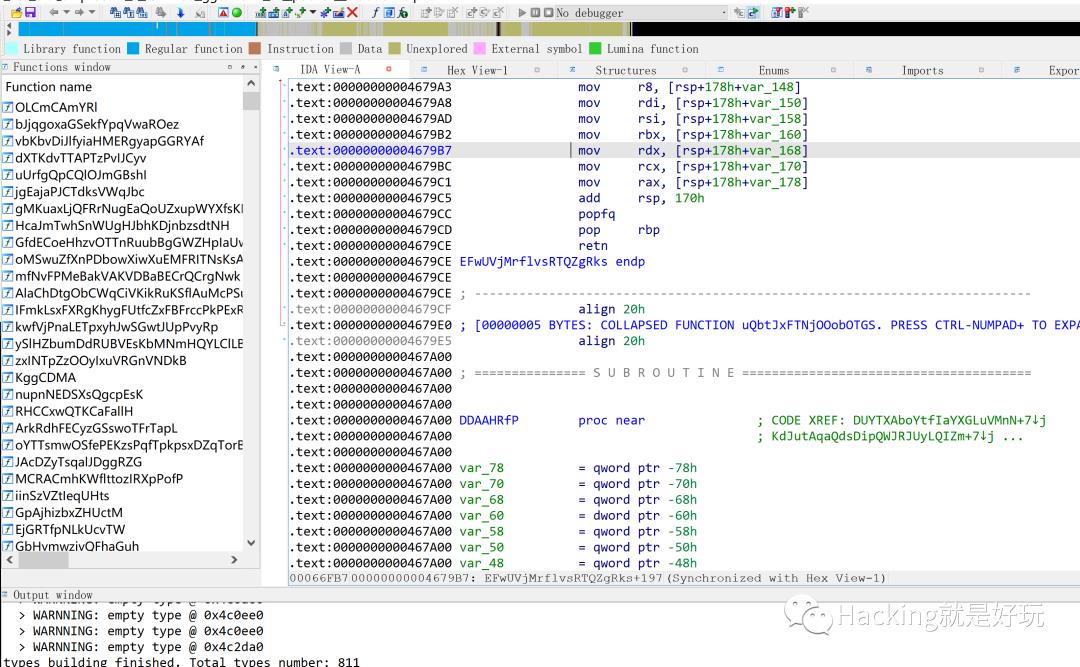
函数名称也都填充了。
与其他工具的对比知名的Go混淆工具有gobfuscate、garble
像gobfuscate,核心思想是将源码以及源码引入的包转移到一个随机目录,然后基于AST语法树修改代码信息,但这样效率有很大问题。之前测试过deimos-C2和sliver的生成混淆,生成一个简单的源码需要半个多小时甚至更长时间,并且混淆的不彻底,像Go的一些内置包、文件名都没有混淆。
像garble采取的混淆中间语言的方法,但是也有混淆不彻底和效率的问题。
相比之下go-strip混淆更彻底,效率快,支持多个平台架构,能比较方便的消除Go编译的信息。
在微信公众号回复 go-strip 即可获得下载地址
Go语言逆向初探
https://bbs.pediy.com/thread-268042.htm
Go二进制文件逆向分析从基础到进阶——综述 - 安全客,安全资讯平台
https://www.anquanke.com/post/id/214940
https://github.com/goretk/gore
https://github.com/goretk/redress
如何在 R 中使用 SVM 进行递归特征消除
【中文标题】如何在 R 中使用 SVM 进行递归特征消除【英文标题】:How to do recursive feature elimination with SVM in R 【发布时间】:2019-05-29 07:48:06 【问题描述】:我有一个看起来像这样的数据集
ID 885038 885039 885040 885041 885042 885043 885044 Class
1267359 2 0 0 0 0 1 0 0
1295720 0 0 0 0 0 1 0 0
1295721 0 0 0 0 0 1 0 0
1295723 0 0 0 0 0 1 0 0
1295724 0 0 0 1 0 1 0 0
1295725 0 0 0 1 0 1 0 0
1295726 2 0 0 0 0 1 0 1
1295727 2 0 0 0 0 1 0 1
1295740 0 0 0 0 0 1 0 1
1295742 0 0 0 0 0 1 0 1
1295744 0 0 0 0 0 1 0 1
1295745 0 0 0 0 0 1 0 1
1295746 0 0 0 0 0 1 0 1
为了进行递归特征消除,我遵循了这些步骤
-
训练 SVM 分类器
计算所有特征的排名标准
移除排名值最小的特征
转到 1。
以下是我为执行相同操作而编写的 R 代码,但是,它没有显示任何错误,并且循环继续训练集的长度。
data <- read.csv("dummy - Copy.csv", header = TRUE)
rownames(data) <- data[,1]
data<-data[,-1]
for (k in 1:length(data))
inTraining <- createDataPartition(data$Class, p = .70, list = FALSE)
training <- data[ inTraining,]
testing <- data[-inTraining,]
## Building the model ####
svm.model <- svm(Class ~ ., data = training, cross=10,metric="ROC",type="eps-regression",kernel="linear",na.action=na.omit,probability = TRUE)
###### auc measure #######
#prediction and ROC
svm.model$index
svm.pred <- predict(svm.model, testing, probability = TRUE)
#calculating auc
c <- as.numeric(svm.pred)
c = c - 1
pred <- prediction(c, testing$Class)
perf <- performance(pred,"tpr","fpr")
plot(perf,fpr.stop=0.1)
auc <- performance(pred, measure = "auc")
auc <- auc@y.values[[1]]
#compute the weight vector
w = t(svm.model$coefs)%*%svm.model$SV
#compute ranking criteria
weight_matrix = w * w
#rank the features
w_transpose <- t(weight_matrix)
w2 <- as.matrix(w_transpose[order(w_transpose[,1], decreasing = FALSE),])
a <- as.matrix(w2[which(w2 == min(w2)),]) #to get the rows with minimum values
row.names(a) -> remove
data<- data[,setdiff(colnames(data),remove)]
print(length(data))
length <- (length(data))
cols_names <- colnames(data)
print(auc)
output <- paste(length,auc,sep=";")
write(output, file = "output.txt",append = TRUE)
write(cols_names, file = paste(length,"cols_selected", ".txt", sep=""))
打印出来的样子
[1] 3
[1] 0.5
[1] 2
[1] 0.5
[1] 2
[1] 0.5
[1] 2
[1] 0.75
[1] 2
[1] 1
[1] 2
[1] 0.75
[1] 2
[1] 0.5
[1] 2
[1] 0.75
但是当我选择任何特征子集时,例如功能 3 并使用上面的代码(没有循环)构建 SVM 模型,我没有得到相同的 AUC 值 0.75。
data <- read.csv("3.csv", header = TRUE)
rownames(data) <- data[,1]
data<-data[,-1]
inTraining <- createDataPartition(data$Class, p = .70, list = FALSE)
training <- data[ inTraining,]
testing <- data[-inTraining,]
## Building the model ####
svm.model <- svm(Class ~ ., data = training, cross=10,metric="ROC",type="eps-regression",kernel="linear",na.action=na.omit,probability = TRUE)
###### auc measure #######
#prediction and ROC
svm.model$index
svm.pred <- predict(svm.model, testing, probability = TRUE)
#calculating auc
c <- as.numeric(svm.pred)
c = c - 1
pred <- prediction(c, testing$Class)
perf <- performance(pred,"tpr","fpr")
plot(perf,fpr.stop=0.1)
auc <- performance(pred, measure = "auc")
auc <- auc@y.values[[1]]
print(auc)
prints output
[1] 3
[1] 0.75 (instead of 0.5)
两个代码相同(一个带有递归循环,另一个没有任何递归循环)仍然存在相同特征子集的 AUC 值不同。
这两个代码的 3 个特征(885041、885043 和 Class)是相同的,但它给出了不同的 AUC 值。
【问题讨论】:
我不明白为什么这个问题被否决了。我还更新了我从头到尾尝试过的内容。我也做了一个快速搜索以确保它不重复......这个问题与编程有关......这就是为什么在 *** 中问...... 如果您能提供数据集的可重现示例,我相信这将是一篇好文章。 我喜欢你研究这个问题,因为很多很多问题都没有事先打扰。 @www 指的是您的图像 - 要重现您的错误,我们需要相同的数据(我们不必自己重新输入)来运行您的代码。此外,如果什么都没发生,通常提供比“它不起作用”更好 - 许多人实际上收到错误但只是说“它不起作用”。您对 www 的回答比再次告诉我它不起作用要好。 如果 SO 提供了一个自动图像阅读器,它可以获取表格位图图像并转换为测试,那么不会有问题,但是你暗示我们需要按顺序重新输入数据测试对您的编码的任何修改或改进。我们中的大多数人都没有足够的动力来重做您的数据输入。几天前有人(你?)发布类似标题的问题时,Rhelp 邮件列表的读者面临同样的问题。引用 csv 文件将无法重现,发布图像文件略有改进,但还不够好。 感谢您的建议,我已将其纳入帖子中。我希望现在代码是可重现的。 【参考方案1】:我认为只使用 交叉验证 就可以了。在您的代码中,您已经使用 10 倍 CV 来测试错误。拆分数据集似乎没有必要。
由于您没有提及调整参数,cost 或 gamma 将被设置为默认值。
library(tidyverse)
library(e1071)
library(caret)
library(ROCR)
library(foreach)
特征名称是数字,似乎svm()在拟合过程后更改了其中的名称。之后要匹配,我会先更改列名。
其次,折叠可以用caret::creadeFolds()代替createDataPartition()。
set.seed(1)
k <- 5 # 5-fold CV
mydf3 <-
mydf %>%
rename_at(.vars = vars(-ID, -Class), .funs = function(x) str_c("X.", x, ".")) %>%
mutate(fold = createFolds(1:n(), k = k, list = FALSE)) # fold id column
# the number of features-------------------------------
x_num <-
mydf3 %>%
select(-ID, -Class, -fold) %>%
ncol()
要进行迭代,foreach() 可以是另一种选择。
cl <- parallel::makeCluster(2)
doParallel::registerDoParallel(cl, cores = 2)
parallel::clusterExport(cl, c("mydf3", "x_num"))
parallel::clusterEvalQ(cl, c(library(tidyverse), library(ROCR)))
#---------------------------------------------------------------
svm_rank <-
foreach(j = seq_len(x_num), .combine = rbind) %do%
mod <-
foreach(cv = 1:k, .combine = bind_rows, .inorder = FALSE) %dopar% # parallization
tr <-
mydf3 %>%
filter(fold != cv) %>% # train
select(-fold, -ID) %>%
e1071::svm( # fitting svm
Class ~ .,
data = .,
kernel = "linear",
type = "eps-regression",
probability = TRUE,
na.action = na.omit
)
# auc
te <-
mydf3 %>%
filter(fold == cv) %>%
predict(tr, newdata = ., probability = TRUE)
predob <- prediction(te, mydf3 %>% filter(fold == cv) %>% select(Class))
auc <- performance(predob, measure = "auc")@y.values[[1]]
# ranking - your formula
w <- t(tr$coefs) %*% tr$SV
if (is.null(names(w))) colnames(w) <- attr(tr$terms, "term.labels") # when only one feature left
(w * w) %>%
tbl_df() %>%
mutate(auc = auc)
auc <- mean(mod %>% select(auc) %>% pull()) # aggregate cv auc
w_mat <- colMeans(mod %>% select(-auc)) # aggregate cv ranking
remove <- names(which.min(w_mat)) # minimum rank
used <-
mydf3 %>%
select(-ID, -Class, -fold) %>%
names() %>%
str_c(collapse = " & ")
mydf3 <-
mydf3 %>%
select(-remove) # remove feature for next step
tibble(used = used, delete = remove, auc = auc)
#---------------------------------------------------
parallel::stopCluster(cl)
对于每一步,你可以得到
svm_rank
#> # A tibble: 7 x 3
#> used delete auc
#> <chr> <chr> <dbl>
#> 1 X.885038. & X.885039. & X.885040. & X.885041. & X.885042… X.88503… 0.7
#> 2 X.885038. & X.885040. & X.885041. & X.885042. & X.885043… X.88504… 0.7
#> 3 X.885038. & X.885041. & X.885042. & X.885043. & X.885044. X.88504… 0.7
#> 4 X.885038. & X.885041. & X.885043. & X.885044. X.88504… 0.7
#> 5 X.885038. & X.885041. & X.885043. X.88504… 0.7
#> 6 X.885038. & X.885041. X.88503… 0.7
#> 7 X.885041. X.88504… 0.7
【讨论】:
以上是关于如何消除Go的编译特征.md的主要内容,如果未能解决你的问题,请参考以下文章