手把手教你用python实现机器学习预测疾病
Posted 程序员石磊
tags:
篇首语:本文由小常识网(cha138.com)小编为大家整理,主要介绍了手把手教你用python实现机器学习预测疾病相关的知识,希望对你有一定的参考价值。
电子存储的医疗成像数据非常丰富,机器学习算法可以使用这种类型的数据集来检测和发现模式和异常。在本文中,我将向您介绍五个医疗保健领域的机器学习项目。
机器和算法可以解读成像数据,就像受过高度训练的放射科医生可以识别皮肤上的可疑斑点、病变、肿瘤和脑部出血一样。因此,机器学习工具和平台的使用,以帮助放射科医生准备增长指数。
机器学习被用于世界各地的许多领域。医疗保健行业也不例外。机器学习可以在预测运动障碍、心脏病、癌症、肺部疾病等方面发挥关键作用。
这些信息,如果提前很好地预测,可以为医生提供重要的信息,然后他们可以为每个患者量身定制诊断和治疗。现在让我们来看看一些医疗领域的机器学习项目。
心脏病预测:

心脏病 描述了影响您心脏的一系列疾病。心脏疾病的疾病包括血管疾病,例如冠状动脉疾病,心律问题(心律不齐)和您天生的心脏缺陷(先天性心脏缺陷)等。
心脏病是世界人口发病率和死亡率的最大原因之一。在临床数据科学部分,心血管疾病的预测被认为是最重要的主题之一。医疗保健行业中的数据量巨大。
在这个数据科学项目中,我将应用机器学习技术对一个人是否患有心脏病进行分类。
您可以从此处下载该项目所需的数据集:
链接:https://pan.baidu.com/s/1aiXSnklK_q0BoaWhLLM2mQ
提取码:6rfj
导入所需的模块:
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
from matplotlib import rcParams
import seaborn as sns
import warnings
warnings.filterwarnings('ignore')
在这里,我们将使用KNeighborsClassifier进行实验:
from sklearn.neighbors import KNeighborsClassifier
现在让我们深入研究数据
df = pd.read_csv('dataset.csv')
print(df.head())
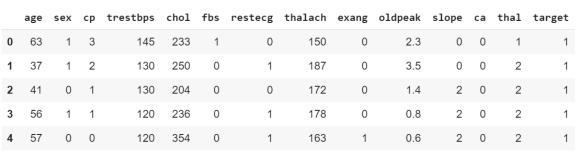
print(df.info())
输出
<class 'pandas.core.frame.DataFrame'>
RangeIndex: 303 entries, 0 to 302
Data columns (total 14 columns):
age 303 non-null int64
sex 303 non-null int64
cp 303 non-null int64
trestbps 303 non-null int64
chol 303 non-null int64
fbs 303 non-null int64
restecg 303 non-null int64
thalach 303 non-null int64
exang 303 non-null int64
oldpeak 303 non-null float64
slope 303 non-null int64
ca 303 non-null int64
thal 303 non-null int64
target 303 non-null int64
dtypes: float64(1), int64(13)
memory usage: 33.2 KB
print(df.describe())
[外链图片转存失败,源站可能有防盗]!链机制,建(https://img-Wpblog.czdnimg.cn/imgonvert/a0ad52ecc264ca0e6473a29c790ee85e.png)]
特征选择
获取数据集中每个特征的相关性
import seaborn as sns
corrmat = df.corr()
top_corr_features = corrmat.index
plt.figure(figsize=(16,16))
#plot heat map
g=sns.heatmap(df[top_corr_features].corr(),annot=True,cmap="RdYlGn")
plt.show()

在目标类的大小大约相等的情况下,使用数据集始终是一个好习惯。因此,让我们检查一下是否相同:
sns.set_style('whitegrid')
sns.countplot(x='target',data=df,palette='RdBu_r')
plt.show()

数据处理
探索数据集后,我发现我需要在训练机器学习模型之前将一些分类变量转换为虚拟变量并缩放所有值。
首先,我将使用该 get_dummies 方法为分类变量创建虚拟列。
dataset = pd.get_dummies(df, columns = ['sex', 'cp',
'fbs','restecg',
'exang', 'slope',
'ca', 'thal'])
from sklearn.model_selection import train_test_split
from sklearn.preprocessing import StandardScaler
standardScaler = StandardScaler()
columns_to_scale = ['age', 'trestbps', 'chol', 'thalach', 'oldpeak']
dataset[columns_to_scale] = standardScaler.fit_transform(dataset[columns_to_scale])
dataset.head()

y = dataset['target']
X = dataset.drop(['target'], axis = 1)
from sklearn.model_selection import cross_val_score
knn_scores = []
for k in range(1,21):
knn_classifier = KNeighborsClassifier(n_neighbors = k)
score=cross_val_score(knn_classifier,X,y,cv=10)
knn_scores.append(score.mean())
plt.plot([k for k in range(1, 21)], knn_scores, color = 'red')
for i in range(1,21):
plt.text(i, knn_scores[i-1], (i, knn_scores[i-1]))
plt.xticks([i for i in range(1, 21)])
plt.xlabel('Number of Neighbors (K)')
plt.ylabel('Scores')
plt.title('K Neighbors Classifier scores for different K values')
plt.show()

knn_classifier = KNeighborsClassifier(n_neighbors = 12)
score=cross_val_score(knn_classifier,X,y,cv=10)
score.mean()
# 输出- 0.8448387096774195
随机森林分类器
from sklearn.ensemble import RandomForestClassifier
randomforest_classifier= RandomForestClassifier(n_estimators=10)
score=cross_val_score(randomforest_classifier,X,y,cv=10)
score.mean()
# 输出 - 0.8113978494623655
皮肤癌的分类:
皮肤癌是美国最常见的疾病之一。据报道,今年美国有多达400万人死于皮肤癌。在这里,您将学习如何使用机器学习创建皮肤癌分类模型。
这是一个巨大的数字,在一个国家就有400万人死于皮肤癌。就像所有这些死亡的人一样,但有一半甚至更多的病例,在疾病的早期没有去看医生而疾病本来是可以预防的。

皮肤癌是美国最常见的疾病之一。在过去的一年中,据报道有多达400万例死于皮肤癌的病例。在本文中,我将使用机器学习创建皮肤癌分类模型。
一个国家真的有400万人死于皮肤癌,这是一个巨大的数字。由于所有这些人都在死亡,但是其中一半甚至更多的病例在疾病可以预防的早期阶段就没有去看医生。
如果人们患上了皮肤癌的症状,他们仍然不愿意去看医生,这不是一个好信号,因为皮肤癌可以在早期阶段治愈。
机器学习对皮肤癌的分类
因此,这是机器学习算法在皮肤癌分类中起作用的地方。正如我之前提到的,皮肤癌可以在疾病的早期阶段很容易治愈,但这是人们不想去看医生的人。
因此,这是一个简单的机器学习算法,可以帮助那些人坐在家里时确定他们是否患有皮肤癌。该机器学习算法基于卷积神经网络(CNN)。
CNN层分类用于皮肤癌检测
让我们从导入库开始
import numpy as np
from skimage import io
import matplotlib.pyplot as plt
现在,我将简单地上传图像,以使用python中的skimage库来训练我们的机器学习模型。
imgb = io.imread('bimg-1049.png')
imgm = io.imread('mimg-178.png')
imgb = io.imread('bimg-721.png')
imgm = io.imread('mimg-57.png')
您可以从下面下载这些图像
链接:https://pan.baidu.com/s/17UB13G3NckDH4mXW7Bf25w
提取码:nh9p
这些图像是良性痣和恶性痣的样本图像,这是一种皮肤问题。
让我们展示这些图像
plt.figure(figsize=(10,20))
plt.subplot(121)
plt.imshow(imgb)
plt.axis('off')
plt.subplot(122)
plt.imshow(imgm)
plt.axis('off')

现在让我们训练模型以进行进一步分类
from keras.models import load_model
model = load_model('BM_VA_VGG_FULL_DA.hdf5')
from keras import backend as K
def activ_viewer(model, layer_name, im_put):
layer_dict = dict([(layer.name, layer) for layer in model.layers])
layer = layer_dict[layer_name]
activ1 = K.function([model.layers[0].input, K.learning_phase()], [layer.output,])
activations = activ1((im_put, False))
return activations
def normalize(x):
# utility function to normalize a tensor by its L2 norm
return x / (K.sqrt(K.mean(K.square(x))) + 1e-5)
def deprocess_image(x):
# normalize tensor: center on 0., ensure std is 0.1
x -= x.mean()
x /= (x.std() + 1e-5)
x *= 0.1
# clip to [0, 1]
x += 0.5
x = np.clip(x, 0, 1)
# convert to RGB array
x *= 255
if K.image_data_format() == 'channels_first':
x = x.transpose((1, 2, 0))
x = np.clip(x, 0, 255).astype('uint8')
return x
def plot_filters(filters):
newimage = np.zeros((16*filters.shape[0],8*filters.shape[1]))
for i in range(filters.shape[2]):
y = i%8
x = i//8
newimage[x*filters.shape[0]:x*filters.shape[0]+filters.shape[0],
y*filters.shape[1]:y*filters.shape[1]+filters.shape[1]] = filters[:,:,i]
plt.figure(figsize = (10,20))
plt.imshow(newimage)
plt.axis('off')
皮肤癌分类模型总结
要查看我们训练有素的模型的总结,我们将执行以下代码
model.summary()
model.summary()
_________________________________________________________________
Layer (type) Output Shape Param #
=================================================================
input_1 (InputLayer) (None, 128, 128, 3) 0
_________________________________________________________________
block1_conv1 (Conv2D) (None, 128, 128, 64) 1792
_________________________________________________________________
block1_conv2 (Conv2D) (None, 128, 128, 64) 36928
_________________________________________________________________
block1_pool (MaxPooling2D) (None, 64, 64, 64) 0
_________________________________________________________________
block2_conv1 (Conv2D) (None, 64, 64, 128) 73856
_________________________________________________________________
block2_conv2 (Conv2D) (None, 64, 64, 128) 147584
_________________________________________________________________
block2_pool (MaxPooling2D) (None, 32, 32, 128) 0
_________________________________________________________________
block3_conv1 (Conv2D) (None, 32, 32, 256) 295168
_________________________________________________________________
block3_conv2 (Conv2D) (None, 32, 32, 256) 590080
_________________________________________________________________
block3_conv3 (Conv2D) (None, 32, 32, 256) 590080
_________________________________________________________________
block3_pool (MaxPooling2D) (None, 16, 16, 256) 0
_________________________________________________________________
block4_conv1 (Conv2D) (None, 16, 16, 512) 1180160
_________________________________________________________________
block4_conv2 (Conv2D) (None, 16, 16, 512) 2359808
_________________________________________________________________
block4_conv3 (Conv2D) (None, 16, 16, 512) 2359808
_________________________________________________________________
block4_pool (MaxPooling2D) (None, 8, 8, 512) 0
_________________________________________________________________
block5_conv1 (Conv2D) (None, 8, 8, 512) 2359808
_________________________________________________________________
block5_conv2 (Conv2D) (None, 8, 8, 512) 2359808
_________________________________________________________________
block5_conv3 (Conv2D) (None, 8, 8, 512) 2359808
_________________________________________________________________
block5_pool (MaxPooling2D) (None, 4, 4, 512) 0
_________________________________________________________________
sequential_3 (Sequential) (None, 1) 2097665
=================================================================
Total params: 16,812,353
Trainable params: 16,812,353
Non-trainable params: 0
现在让我们可视化我们训练过的模型的输出
activ_benign = activ_viewer(model,'block2_conv1',imgb.reshape(1,128,128,3))
img_benign = deprocess_image(activ_benign[0])
plot_filters(img_benign[0])
plt.figure(figsize=(20,20))
for f in range(128):
plt.subplot(8,16,f+1)
plt.imshow(img_benign[0,:,:,f])
plt.axis('off')

现在让我们缩放和可视化一些过滤器
plt.figure(figsize=(10,10))
plt.subplot(121)
plt.imshow(img_benign[0,:,:,49])
plt.axis('off')
plt.subplot(122)
plt.imshow(img_malign[0,:,:,49])
plt.axis('off')

plt.figure(figsize=(10,10))
plt.subplot(121)
plt.imshow(img_benign[0,:,:,94])
plt.axis('off')
plt.subplot(122)
plt.imshow(img_malign[0,:,:,94])
plt.axis('off')

测试模型
现在让我们可视化并测试我们训练后的模型的实际能力
def plot_filters32(filters):
newimage = np.zeros((16*filters.shape[0],16*filters.shape[1]))
for i in range(filters.shape[2]):
y = i%16
x = i//16
newimage[x*filters.shape[0]:x*filters.shape[0]+filters.shape[0],
y*filters.shape[1]:y*filters.shape[1]+filters.shape[1]] = filters[:,:,i]
plt.figure(figsize = (15,25))
plt.imshow(newimage)
activ_benign = activ_viewer(model,'block3_conv3',imgb.reshape(1,128,128,3))
img_benign = deprocess_image(activ_benign[0])
plot_filters32(img_benign[0])

activ_malign = activ_viewer(model,'block3_conv3',imgm.reshape(1,128,128,3))
img_malign = deprocess_image(activ_malign[0])
plot_filters32(img_malign[0])

基于机器学习的肺分割:
肺分割是机器学习在医疗保健领域最有用的任务之一。肺CT图像分割是肺图像分析的一个必要的初始步骤,它是提供准确的肺CT图像分析如肺癌检测的初始步骤。
Dicom实际上是医学影像的存储库。这些文件包含大量元数据。这种分析的像素大小/粗度不同于不同的分析,这可能会对CNN方法的性能产生不利影响。
在本文中,我将向您介绍机器学习在医疗保健中的应用。我将向您展示如何使用python进行机器学习来完成肺分割任务。
肺分割是医疗保健中机器学习最有用的任务之一。肺部CT图像分割是进行肺部图像分析所必需的第一步,这是提供准确的肺部CT图像分析(例如检测肺癌)的初步步骤。
现在,让我们看看如何将机器学习用于肺部分割任务。在开始之前,我将导入一些软件包并确定可用的患者:
import numpy as np
import pandas as pd
import dicom
import os
import scipy.ndimage
import matplotlib.pyplot as plt
from skimage import measure, morphology
from mpl_toolkits.mplot3d.art3d import Poly3DCollection
# Some constants
INPUT_FOLDER = 'path to sample_images'
patients = os.listdir(INPUT_FOLDER)
patients.sort()
加载数据以进行肺分割
Dicom是医学成像中的事实上的存储库。这是我第一次使用它,但是看起来很简单。这些文件包含很多元数据。此分析像素大小/粗度因分析而异,这可能会对CNN方法的性能产生不利影响。
下面是加载分析的代码,其中包含多个切片,我们将它们简单地保存在Python列表中。数据集中的每个记录都是一个分析。缺少元数据字段,即Z方向上像素的大小,即切片的厚度。幸运的是,我们可以推断出它并将其添加到元数据中:
def load_scan(path):
slices = [dicom.read_file(path + '/' + s)