利用朴素贝叶斯来预测是否患有糖尿病
Posted Python数据分析之旅
tags:
篇首语:本文由小常识网(cha138.com)小编为大家整理,主要介绍了利用朴素贝叶斯来预测是否患有糖尿病相关的知识,希望对你有一定的参考价值。
一.概率学派

1.传统统计学派观点:(1)观察到的数据被认为是随机的,因为它们是随机过程的实现,因此每次观察系统时都会发生变化(2)模型参数被认为是固定的。参数的值是未知的,但它们是固定的,因此我们对它们进行条件设置存在问题:对小样本事件并不能进行准确的评估,若想得到相对准确的结论往往需要大量的现场实验
2.贝叶斯学派观点:(1)数据被认为是固定的。他们使用的是随机的,但是一旦他们被拿到手了,就不会改变(2)贝叶斯用概率分布来描述模型参数的不确定性,这样一来,他们就是随机的了相对传统频率学派优点:利用已有的先验信息,可以得到分析对象准确的后验分布分析思路:1.根据已有的经验和知识推断一个先验概率2.新证据(数据集)不断积累的情况下调整这个概率
二.朴素贝叶斯简介
1.与贝叶斯关系贝叶斯分类是分类算法总称,这类算法均以贝叶斯定理为基础(也就是贝叶斯学派观点)。朴素贝叶斯是是一种贝叶斯分类算法,在许多场合可以应用。2.关键:对已知类别,假设所有属性相互独立3.数学表达式:(d为属性数目,xi为x在第i个属性上取值
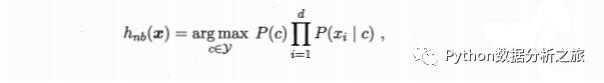
朴素贝叶斯,贝叶斯分类与贝叶斯原理三者关系如下图: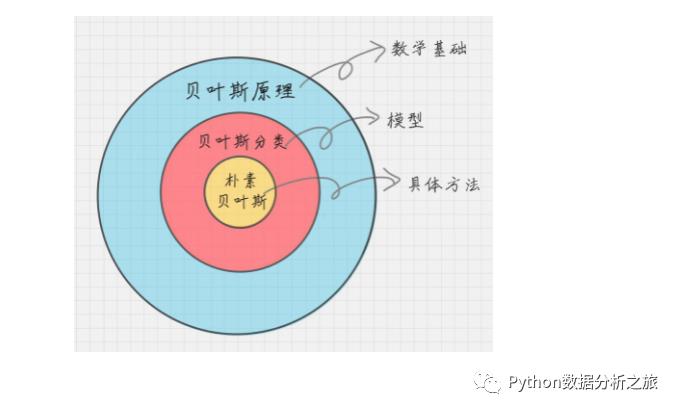
三.预测是否患有糖尿病
import numpy as npimport pandas as pdimport seaborn as sns%matplotlib inline#读取数据data=pd.read_csv('./diabetes.csv')#查看数据data.head()
(0)怀孕次数 --- Number of times pregnant(1)2小时口服葡萄糖耐量试验中的血浆葡萄糖浓度 --- Plasma glucose concentration a 2 hours in an oral glucose tolerance test(2)舒张压(毫米汞柱)--- Diastolic blood pressure (mm Hg)(3)三头肌皮褶厚度 (毫米) --- Triceps skin fold thickness (mm)(4)2小时血清胰岛素(mu U/ml) --- 2-Hour serum insulin (mu U/ml)(5)体重指数(BMI)--- Body mass index (weight in kg/(height in m)^2)(6)糖尿病血系功能 --- Diabetes pedigree function(7)年龄(年)--- Age (years)(8)类别:过去5年内是否有糖尿病 --- Class variable (0 or 1,1 yes 0 no)
#字段名称names=['pregnant','glucose','bloodPressure','skinThickness','insulin','BMI','diePedigree','age','cat']#重置列名data.columns=names#查看数据data.head()
#查看数据信息data.info()#没有发现缺失值
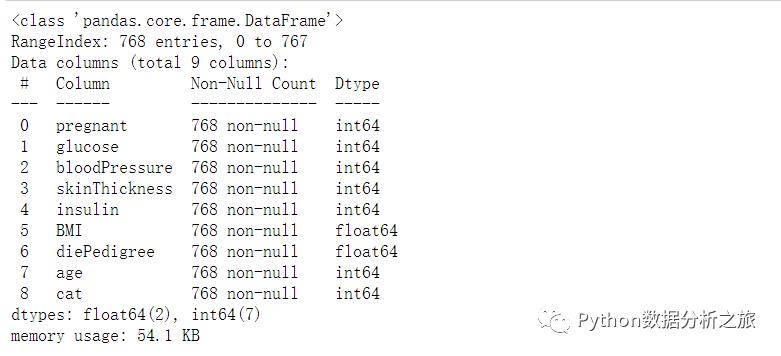
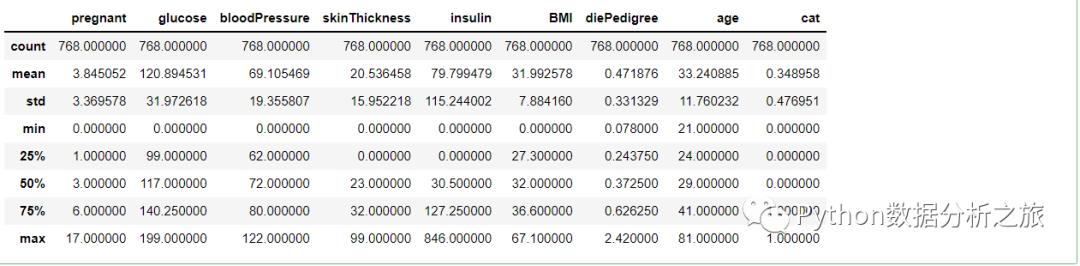
#查看数据型数据信息data.describe()
#目标值简单分布data.cat.value_counts()#我们发现数据分布不是很均匀,后期拆分数据集时要适当处理

#热力图sns.heatmap(data.corr())#发现bloodPressure,skinThickness,insulin相关性不是很强

from sklearn.model_selection import train_test_split#分离数据集,这里我们按照目标值占比进行分离数据集stratify=data.catX_train,X_test,y_train,y_test=train_test_split(data.loc[:,data.columns!='cat'],data['cat'],stratify=data.cat)
from sklearn.ensemble import RandomForestClassifier#随机森林分类进行特征选择rf=RandomForestClassifier(n_estimators=100,random_state=0)#构建模型rf.fit(X_train,y_train)#输出训练集准确率print(rf.score(X_train,y_train))#输出测试集准确率print(rf.score(X_test,y_test))#模型有点过拟合

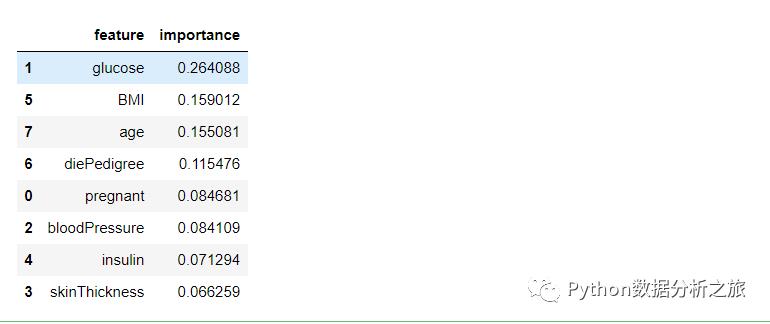
#特征重要性importances = rf.feature_importances_importances

from pyecharts.charts import Scatterfrom pyecharts.charts import Barfrom pyecharts import options as optsfrom pyecharts.charts import Pageimportances_df = pd.DataFrame({'feature' : data.columns[:-1],'importance' : importances})importances_df.sort_values(by = 'importance', ascending = False, inplace = True)#作图bar = Bar()bar.add_xaxis(importances_df.feature.tolist())bar.add_yaxis('importance',importances_df.importance.tolist(),label_opts = opts.LabelOpts(is_show = False))bar.set_global_opts(title_opts = opts.TitleOpts(title = '糖尿病数据各特征重要程度'),xaxis_opts = opts.AxisOpts(axislabel_opts = opts.LabelOpts(rotate = 30)),datazoom_opts = [opts.DataZoomOpts()])bar.render('diabetes_importances_bar.html')bar.render_notebook()

#查看特征重要性importances_df#后期我们用glucose,BMI, age,diePedigree来建模,这四个特征重要性比较高
#构建模型#这里我们用暂且用高斯朴素贝叶斯,它主要用于处理连续性变量数据,它的模型假设是每个维度都符合高斯分布from sklearn.naive_bayes import GaussianNBNBmodel=GaussianNB()#复制训练集数据X_train_=X_train.copy()#构架模型NBmodel.fit(X_train_[['glucose','BMI', 'age','diePedigree']],y_train)#准确率NBmodel.score(X_train_[['glucose','BMI', 'age','diePedigree']],y_train)

from sklearn.metrics import roc_curve,auc#预测测试集y_pred_score=NBmodel.predict(X_test[['glucose','BMI', 'age','diePedigree']])#输出结果y_pred_score
print("测试集准确率为:",NBmodel.score(X_test[['glucose','BMI', 'age','diePedigree']],y_test))#我们发现测试集准确率不是很高,其实训练集准确率不是很高早就对这个结果有预兆#主要因为是朴素贝叶斯是个很简单的算法,并且我们所选属性之间并不是相互独立#还有我们对所选特征没有进行相应处理,建议用神经网络效果会更好
以上是关于利用朴素贝叶斯来预测是否患有糖尿病的主要内容,如果未能解决你的问题,请参考以下文章