综述:应用自然语言处理进行蛋白质研究
Posted BioRain
tags:
篇首语:本文由小常识网(cha138.com)小编为大家整理,主要介绍了综述:应用自然语言处理进行蛋白质研究相关的知识,希望对你有一定的参考价值。
排版:佩琪
审核:恺忻

综述
应用自然语言处理进行蛋白质研究
写在前面:今天想跟大家分享一篇用NLP研究蛋白质的综述。小编接触这个领域时间不长,如果有写的不对的地方,希望大家多多包容,也欢迎大家批评指正~一起学习,一起进步(⁎⁍̴̛ᴗ⁍̴̛⁎)
背景
本文综述了应用自然语言处理Natural language processing (NLP)研究蛋白质的成功,未来前景以及存在的困难。探索了蛋白质序列和自然语言在概念上的异同,总结了可以用机器学习研究的蛋白质相关任务,并讨论了NLP应用于蛋白质研究的趋势和挑战。
1
自然语言与蛋白质序列的异同
自然语言(以英语为例)由字母构成,不同的字母通过固定搭配组成能够表达意义的单位:单词。自然语言还具备信息完整性(information completeness),即知道了一句话的全部字母,就可以完全了解这句话想要传达的全部信息。从这个角度上看,蛋白质与自然语言有很大的相似处:蛋白质由氨基酸序列构成,也有由特定氨基酸序列组合成的复用模块(reused module)。同样,一旦氨基酸序列被确定,蛋白质的结构和功能也就确定了,即具备信息完整性。
同时,自然语言与蛋白质序列也存在一定的差异:自然语言有明确的词库、统一标点,句子长度相差也不大;而蛋白质缺乏明确的词库,序列长度差异也很大。自然语言中特定的单词往往会有关键影响,而在蛋白质中,这种影响是累加性的。最后,自然语言基本没有远距离互动(distant interactions),但这在蛋白质中是常见的,并且由于三维结构,距离很远的氨基酸残基之间也可以产生相互作用。
2
基于序列的预测任务种类
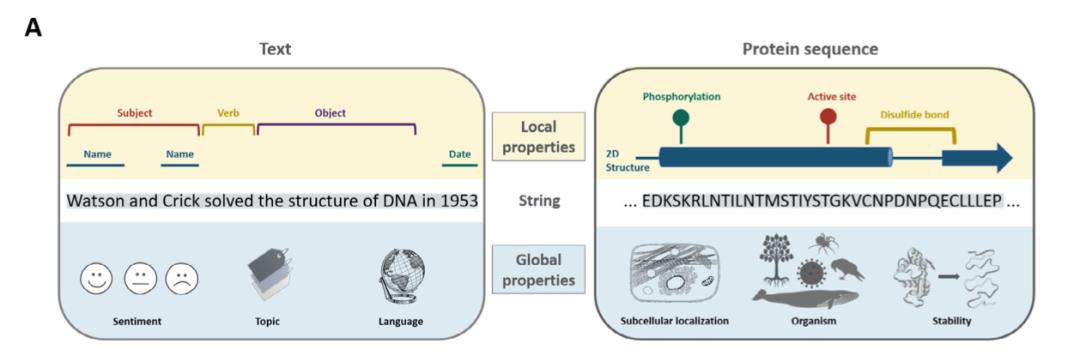
图表 1 文本和蛋白质的预测任务
NLP方法被用来研究基于序列(文本/氨基酸)的预测任务。从最基础的层面上来说,根据关注的范围,基于序列的预测任务大致可以分为局部预测和整体预测。对于自然语言,整体预测包括预测句子所表达的情绪,态度;局部预测包括判断句子中某个词的词性等。对于蛋白质,整体预测包括预测蛋白质的种类,热稳定性,功能等;局部预测则可以是预测某位点特定的残基类型,2D和3D结构,翻译后修饰情况等。
3
Tokenization和bag-of-words(BoW)
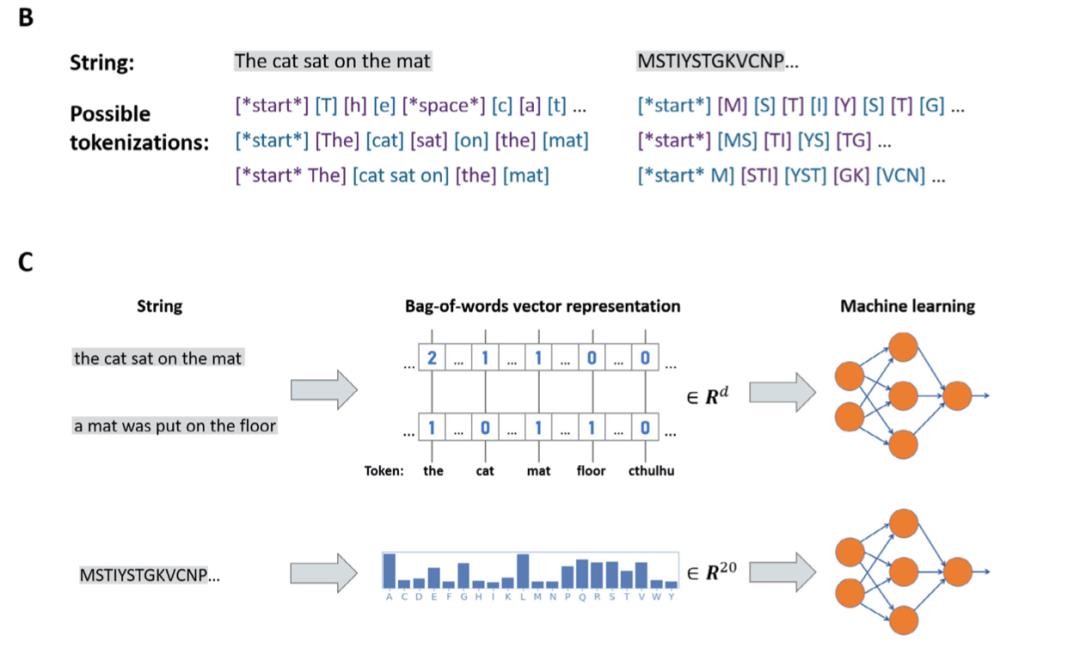
图表 2 B: 文本和蛋白质的不同tokenization;C: Bag-of-words表示,随后通过机器学习分析
基于计算的文本分析需要tokenization。对于文本(以英语为例),tokens可以是单词,单个字母或者子词(sub-word segmentation)。根据应用场景,这几种方法各有优缺点。对于蛋白质也是类似的,最简单、常用的tokens是单个氨基酸残基。因为蛋白质没有定义清晰的词汇库,“单词”tokens不好明确界定,正因如此,sub-word便成为一个很有前景的tokens,也有希望能探索发现蛋白质中“单词”或motifs。
BoW是在文本分析中最简单、最常用的特征提取方法。BoW忽略文本中tokens的顺序,可以对单个tokens或多个tokens组合出现的次数,频率以及有或无(binary features)进行统计。BoW快速,有效,简单并适用于不同长度的大文本。然而由于tokens的顺序在BoW中丢失,对于一些复杂任务,BoW可能有些过于简单。对于蛋白质来说,常见的BoW tokens包括单个氨基酸,或者也可以是2D结构以及一些特定的氨基酸残基。
4
情景化词嵌入(Contextualized embeddings)
词嵌入(Word embeddings)是一系列将tokens(如:单词)表示为固定大小向量的算法,(类别,功能)相近的词会获得相似的表示。词嵌入相比于BoW,给tokens提供了一种有效的低维表示。Word2vec就是一种常用的词嵌入算法。由于自然语言词库庞大,低维化的嵌入在NLP中非常流行。但对于仅由约20个氨基酸构成的蛋白质来说,如果tokens是氨基酸残基,那么词嵌入就没有明显的优势了。但如果tokens是氨基酸组合,词嵌入的方法就可以提供一个更为实用的表示。
相比于Word2vec和其他相似方法,情景化词嵌入考虑了语境和顺序。同样的词在不同的语境下,用Word2vec会产生相同的表示,但用情景化词嵌入则会有不同的表示。尽管后者更复杂,计算成本更高,但往往会在基准测试(benchmark)中产生SOTA(state-of-the-art)的结果。典型的情景化词嵌入是基于神经网络的,一些流行的深度学习架构包括长短期记忆(long short-term memory),sequence-to-sequence(seq2seq)和注意力(attention)模型。深度学习架构往往依赖迁移学习,如果某个问题的数据更加稀有,则模型会在另一个问题上训练然后回到这个问题进行微调(fine-tuned or transferred)。目前,已有使用情景化词嵌入模型(如:ELMO)对蛋白质进行监督学习任务的工作。
5
深层语言模型(Deep language models)
得益于深层语言模型ULMFiT, BERT, XLNet和其他BERT变体,NLP达到了“Imagenet moment”:即便在数据很少的情况下,模型、数据集和预训练任务可为大多数任务提供强大的现成性能。
图表 3 预训练和fine-tuning
语言模型在被训练去根据语境预测文本中的某个tokens时,可以产生极好的泛化能力去理解语言的结构,从而明确正确答案的类型而非给出单一的答案。用于预训练的语言模型的一个关键优势是它们是自我监督(self-supervised)的(如在掩码语言masked language task任务中):该模型预测明确的基本事实,但不需要标记数据,使其可用于任何语料库。目前,所有SOTA的神经语言模型都是基于注意力的,通常使用 Transformer 架构。这些模型具有以下特征:1. 在各种benchmark中取得SOTA;2. 在大型文本语料库进行自监督语言建模预训练;3. 拥有巨大的、深度的神经网络,并且可以从更大、更深的模型和数据集中获得持续的效果提升。
深层语言模型在蛋白质和基因组学的研究中也展现出了前景。蛋白质语言建模中成功的架构包括LSTM和注意力模型。下游的任务包括探索蛋白质的分类起源,对序列进行稳定性分析等。蛋白质模型发展的进度取决于有代表性的、包含各种蛋白质相关任务的benchmarks,例如Berkeley在2019年提出的TAPE(Tasks Assessing Protein Embeddings)。TAPE 用便捷、标准化的格式组合了各种不同的任务,并评估了不同的模型:包括BERT-like transformer,CNN(convolutional neural networks),LSTM和非深度学习方法。对所有模型来说,语言模型的预训练都能提升模型效果。
展望
深度学习并非万灵药,它存在一些缺点:训练慢、对文本或者序列的长度敏感、容易过拟合、模型很难解释等。将深层模型与专业知识结合会比一味追求更复杂的模型可能更能提升模型效果。目前为止,应用NLP模型研究蛋白质效果提升很重要的两点包括fine-tuning预训练模型以及更丰富,高质量的数据库。同时,一些竞赛如CAFA,CASP等也在激励蛋白质预测领域的研究,并为其提供了严格的测试来衡量算法的优劣。更重要的一点是对于benchmarks的研究,计算蛋白质的benchmarks研究与NLP和其他机器学习领域还有相当一段距离,建立标准化、客观的标准是衡量不同模型的关键也是未来应当努力的方向。
参考文献:
往期内容
往期精彩不容错过
2021.6.22
以上是关于综述:应用自然语言处理进行蛋白质研究的主要内容,如果未能解决你的问题,请参考以下文章