GO Term Finder:一个强大的GO功能注释分析工具
Posted 医知圈
tags:
篇首语:本文由小常识网(cha138.com)小编为大家整理,主要介绍了GO Term Finder:一个强大的GO功能注释分析工具相关的知识,希望对你有一定的参考价值。
大家好,我是小伍,今天给大家分享下GO分析,这其实是一个很基本的分析,我比较了下其它的一些工具,感觉这个也是相当不错,现分享给大家。
The GO Term Finder (Version 0.83) searches for significant shared GO terms, or parents of those GO terms, used to describe the genes in your list to help you discover what the genes may have in common. To map annotations of a group of genes to more general terms and/or to bin them in broad categories, use the GO Slim Mapper.
Gene Ontology(GO)项目是一个通过共同的语义term来描述基因生物学功能的项目。这里的分析主要包括三个层面:分子功能、生物过程和细胞组分。
为了尽可能详细的提供功能信息,基因产物被尽可能的富集到最低层的功能term上。比如说,如果一个基因产物被定为在perinuclear space(核周区间),那么他只会定位到这个特定的term,而不会定位到它的父亲节点或其他上级term中。在此例中,perinuclear space就是nucleus的子节点,然而对于一些其他的目的,比如为了分析微阵列数据的结果,那么单独对于某一个基因进行注释已经不能满足我们的分析要求了。而是需要更为复杂的统计学计算过程,即通过超几何分布等算法,从GO term tree上找到和input gene list显著相关的功能term。GO Term Finder为我们提供了这样的算法和分析过程。
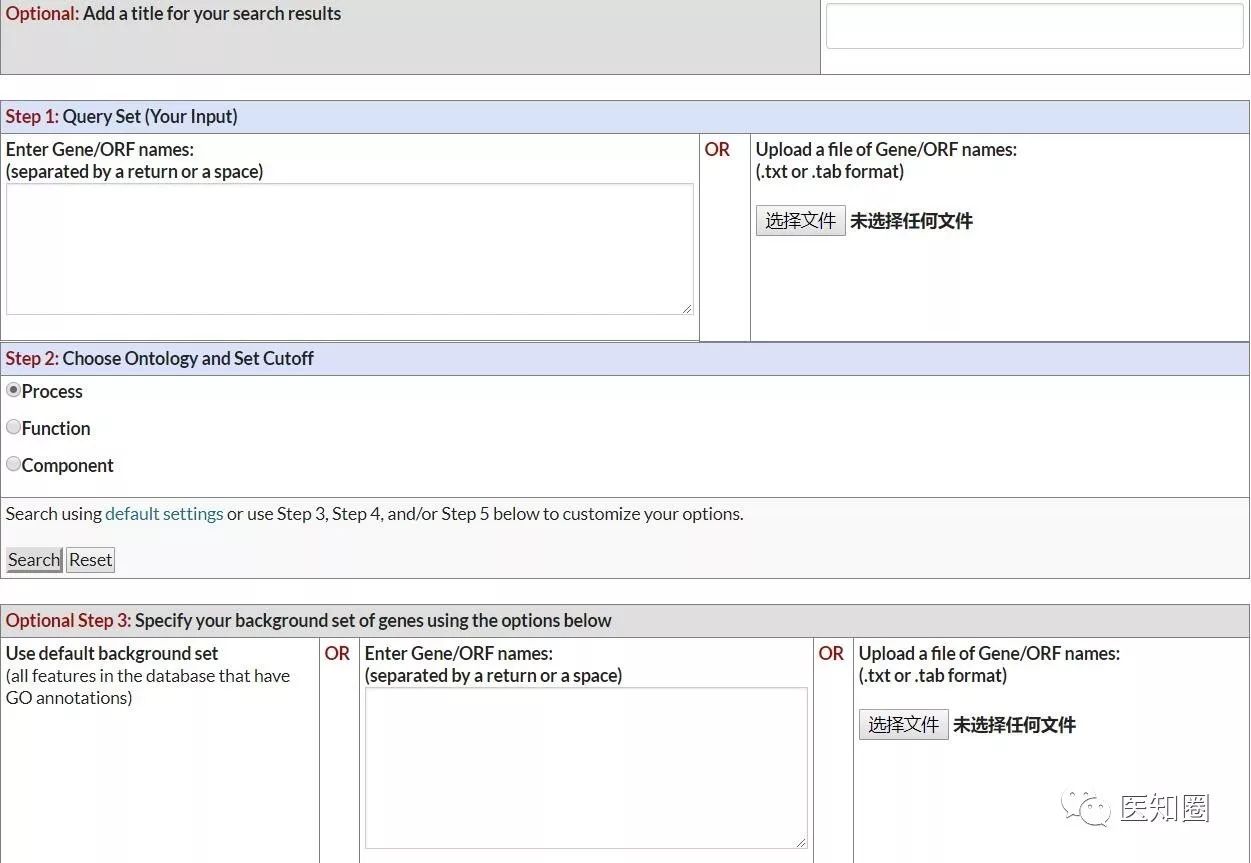
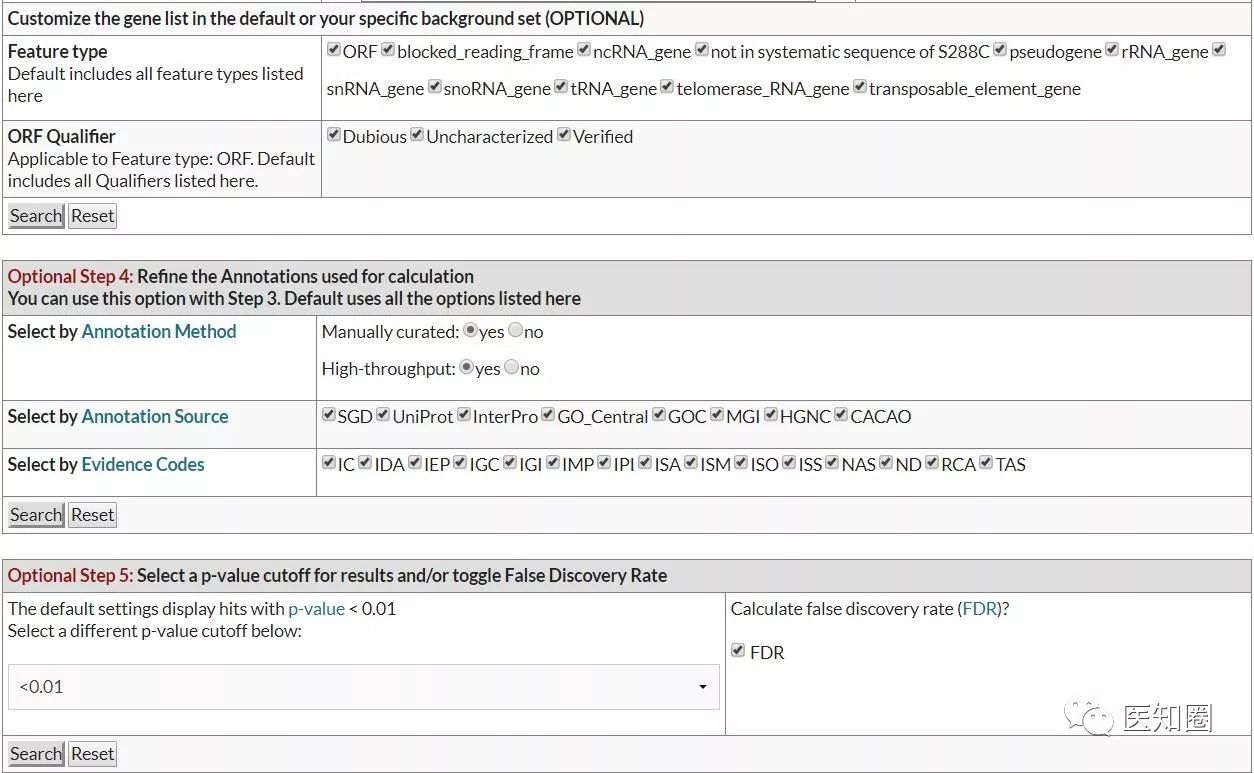
我们看下帮助文件:
图为Go Term Finder的主界面,可以看到数据库中包括多个分析工具供用户选择。接下来就介绍一下用Go Term Finder进行GO 功能注释分析的过程。
第一步:导入gene list
第二步:基于超几何分布实现功能富集分析
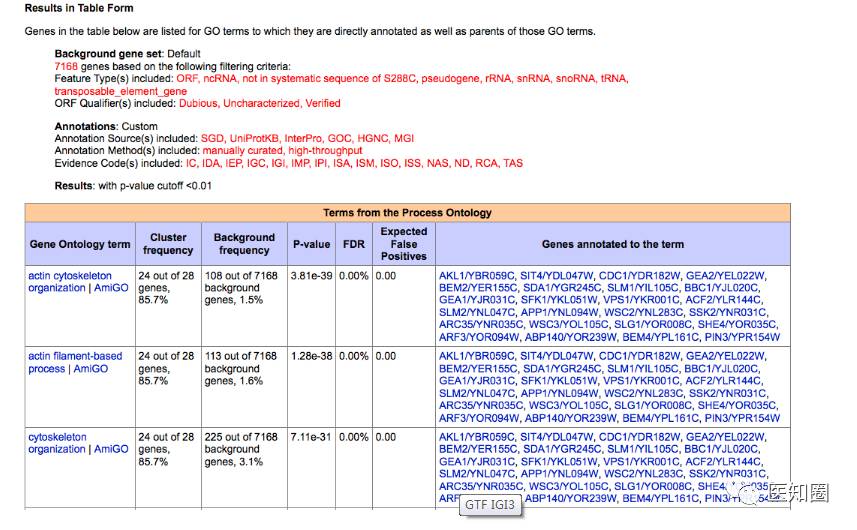
如图为富集出的结果,第一列为对应GO term,第二列为富集到对应term的基因占input基因的比例,第三列为背景频率,后面依次为超几何检验的P值,FDR校正,以及具体富集到term的基因。
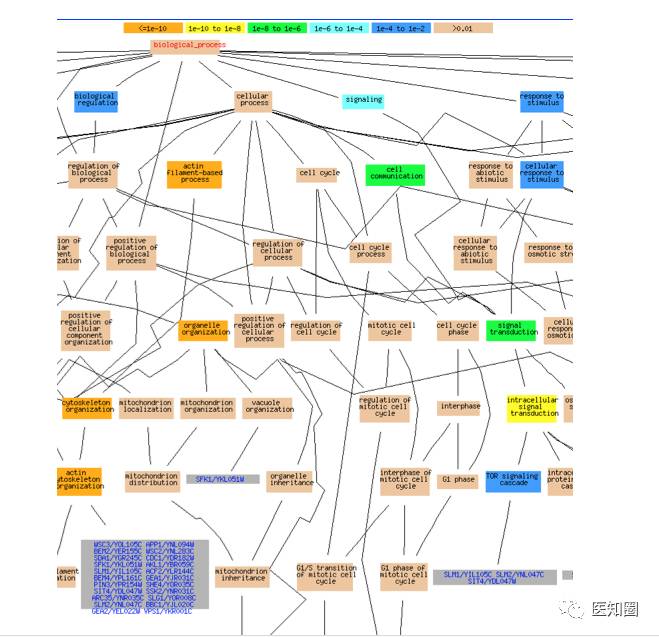
数据库还提供了可视化的结果,如图,我们可以看到由多个GO term构成的树形图。从上到下级别越来越低,上面的为父亲节点,下面的为子节点。同时用不同颜色进行标记,如橙色代表显著性P值小于1e-10,黄色term代表1e-10到1e-8。便于用户直观的分析显著的功能,以及其上下级的功能注释。

以上是关于GO Term Finder:一个强大的GO功能注释分析工具的主要内容,如果未能解决你的问题,请参考以下文章