MATLAB | 如何使用MATLAB绘制序列logo图
Posted slandarer
tags:
篇首语:本文由小常识网(cha138.com)小编为大家整理,主要介绍了MATLAB | 如何使用MATLAB绘制序列logo图相关的知识,希望对你有一定的参考价值。
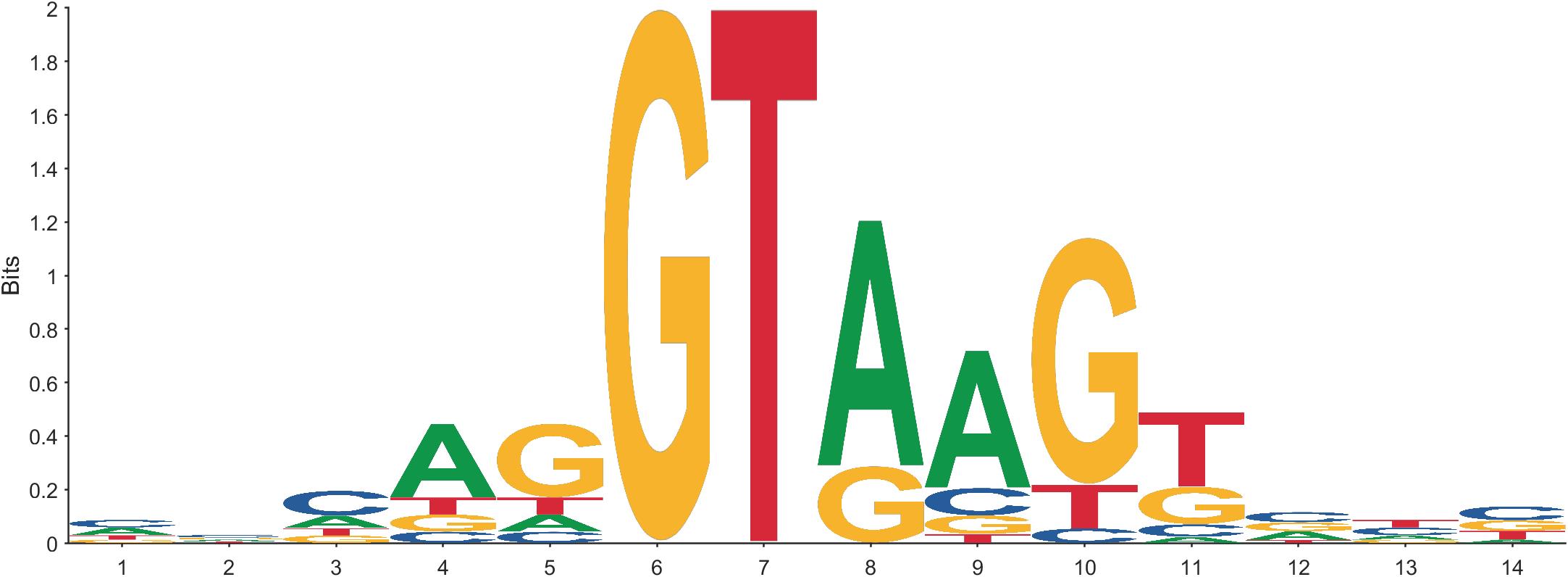
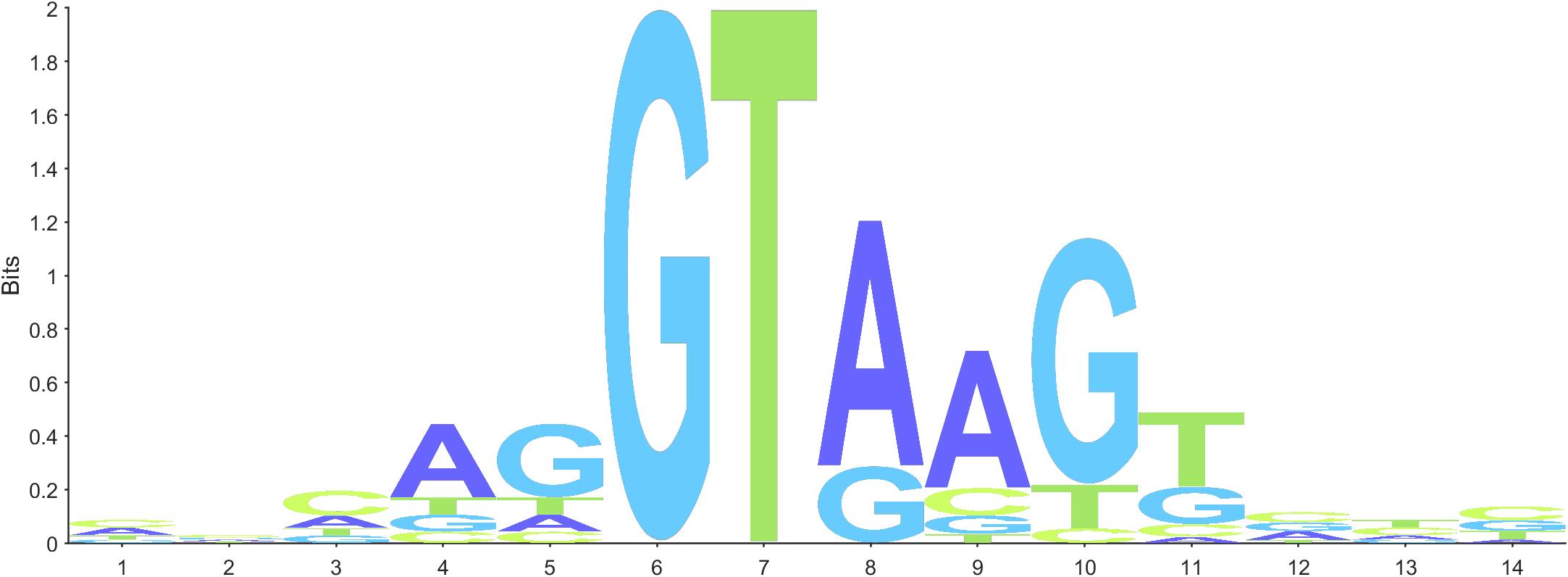
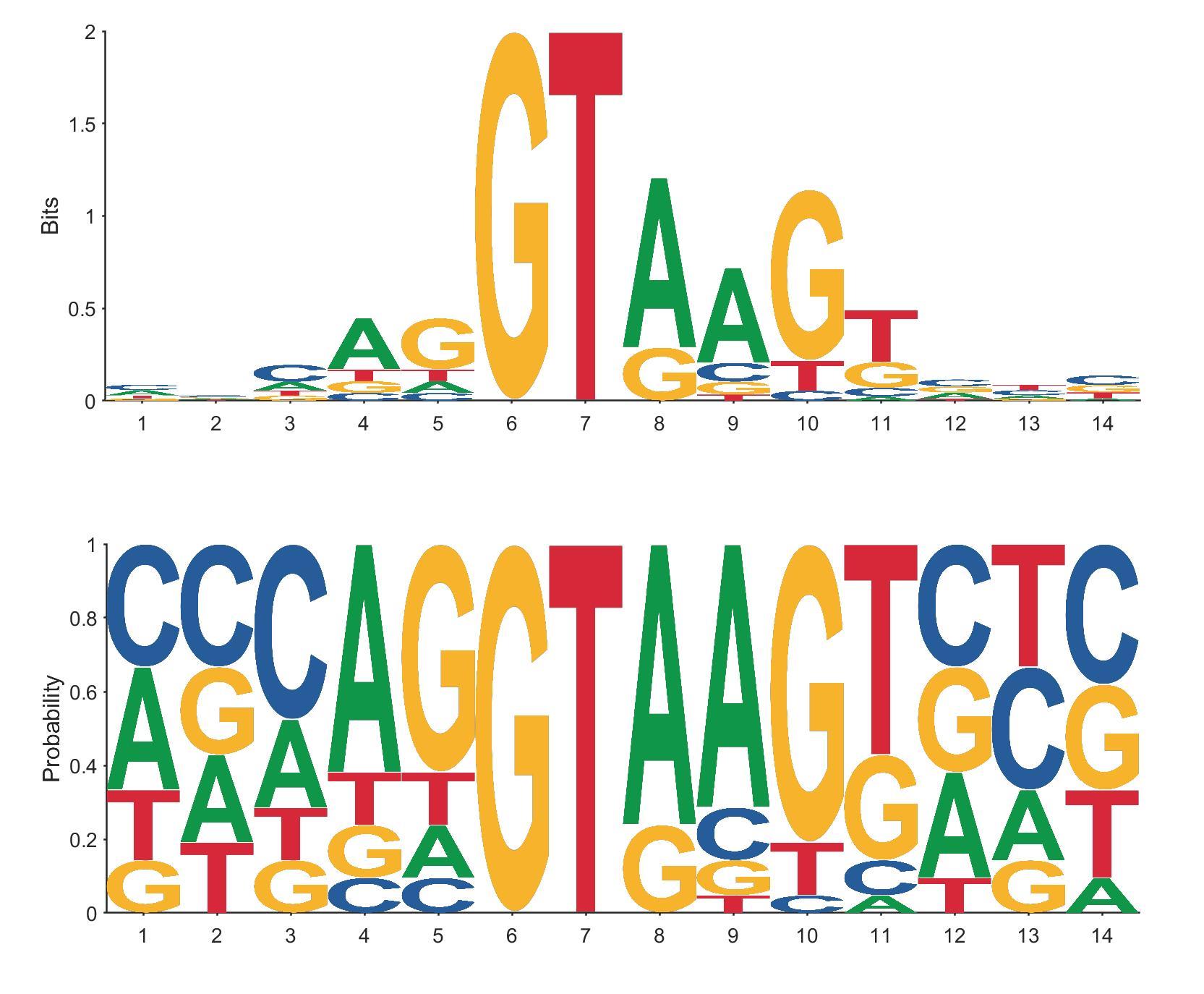
这次开发了一个生物信息学比较常用的序列logo图绘制MATLAB代码包,绘制效果如下:
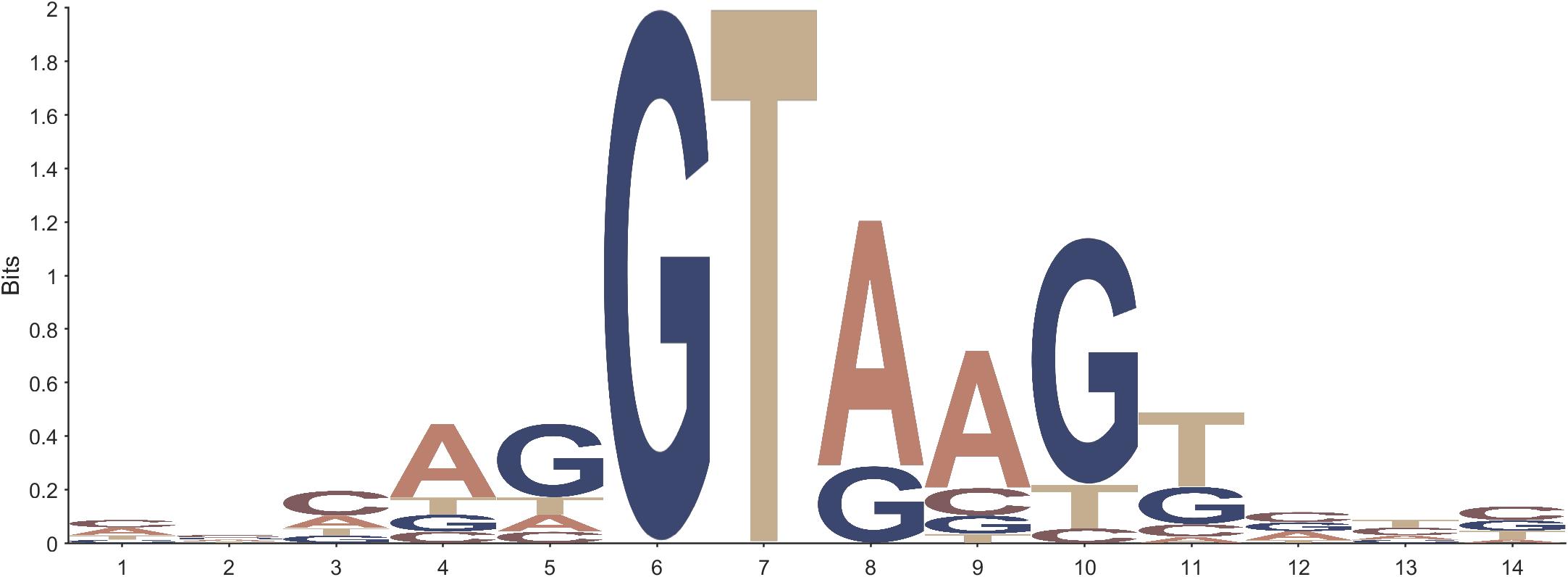
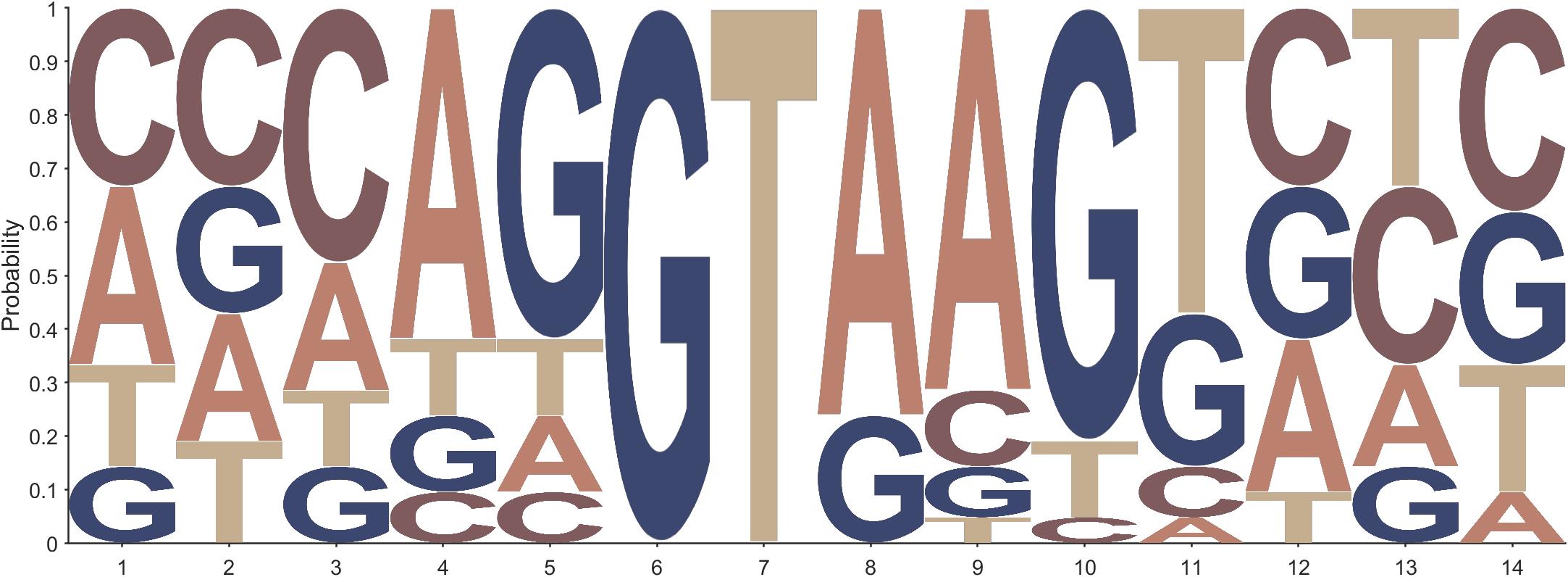
数据来自基迪奥生物项目编号为seqlogojrois9l2jit的示例数据。同时本工具函数参考以下文献:
- Tareen A, Kinney J B. Logomaker: beautiful sequence logos in Python[J]. Bioinformatics, 2020, 36(7): 2272-2274.
- Crooks G E, Hon G, Chandonia J M, et al. WebLogo: a sequence logo generator[J]. Genome research, 2004, 14(6): 1188-1190.
教程部分
1 数据格式
char矩阵
数据使用char类型矩阵,每一行代表一条序列:
Data =['GNYLGLTVETISRLL';
'GNYLGLTVETISRLL';
'GNYLGLTVETISRLL';
'GNYLGLTIETISRLL']
名称与序列txt
当然我们可以从txt文件中读取数据,若数据是这样一行名称一行序列的格式:
>seq01
ACCCTGTAAGTTTT
>seq02
TCAGTGTAAGTATC
>seq03
CATTCGTAAGTACC
>seq04
CGCTGGTAAGGACT
>seq05
ACCGGGTGAGCGCG
则可通过如下代码读取:
Data=readcell('seqlogo_DNA.txt');
Data=Data(2:2:end,1);
Data=reshape([Data:],[],length(Data))';
序列txt
若txt文件中只有序列:
GNYLGLTVETISRLL
ASYLGLRLETVCRSV
SEMTGTTLHTVSRLL
AEMTGTTLHTVSRIL
AEMTGTTLHTVSRIL
AEMTGTTVETTIRVM
ASRVGLTVQTVSTIV
AARLGLTPETFSRVL
ADYLGTTPETVSRTL
ADMLGSKRETVSRQL
ANYIGTSPETISRKI
ATFIGTTPETISRKF
SAFIGTTPETISRKL
ADVLGLSVVHMNRVI
ADALGLTPIHINRML
AEAIGSTRVTVTRLL
GNYLGLTVETISRLL
GNYLGLTVETISRLL
GNYLGLTIETISRLL
GNYLGLTIETISRLL
GNYLGLTVETISRLL
GNYLGLTVETISRLL
则可通过如下代码读取:
Data=readcell('seqlogo_protein_3.txt');
Data=reshape([Data:],[],length(Data))';
2 单位显示
有Bits和Probability供用户选择,默认bits。logo图纵坐标的单位常见有两种,一种是百分比,另一种是Bits。对于Probability,很好理解,每个字母的出现频率;对于Bits,可参考下面的公式:
R s e q = S max − S o b s = log 2 N − ( − ∑ n = 1 N p n log 2 p n ) R_s e q=S_\\text max -S_o b s=\\log _2 N-\\left(-\\sum_n=1^N p_n \\log _2 p_n\\right) Rseq=Smax −Sobs=log2N−(−n=1∑Npnlog2pn)
p n p_n pn是相应位置n上相应字符出现频率,N是不同字符的总数量(核酸为4,蛋白质为20)。因此,对于图中的y轴的最大数值就不难理解,核酸序列的最大值为 log 2 4 = 2 \\log_2 4 = 2 log24=2bits,蛋白序列为 log 2 20 ≈ 4.32 \\log_2 20≈4.32 log220≈4.32 bits。
可以通过设置函数的Method属性来调整显示单位,可选值为'Bits'/'Prob',其中Bits为默认值。
3 核酸序列绘制示例
将Type属性设置为NA即可绘制核酸序列logo图,当然参数的默认值就是NA所以不设置也可以,以下给出绘制核酸序列显示单位分别为Bits和Probability的绘制代码和绘图结果:
Data=readcell('seqlogo_DNA.txt');
Data=Data(2:2:end,1);
Data=reshape([Data:],[],length(Data))';
figure()
seqLogo(Data)
figure()
seqLogo(Data,'Method','Prob')

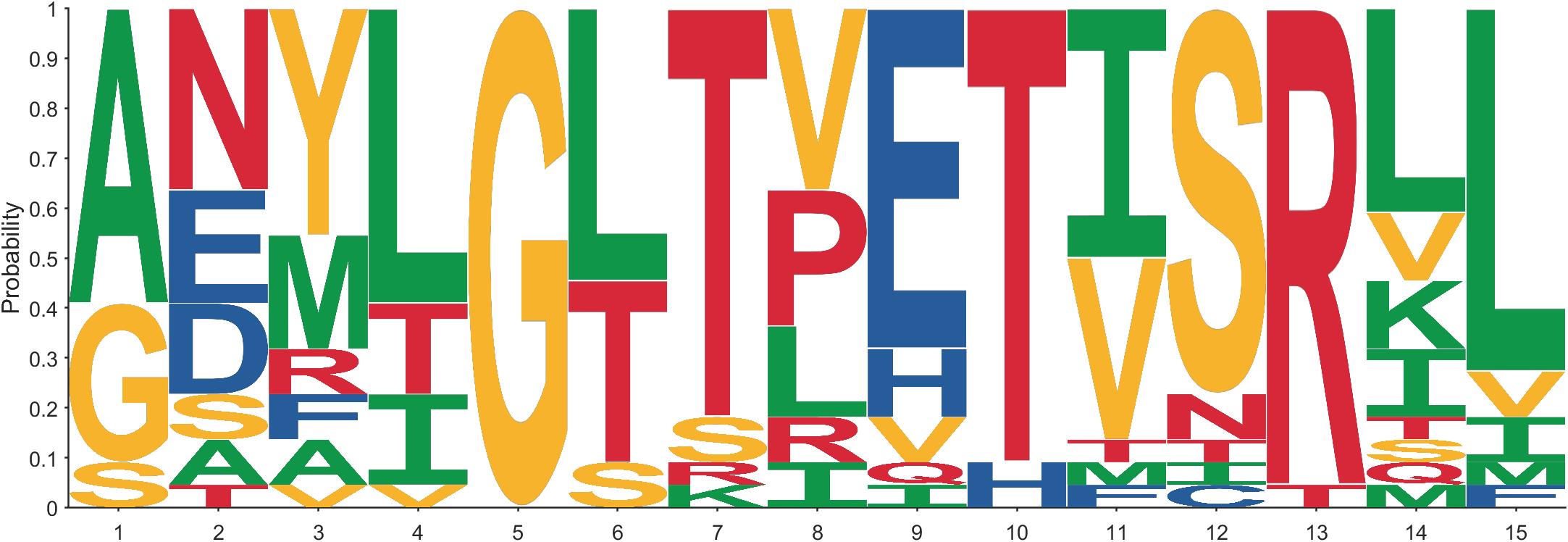
4 蛋白序列绘制示例
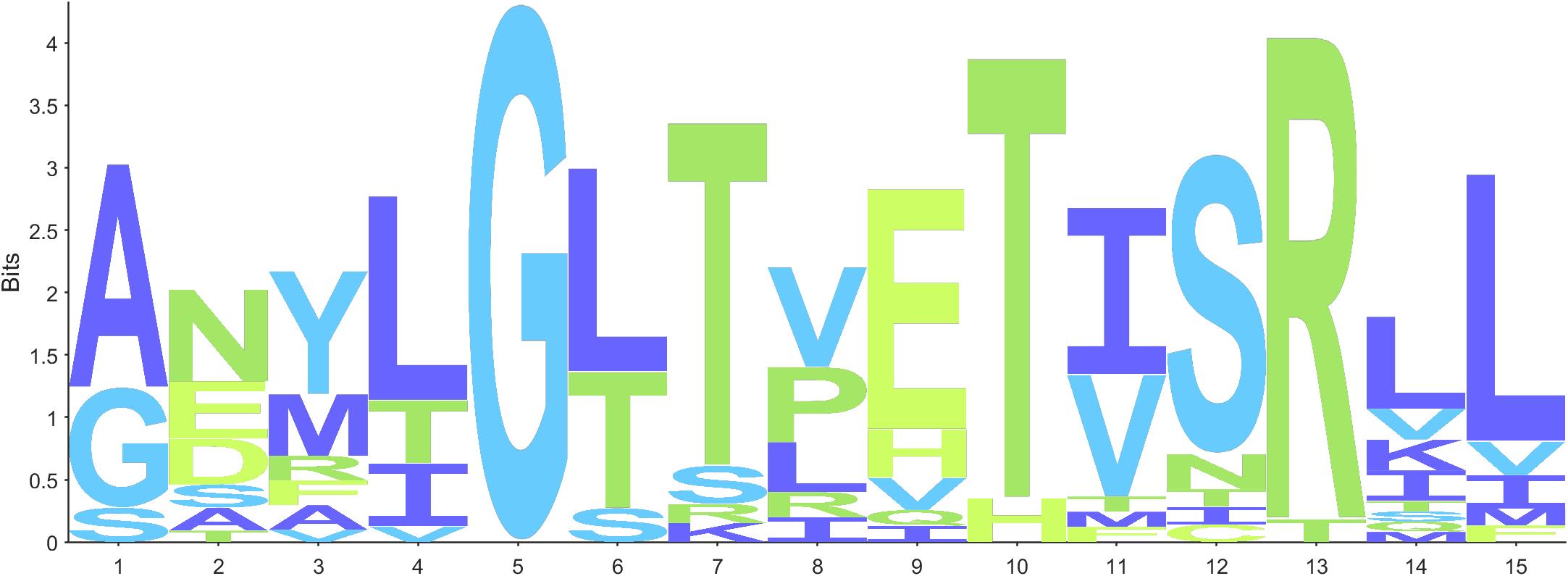
将Type属性设置为PR即可绘制蛋白序列logo图:
Data=readcell('seqlogo_protein_2.txt');
Data=Data(2:2:end,1);
Data=reshape([Data:],[],length(Data))';
figure()
seqLogo(Data,'Type','PR')
figure()
seqLogo(Data,'Type','PR','Method','Prob')

5 核酸序列配色
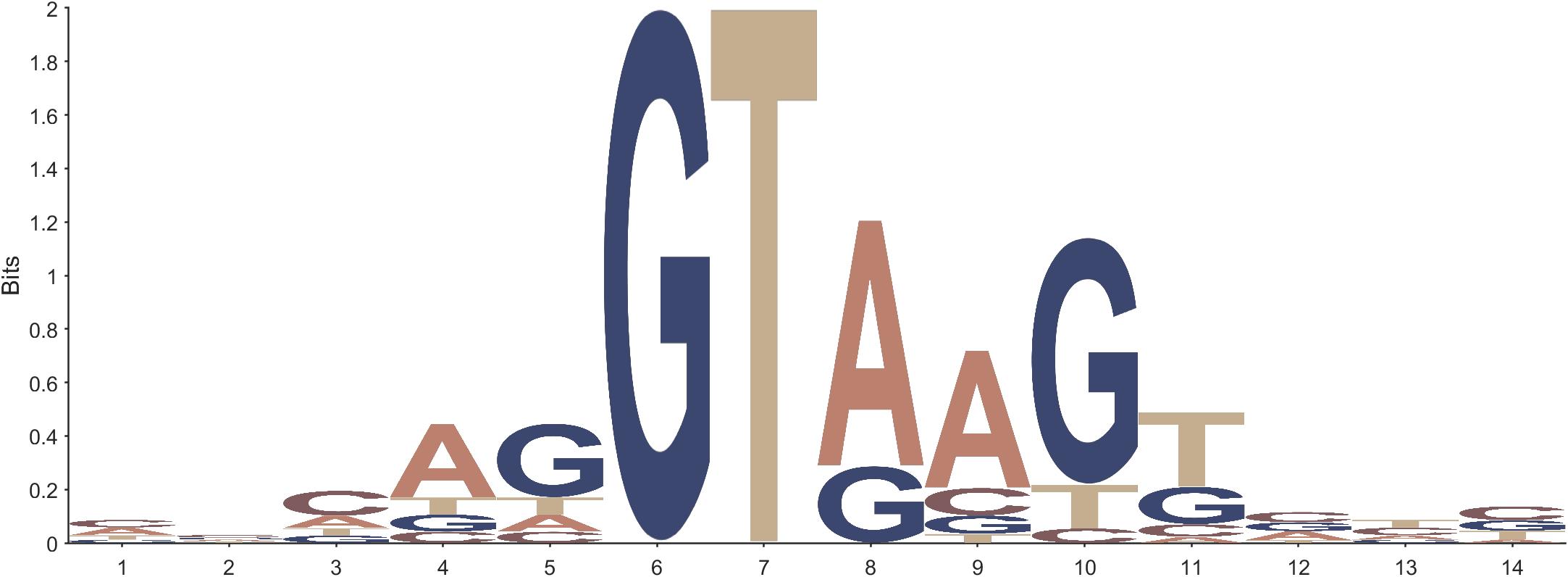
对于每个字母都可以单独设置颜色,自由度比较高,举个例子,将颜色设置为:
‘C’,[205,255,101]./255,
‘A’,[104,101,255]./255,
‘TU’,[164,230,101]./255,
‘G’,[104,203,254]./255:
Data=readcell('seqlogo_DNA.txt');
Data=Data(2:2:end,1);
Data=reshape([Data:],[],length(Data))';
Color='C',[205,255,101]./255,'A',[104,101,255]./255,'TU',[164,230,101]./255,'G',[104,203,254]./255;
% Color='C',[127,91,93]./255,'A',[187,128,110]./255,'TU',[197,173,143]./255,'G',[59,71,111]./255;
figure()
seqLogo(Data,'Color',Color)
figure()
seqLogo(Data,'Method','Prob','Color',Color)

设置为:
‘C’,[127,91,93]./255,
‘A’,[187,128,110]./255,
‘TU’,[197,173,143]./255,
‘G’,[59,71,111]./255
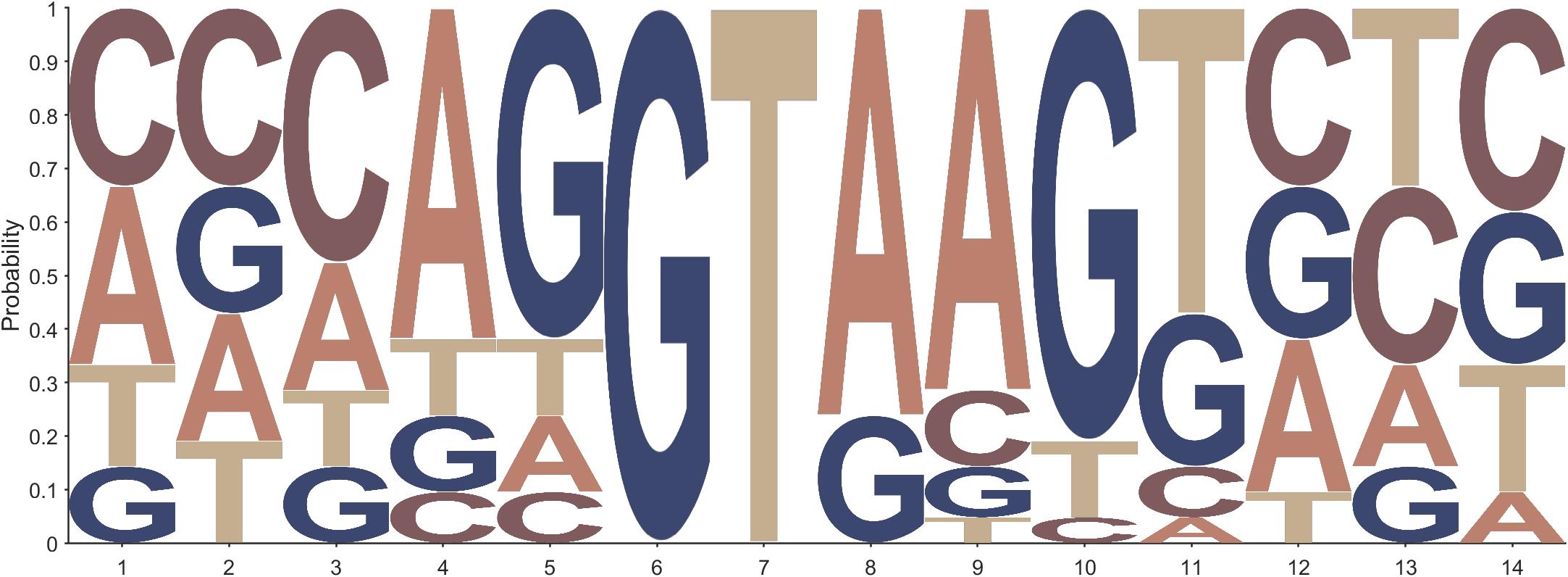
5 蛋白序列配色
对于每个字母都可以单独设置颜色,自由度比较高,下面可以设置任意多组颜色,随意划分分组:
Data=readcell('seqlogo_protein_3.txt');
Data=reshape([Data:],[],length(Data))';
Color='CDEFH',[205,255,101]./255,'AIKLM',[104,101,255]./255,'TUNPQR',[164,230,101]./255,'GSVWY',[104,203,254]./255;
% Color='CDEFH',[127,91,93]./255,'AIKLM',[187,128,110]./255,'TUNPQR',[197,173,143]./255,'GSVWY',[59,71,111]./255;
figure()
seqLogo(Data,'Type','PR','Color',Color)
figure()
seqLogo(Data,'Type','PR','Method','Prob','Color',Color)

.


6 子图
当然subplot函数啥的也可以用:
Data=readcell('seqlogo_DNA.txt');
Data=Data(2:2:end,1);
Data=reshape([Data:],[],length(Data))';
figure()
subplot(2,1,1)
seqLogo(Data)
subplot(2,1,2)
seqLogo(Data,'Method','Prob')
工具函数完整代码
仅代码无法运行,需要文件夹内有logoData.mat文件,此处仅做展示,完整代码+mat文件+示例数据可以从文末提供的MATHWORKS的fileexchange链接地址下载,或者从文末网盘链接下载。
function seqLogo(varargin)
% @author : slandarer
% gzh : slandarer随笔
% Zhaoxu Liu / slandarer (2023). sequence logos (序列logo图)
% (https://www.mathworks.com/matlabcentral/fileexchange/123060-sequence-logos-logo),
% MATLAB Central File Exchange. 检索来源 2023/1/10.
% =========================================================================
% Color | 配色 | 'A',[]./255,'C',[]./255,... ...
% Method | 比例计算方法 | Bits/Prob
% Type | 种类(核酸/蛋白质) | NA/PR
coe.arginList='Color','Method','Type';
% 数据预定义
logoData=load('logoData.mat');
coe.Color='CDEFH',[37,92,153]./255,'AIKLM',[16,150,72]./255,'TUNPQR',[214,40,57]./255,'GSVWY',[247,179,43]./255;
coe.Method='Bits';
coe.Type='NA';
% 坐标区域获取
if isa(varargin1,'matlab.graphics.axis.Axes')
coe.ax=varargin1;varargin(1)=[];
else
coe.ax=gca;
end
% 获取其他数据
coe.Data=varargin1;varargin(1)=[];
for i=1:2:(length(varargin)-1)
tid=ismember(coe.arginList,varargini);
if any(tid)
coe.(coe.arginListtid)=varargini+1;
end
end
% 获取版本信息
tver=version('-release');
verMatlab=str2double(tver(1:4))+(abs(tver(5))-abs('a'))/2;
if verMatlab<2017
hold on
else
hold(coe.ax,'on')
end
% 颜色计算
coe.CData=zeros(length(logoData.logoName),3);
for i=1:2:length(coe.Color)
tLogo=coe.Colori;
for j=1:length(tLogo)
tPos=find(logoData.logoName==tLogo(j));
coe.CData(tPos,:)=coe.Colori+1;
end
end
% 统计基因出现次数
coe.Count=zeros(length(logoData.logoName),size(coe.Data,2));
for i=1:length(logoData.logoName)
coe.Count(i,:)=sum(coe.Data==logoData.logoName(i),1);
end
% 开始绘图
if strcmpi(coe.Method,'Prob')
coe.ax.YLim=[0,1];
coe.ax.DataAspectRatio=