论文阅读MONAI Label:人工智能辅助的 3D 医学图像交互式标注框架
Posted Tina姐
tags:
篇首语:本文由小常识网(cha138.com)小编为大家整理,主要介绍了论文阅读MONAI Label:人工智能辅助的 3D 医学图像交互式标注框架相关的知识,希望对你有一定的参考价值。
Abstract
缺乏带注释的数据集是训练监督 AI 算法的主要挑战,因为手动注释既昂贵又耗时。为了解决这个问题,我们提出了 MONAI Label,这是一个免费的开源平台,有助于开发基于 AI 的应用程序,旨在减少注释 3D 医学图像数据集所需的时间。
通过 MONAI Label,研究人员可以开发专注于其专业领域的注释应用程序。它允许研究人员轻松地将他们的应用程序部署为服务,临床医生可以通过他们首选的用户界面使用这些服务。目前,MONAI Label 很容易支持本地安装(3DSlicer)和基于 Web(OHIF)的前端,并提供两种主动学习(Active learning)策略来促进和加速分割算法的训练。
MONAI Label 允许研究人员通过将其提供给其他研究人员和临床医生等方式对他们的标注应用程序进行渐进式改进。最后,MONAI Label 提供了示例标签应用程序,即 DeepEdit 和 DeepGrow,展示了注释时间大幅减少。
Introduction
目前的标注方法: 自动和半自动分割方法可作为医学图像和生物医学图像分析的开源软件包的一部分。例如,ITK-SNAP[16]提供了半自动活动轮廓分割[17];3D Slicer [18]和 MITK[19],基于边界点的自动分割[20、21], 通过NVIDIA Clara AI辅助注释提供了DeepGrow[9]以及其他半自动分割方法,如区域增长[23]和水平集[24]
另一方面,ilastik[25]等生物医学图像分析工具有助于在分割、目标检测、计数和跟踪等任务中使用机器学习算法。然而,ilastik不包括训练深度卷积网络的选项,这使得该系统与其他使用深度学习算法的工具相比处于劣势。另一个流行的生物医学图像分析开源平台是BioMedisa[26,27],一个在线平台为大体积图像的半自动分割开发的平台。它还提供了不同的基于GPU的算法和加权随机游动,用于预分割切片的智能插值。与基于CPU的半自动分割工具相比,Biomedisa在生物医学图像分析方面具有显著优势。它是专门开发用于集群或并行计算机架构的。这意味着,将Biomedisa与单个小型GPU结合使用并不总是那么容易
除了开源软件包外,商业解决方案中还提供了用于标注图像的自动和半自动方法,如SuperAnnotate(https://superannotate.com/)、V7 Labs(https://www.v7labs.com/)和Segments(https://segments.ai/)。然而,这些平台背后的算法细节不是开源的,这使得它们对科学界的吸引力降低
MONAI Label是一个免费、智能的开源图像标注和学习工具,使用户能够创建标注的数据集,并建立基于人工智能的标注模型,用于临床评估。它使研究人员能够以无服务器的方式使用标签应用程序建立、训练和执行推理,其中定制的标签应用程序通过MONAI Label服务器作为一项服务暴露。MONAI Label目前提供三种分割方法:两种互动式(DeepEdit和DeepGrow)和一种非互动式(自动分割),它基于任何类型的UNet。除此之外,它还提供了基于涂鸦的交互式分割方法,该方法依靠基于能量的优化来提出或完善分割标签[28, 14, 29]。
MONAI Label提供主动学习策略,以促进三维医学图像分割的深度学习算法的训练。具体来说,它计算出不确定值和认识不确定值,对未标记的图像进行排序,使临床医生/用户能够首先分割较难的样本。它还支持基于网络的医学成像的DICOMweb标准,使用户能够将MONAI标签整合到他们的图片存档和通信系统(PACS)、XNAT[30]、图像数据共享(IDC)[31]或任何其他DICOM系统。这一功能允许网络开发者使用行业标准的工具集来释放医疗图像的力量。此外,MONAI 标签支持两种流行的图形用户界面 (GUIs)。3DSlicer和OHIF。见图
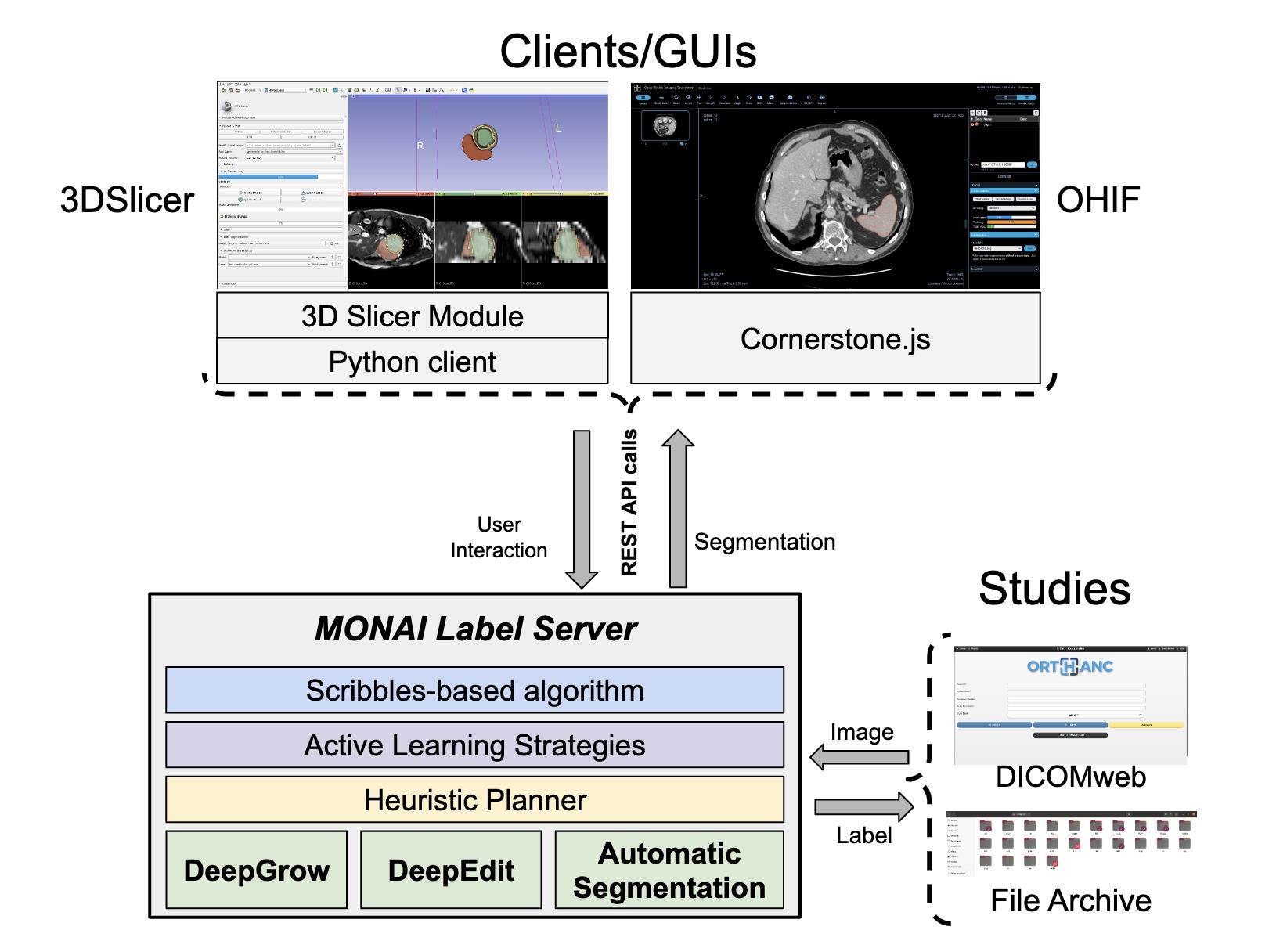
MONAI Label 平台概述: MONAI Label 由三个主要的高级模块组成,即(i)客户端 Clients/GUIs、(ii)服务器 Server 和(iii)Studies。
在客户端,MONAI Label 支持不同的图形用户界面 (GUI) (3DSlicer/OHIF) 用于数据查看和注释。
在服务器端,提供了一系列 AI 辅助的交互式标注方法,包括基于点击的 DeepEdit 和 DeepGrow 以及基于涂鸦(scribbles-based)的标签细化算法。该服务器还包括主动学习策略和启发式规划器,用于提高底层注释方法的效率。
Studies提供要注释的数据集的数据库,以及存储标注结果
DeepGrow
DeepGrow 是一种交互式分割模型,用户通过积极和消极的点击来指导分割。感兴趣区域内的正面点击会扩展分割以包括该位置,而负面点击用于收缩分割以从感兴趣区域中排除点击区域[9]。 DeepGrow 模型的训练过程不同于传统的深度学习分割,因为训练过程中涉及正负引导(点击)的模拟过程。正负引导图是基于依赖于预测的假阴性和假阳性生成的。 DeepGrow 2D 和 3D 都允许用户一次只标注一个标签。 DeepGrow 2D 允许用户一次注释图像一个切片,而 DeepGrow 3D 可以注释整个体积
注:目前monailabel上没有区分2D和3D。
这个功能类似于区域生长,你在前景点一个点,他就会自动填充相似的区域。只不过这里用的底层算法是深度学习
Automated Segmentation
这种 MONAI 标签注释方法本质上是一种基于标准卷积神经网络(CNN)(即 UNet)。研究人员可以使用在MONAI Core(使用monai构建的模型)中创建的任何可用网络。在类和接口方面,该应用程序遵循与DeepGrow和DeepEdit方法相同的结构。这个注释应用程序和交互式应用程序之间最重要的区别是添加到输入图像中的额外信息。在此应用程序中,表示前景和背景点击的额外通道不会添加到输入图像中。请注意,然后可以使用客户端中提供的标准分割工具手动进一步编辑自动分割。
大概意思是:使用预训练好的模型先执行一次分割,如果分割效果不理想,可以在 3D slicer 上面手动修正。
图形用户界面
3DSlicer(适用于本地安装)
3DSlicer 是一个开源多平台软件包,广泛用于生物医学和医学成像研究 [18]。与 MONAI Label 一起工作的模块处理由用户交互创建的调用/事件。当前版本支持点击交互并允许用户上传图片和标签。 MONAI 标签服务器支持其他交互,例如闭合曲线、ROI 或任何其他。研究人员可以修改此模块以使其更具动态性或针对他们的 MONAI 标签分割应用程序进行定制
Open Health Imaging Foundation(OHIF)
适用于web端,比如要直接和PACS通信,从PACS获取数据,并返回结果。
OHIF 查看器是一个开源和基于 Web 的查看器 [32]。它基于 Cornerstone.js,并与支持 DICOMweb 的图像档案一起开箱即用。 MONAI Label 嵌入了 OHIF,它与 DICOMweb 服务器支持一起工作。它还允许用户创建自己的标签掩码并与 MONAI 标签服务器交互。同样重要的是要注意 OHIF 已集成到 XNAT 和其他大型计划/中心中,这意味着它更容易集成到复杂的工作流程中。
开发和部署 MONAI Label 应用程序
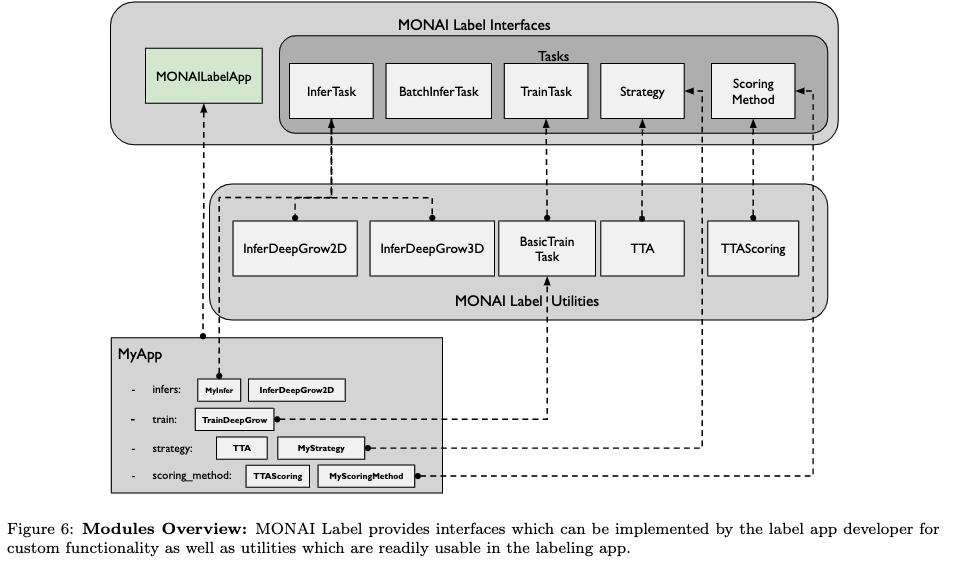
MONAI 标签应用程序开发人员可以选择在其应用程序中实现部分或全部以下功能
-
主动学习(Active Learning):MONAI Label 提供了实现自定义主动学习技术所需的所有必要回调。 MONAI Label 本身支持简单的主动学习技术,例如顺序和随机采样,以及更高性能的主动学习技术,例如测试时间增强 [37](任意不确定性)和使用 Dropout 的蒙特卡罗计算(认知不确定)
-
推理(Inference):此接口允许开发人员在 MONAI 标签应用程序中使用或定义推理任务,该任务将定义用户交互的行为(即点击、涂鸦、ROI 等)。开发人员将需要定义推理模型和推理器(简单推理器、滑动窗口推理器等),以及 pre- and post-transform。
-
训练(training):此界面允许开发人员为他们自己的 MONAI 标签应用程序定义训练任务,其中可能包括一个或多个具有适当预处理和后处理的模型。在这里,开发人员还决定了最佳的数据拆分策略和验证指标
-
数据变换(transforms): 在此文件中,开发人员可以在训练深度学习算法之前、之后或期间创建自定义转换以应用于数据集
-
主模块(Main Module):开发者定义App的核心结构,并声明上面定义的模块,为每个模块提供ID,以便它们可以独立使用,也可以根据需要一起使用
-
需求文件:在这里,研究人员可以选择指定他们的应用程序所需的外部库(例如 TensorFlow、Catalyst、Kornia [38] 等)
Discussion
我们推出了 MONAI Label,这是一个免费和开源的图像标记和学习工具,使用户能够创建带注释的数据集并构建用于临床评估的 AI 注释模型。 MONAI Label 减少了注释新数据集的时间和精力,并通过两个不同的用户界面:3DSlicer 和 OHIF 不断学习用户交互,从而使 AI 适应手头的任务
MONAI Label 采用三种主要的不同标注算法:两种交互式(DeepGrow 和 DeepEdit),以及一种非交互式自动分割算法。除此之外,还可以使用基于涂鸦的算法来快速分割图像。
MONAI 标签平台与 MONAI Core之外的其他库兼容。它的开发旨在帮助研究人员和临床医生促进 3D 医学图像注释,并允许他们轻松实施新的机器学习或深度学习算法。出于这个原因,我们设计它的方式是让用户可以无缝地使用不同的深度学习框架和高级库,如 Ignite 和 PyTorch Lightning
最后,MONAI Label 提供了一个启发式规划器(heuristic planner),它考虑可用的 GPU 和训练集的强度和空间信息,以定义训练和推理期间使用的数据转换和超参数
以上是关于论文阅读MONAI Label:人工智能辅助的 3D 医学图像交互式标注框架的主要内容,如果未能解决你的问题,请参考以下文章