如何用DPS软件做多重数据的差异显著性分析 输出结果有 a ab b的
Posted
tags:
篇首语:本文由小常识网(cha138.com)小编为大家整理,主要介绍了如何用DPS软件做多重数据的差异显著性分析 输出结果有 a ab b的相关的知识,希望对你有一定的参考价值。
参考技术A四张图
图一 选择数据
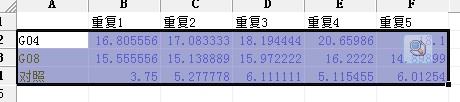
图二 统计运算命令
图三 选项设置 (LSD最小显著差数法,SNK也称q法或复极差检测或Student-Newman-Keul法,新复极差法也称SSR或最短显著极差法,自己选吧,当大于等于三组时,显著尺度SNK>SSR>LSD)

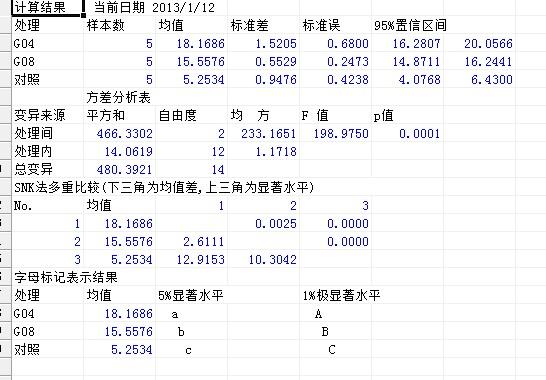
图四 结果显示(不解释了,一目了然)
DPS数据处理系统是浙江大学研制多功能数理统计和数学模型处理软件系统。它将数值计算、统计分析、模型模拟以及画线制表等功能融为一体。DPS系统兼有Excel电子表格软件和若干专业统计分析软件的功能。与Excel电子表格软件比较,DPS平台具有更加强大的统计分析和数学模型模拟分析功能。与国外同类专业统计分析软件系统(如SAS、STAT、STATISTICA等)相比,DPS系统具有操作简便,在统计分析和模型模拟方面功能齐全,易于掌握,其工作界面友好。
2 在药学领域中的应用
2. 1 数据基本参数计算 试验资料经整理之后,可以计算出一系列的统计指标以说明资料的特征和对资料进行进一步的统计分析。最常用的统计指标是和( sum) 、均值(mean) 、方差( variance) 、标准差( standard deviation) 、斜差( skewness)和峰度( kurtosis)等。
例1,从某药厂生产的六味地黄胶囊中随机抽取10粒,测
得胶囊装量为0. 315 1, 0. 320 4, 0. 324 2, 0. 319 0, 0. 332 0,0. 326 1, 0. 324 7, 0. 334 3, 0. 328 9, 0. 311 6 g,试计算该组数据的均数、几何均数、中位数、标准差、样本方差。操作步骤: ①在DPS工作表中按行输入数据,再选中数据区域(单击并拖动) ,将资料定义成数据块; ②单击菜单栏中“数据分析”→“基本参数估计”,就可立即得到这些基本参数。见图1。图1下部的计算结果, 得到偏度系数skew 等于- 0. 196 16, uskew =- 0. 285 51,显著水平P = 0. 775 26;因P > 0. 05,说明资料所属总体分布的偏度系数γ1为0。又峰度系数kurt等于- 0. 67727, ukurt = - 0. 507 60,显著水平P = 0. 611 73;因P > 0. 05,说明资料所属总体分布的峰度系数γ2为0,即属于正态峰度。因此,这10粒胶囊的装量数据属于正态分布。
图1 基本参数估计界面与结果
2. 2 两样本均值差异t检验 在DPS中,系统将同时给出如两分布方差相等时均值差异显著性、两分布方差不等时均值差异显著性、两分布各个样本数据配对时(两组数据的样本数配对、相等)其差值差异显著性以及当两分布方差差异显著时的均值检验结果。
例2,某药厂从丹参中提取丹参酮浸膏,为试验新工艺是否能提高出膏量,现采用新旧两种工艺,各试验10次,提取的干浸膏总量分别为,旧工艺: 78. 1, 72. 4, 76. 2, 74. 3, 77. 4, 78. 4,76. 0, 75. 5, 76. 7, 77. 3 g,新工艺: 79. 1, 81. 0, 77. 3, 79. 1, 80. 0,79. 1, 79. 1, 77. 3, 80. 2, 82. 1 g,新工艺是否提高出膏量? 操作步骤: ①在DPS工作表中将两个处理的样本观察值分两行填入,然后定义成数据块; ②按例1操作判断数据是否属于正态分布; ③单击菜单栏中“试验统计”→“次数分布及平均数比较”→“Student t测验”,就可立即得到分析结果。结果见图2。从图2可以看出,两个处理的均值分别为76. 23和79. 43,标准差分别为1. 823 3和1. 491 5。两分布方差齐性检验F = 1. 495 4,P = 0. 559 0,说明两总体方差齐性,这时,其均值差异显著性测验t = 4. 295 7,显著性水平P = 0. 000 4,差异有极显著性。以文献[ 4 ]的数据为例,按上述方法处理,结果与文献相同。
2. 3 方差分析 方差分析是以各数据来自正态、等方差这一条件为前提,当正态、等方差的条件不满足时,应将原始数据进行转换以满足正态、等方差条件后再作方差分析。DPS系统提供了4种数据转换的常用手段,分别是平方根转换,反正弦平方根转换、倒数转换。
图2 基本参数t检验界面与结果
例3,用3种不同的方法测定药物中某种成分的含量。第1法: 9. 29 % , 9. 44 % , 9. 33 % , 9. 56 %;第2法: 10. 16 % , 10. 08% , 10. 03 % , 10. 11 %;第3法: 10. 60 % , 10. 43 % , 10. 65 % ,10. 48 %。试判断3种测量方法测定结果有无显著性差异。操作步骤: ①在DPS工作表中将3个处理的样本观察值分3行填入,然后定义成数据块; ②按例1操作判断数据是否属于正态分布; ③单击菜单栏中“试验统计”→“方差齐性测验”,就可立即得到分析结果。见图3; ④单击菜单栏中“试验统计”→“完全随机设计”→“单因素试验统计分析”,按回车键执行该选项功能。这时系统将会提示用户选择数据转换方式,如此时直接回车表示不转换。选择数据转换方式后回车,系统将立即给出分析结果,包括: ①方差分析表,列出处理间和处理内的平方
和、均方以及F值,误差项在处理平方和内部是合并的; ②各个处理间的SSR检验。见图4。由图3中P > 0. 05,表明方差齐性。在分析结果中,如果P < 0. 05,则表明各个处理的方差具有显著的异质性。这时不宜采用方差分析方法进行统计检验,而可考虑对数据进行适当的转换,然后再作方差分析。图4多重比较结果,在DPS中,各个处理凡后面具有相同字母者,表示它们之间的差异不显著:只有当某两个处理后面跟的是完全不同的字母时,它们之间才有显著差异。结果3种测量方法测定结果差异有显著性。以文献[ 4 ]的数据为例,按上述方法处理,结果与文献相同。
图3 方差齐性检验界面与结果
3 讨论
在采用Excel电子表格程序进行t检验、方差分析时,程序没有提供数据的正态性检验和多方差齐性检验以及多个样本均数两两间的比较,需要另外编制程序来实现这些功能[ 5 ] 。而DPS数据处理系统则提供了这些工具,可以很方便地完成数据的正态性检验、多个方差齐性检验以及多个样本均数两两间的比较。本文介绍了定量资料统计分析最常用的数据基本参数计算、t检验、方差分析方法。结果准确,简便易行。
差异表达基因分析:差异倍数(fold change), 差异的显著性(P-value)
参考技术A Differential gene expression analysis:差异表达基因分析Differentially expressed gene (DEG):差异表达基因
差异表达分析是目前比较常用的识别疾病相关miRNA以及基因的方法,目前也有很多差异表达分析的方法,但比较简单也比较常用的是Fold change方法。
它的优点是计算简单直观,缺点是没有考虑到差异表达的统计显著性;通常以2倍差异为阈值,判断基因是否差异表达。Fold change的计算公式如下:
即用疾病样本的表达均值除以正常样本的表达均值。
差异表达分析的目的: 识别两个条件下表达差异显著的基因,即一个基因在两个条件中的表达水平,在排除各种偏差后,其差异具有统计学意义。我们利用一种比较常见的T检验(T-test)方法来寻找差异表达的miRNA。T检验的主要原理为:对每一个miRNA计算一个T统计量来衡量疾病与正常情况下miRNA表达的差异,然后根据t分布计算显著性p值来衡量这种差异的显著性,T统计量计算公式如下:
差异倍数(fold change)
fold change翻译过来就是倍数变化,假设A基因表达值为1,B表达值为3,那么B的表达就是A的3倍。一般我们都用count、TPM或FPKM来衡量基因表达水平,所以基因表达值肯定是非负数,那么fold change的取值就是(0, +∞).
为什么我们经常看到差异基因里负数代表下调、正数代表上调?因为我们用了log2 fold change。
当expr(A) < expr(B)时,B对A的fold change就大于1,log2 fold change就大于0(见下图),B相对A就是上调;
当expr(A) > expr(B)时,B对A的fold change就小于1,log2 fold change就小于0。
通常为了防止取log2时产生NA,我们会给表达值加1(或者一个极小的数),也就是log2(B+1) - log2(A+1). 【需要一点对数函数的基础知识】
为什么不直接用表达之差,差值接有正负啊?
假设A表达为1,B表达为8,C表达为64;直接用差值,B相对A就上调了7,C就相对B上调了56;用log2 fold change,B相对A就上调了3,C相对B也只上调了3.
通过测序观察我们发现,不同基因在细胞里的表达差异非常巨大,所以直接用差显然不合适, 用log2 fold change更能表示相对的变化趋势。
虽然大家都在用log2 fold change,但显然也是有缺点的:
一、到底是5到10的变化大,还是100到120的变化大?
二、5到10可能是由于技术误差导致的。所以当基因总的表达值很低时,log2 fold change的可信度就低了,尤其是在接近0的时候。
A disadvantage and serious risk of using fold change in this setting is that it is biased[7] and may misclassify differentially expressed genes with large differences (B − A) but small ratios (B/A), leading to poor identification of changes at high expression levels. Furthermore, when the denominator is close to zero, the ratio is not stable, and the fold change value can be disproportionately affected by measurement noise.
差异的显著性(P-value)
这就是统计学的范畴了,显著性就是根据假设检验算出来的。
假设检验首先必须要有假设,我们假设A和B的表达没有差异(H0,零假设),然后基于此假设,通过t test(以RT-PCR为例)算出我们观测到的A和B出现的概率,就得到了P-value, 如果P-value<0.05,那么说明小概率事件出现了,我们应该拒绝零假设,即A和B的表达不一样,即有显著差异。
显著性只能说明我们的数据之间具有统计学上的显著性,要看上调下调必须回去看差异倍数。
对于得到的显著性p值,我们需要进行多重检验校正(FDR),比较常用的是BH方法(Benjamini and Hochberg, 1995)。
这里只说了最基本的原理,真正的DESeq2等工具里面的算法肯定要复杂得多。
这张图对q-value(校正了的p-value)取了负log,相当于越显著,负log就越大,所以在火山图里,越外层的岩浆就越显著,差异也就越大。
只需要看懂DEG结果的可以就此止步,想深入了解的可以继续。
下面可以继续讨论的问题有:
1、RNA-seq基本分析流程/2、
2、DEG分析的常用算法/3、
3、常见DEG工具的方法介绍和相互比较
前言
做生物生理生化生信数据分析时,最常听到的肯定是“差异(表达)基因分析”了,从最开始的RT-PCR,到基因芯片microarray,再到RNA-seq,最后到现在的single cell RNA-seq,统统都在围绕着差异表达基因做文章。
(开个脑洞:再下一步应该会测细胞内特定空间内特定基因的动态表达水平了)
表达量 :我们假设基因转录表达形成的mRNA的数量反映了基因的活性,也会影响下游蛋白和代谢物的变化。我们关注的是 基因的表达 ,不是结构,也是不是isoform。
为什么差异基因分析这么流行?
一是中心法则得到了确立,基因表达是核心的一个环节,决定了下游的蛋白组和代谢组;
二是建库测序的普及,获取基因的表达水平变得容易。
在生物体内,基因的表达时刻都在动态变化,不一定服从均匀分布,在不同时间、发育程度、组织和环境刺激下,基因的表达肯定会发生变化。
差异基因分析主要应用在:
发育过程中关键基因的表达变化 - 发育研究
突变材料里什么核心基因的表达发生了变化 - 调控研究
细胞在受到药物处理后哪些基因的表达发生了变化 - 药物研发
目前我们对基因和转录组的了解到什么程度了?
基本的建库方法?建库直接决定了我们能测到什么序列,也决定了我们能做什么分析!
基因表达的normalization方法有哪些?
第一类错误、第二类错误是什么?
多重检验的校正?FDR?
10x流程解释
The mean UMI counts per cell of this gene in cluster i
The log2 fold-change of this gene's expression in cluster i relative to other clusters
The p-value denoting significance of this gene's expression in cluster i relative to other clusters, adjusted to account for the number of hypotheses (i.e. genes) being tested.
The differential expression analysis seeks to find, for each cluster, genes that are more highly expressed in that cluster relative to the rest of the sample. Here a differential expression test was performed between each cluster and the rest of the sample for each gene.
The Log2 fold-change (L2FC) is an estimate of the log2 ratio of expression in a cluster to that in all other cells. A value of 1.0 indicates 2-fold greater expression in the cluster of interest.
The p-value is a measure of the statistical significance of the expression difference and is based on a negative binomial test. The p-value reported here has been adjusted for multiple testing via the Benjamini-Hochberg procedure.
In this table you can click on a column to sort by that value. Also, in this table genes were filtered by (Mean UMI counts > 1.0) and the top N genes by L2FC for each cluster were retained. Genes with L2FC < 0 or adjusted p-value >= 0.10 were grayed out. The number of top genes shown per cluster, N, is set to limit the number of table entries shown to 10000; N=10000/K^2 where K is the number of clusters. N can range from 1 to 50. For the full table, please refer to the "differential_expression.csv" files produced by the pipeline.
不同单细胞DEG鉴定工具的比较
Comparative analysis of differential gene expression analysis tools for single-cell RNA sequencing data
For data with a high level of multimodality, methods that consider the behavior of each individual gene, such as DESeq2, EMDomics, Monocle2, DEsingle, and SigEMD, show better TPRs. 这些工具敏感性高,就是说不会漏掉很多真的DEG,但是会包含很多假的DEG。
If the level of multimodality is low, however, SCDE, MAST, and edgeR can provide higher precision. 这些工具精准性很高,意味着得到的DEG里假的很少,所以会漏掉很多真的DEG,不会引入假的DEG。
time-course DEG analysis
Comparative analysis of differential gene expression tools for RNA sequencing time course data
参考:
Question: How to calculate "fold changes" in gene expression?
Exact Negative Binomial Test with edgeR
Differential gene expression analysis
以上是关于如何用DPS软件做多重数据的差异显著性分析 输出结果有 a ab b的的主要内容,如果未能解决你的问题,请参考以下文章