circRNA鉴定工具之CIRIquant
Posted
tags:
篇首语:本文由小常识网(cha138.com)小编为大家整理,主要介绍了circRNA鉴定工具之CIRIquant相关的知识,希望对你有一定的参考价值。
参考技术ACIRIquant 与其他同类算法相比,其在鉴定circRNA的过程中降低了假阳性率。在circRNA的定量和差异表达分析方面,CIRIquant也考虑到RNase R处理不均等因素对最终结果的影响,通过qPCR验证其结果相较于其他方法拥有更高的准确度。同时,CIRIquant还提供对circRNA与线性转录本比例的分析,为circRNA生物发生和调控的相关研究提供基础。
Softwares:
Python packages:
从 git-hub 或者 SourceForge 处获得最新版本
直接使用pip安装
pip install CIRIquant
参数使用:
注意:
CIRIquant需要输入一个配置文件
对于线下提供的circRNA定量表需要有junction位点的bed文件,第4列的格式必须为 “chrom:start|end” 。例如:
Usage
推荐:使用CIRI2进行circRMA的预测
使用提供的bed文件时:
使用其它鉴定工具时:
例如使用find_circ鉴定的circRNA结果
The main output of CIRIquant is a GTF file, that contains detailed information of BSJ and FSJ reads of circRNAs and annotation of circRNA back-spliced regions in the attribute columns
CIRIquant 输出的是一个GTF文件,其中包括circRNA的BSJ和FSJ reads 的详细信息,以及属性列中对circRNA反向拼接区域的注释。
每一列的信息
attributes包含的key与value:
当拥有RNase R处理和未经处理的样本时,CIRIquant 可以评估RNase R数据中处理前的circrna的表达水平。
为了去除RNase R处理效果,需要两个步骤:
然后,CIRIquant将输出RNaseR数据中鉴定到的circrna的表达水平,标题行将包含额外的RNaseR处理效率信息。
Usage:
对于没有重复的样品,差异表达和差异剪接分析使用 CIRI_DE
输出结果
对于生物学重复,推荐使用edgeR分析,使用 prep_CIRIquant 去生成circRNA表达水平/junction比率的矩阵,使用 CIRI_DE_replicate 用于DE分析。
Step1: 准备CIRIquant 的输出文件
需要提供一个文本文件,包含样本信息和CIRIquant输出的GTF文件的路径信息
默认情况下,前三列是必需的。对于paired 样本,还可以添加一个主题名称列。
注意: 如果使用 CIRI_DE 进行差异表达分析,第3列group列必须是“C”或者“T”
然后,运行 prep_CIRIquant 汇总所以样本的circRNA的表达量
输出的结果可以使用DEseq2与edgeR进行分析。
Step2: 准备 StringTie 的输出
StringTie的输出应该位于 output_dir/gene/prefix_out.gtf 下。 需要使用stringTie中的 prepDE.py 来生成用于标准化的基因计数矩阵。
例如,可以提供一个 sample_gene.lst 包含样本 ID 和 StringTie 输出路径的文本文件:
然后使用生成的 gene_count_matrix.csv 去运行 prepDE.py -i sample_gene.lst 。
Step3: 差异表达分析
使用 CIRI_DE_replicate 进行差异表达分析,需要安装edgeR包
输出结果是未过滤的,可以设定阈值对结果进行过滤。
参考资料:
全平台全维度脂质鉴定的新利器 -- LipidIMMS Analyzer
引言
脂质是细胞膜最主要的成分,具有许多重要的生理功能。对脂质的系统分析(脂质组学)近年来也是发展迅速。然而,由于脂质同分异构体众多,如何对脂质进行大规模地准确鉴定仍然是当前最大的难点之一。目前最常用的脂质鉴定工具(数据库)包括:LipidMaps, LipidBlast, Lipid Data Analyzer,以及商用的软件SimLipid等等。
那么还有没有更简单好用,经济免费的工具呢?
今天,小编要给大家介绍一个新的利器,LipidIMMS Analyzer(重要提示:后文附带详细的使用说明)。
这项工作最近发表在Bioinformatics杂志上 [1] 。通讯作者是中科院上海有机化学研究所-生物与化学交叉研究中心的朱正江教授。
相比于传统的工具,该软件能够结合多个维度的信息,包括一级的精确质量(m/z),保留时间(retention time, RT),碰撞截面积(collision cross-section, CCS)以及二级碎片谱图(MS/MS),从而第一次实现了全维度高精度的脂质鉴定。
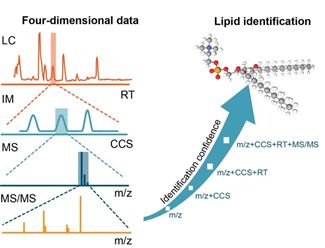
图一:基于全维度信息的脂质精确鉴定
与此对应,在该软件当中还内嵌了一个超级大的脂质数据库,包含了4个大类,25个小类,超过26万个脂质结构,每个脂质均含有四个维度的信息(m/z, RT, CCS, MS/MS)。在该数据库中,碰撞截面积和二级碎片谱图的精准预测主要依赖于该课题组早前开发的 LipidCCS [2] 和 LipidAnalyzer [3] 工具。此外,作者还提供了HILIC柱和RP柱上的两种不同的保留时间可以供用户个性化的使用。
那么问题来了,该工具是否只能支持离子淌度质谱呢?当然不是,实际上该工具可以支持全平台数据的脂质鉴定,不管是哪家的仪器,不管是离子淌度质谱(IM-MS),常规的LC-MS,甚至是Direct infusion - MS的数据,它通通能够处理。
既然这么强大,那么就来摆事实讲道理,实际操练一下吧。
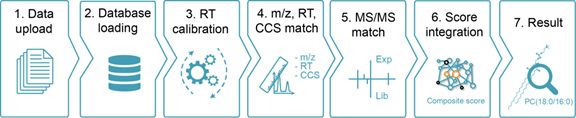
图二:基于全维度信息的脂质精确鉴定
总的来说,完成整个鉴定的过程只需要七步,而且每一步都可以进行交互式的鉴定,具体的数据格式和准备还请大家参考网站上所提供的教程。那么我们一步一步来简单了解一下吧~准备好了吗??
[1]. Z. Zhou, X. Shen, X. Chen, J. Tu, X. Xiong, and Z.-J. Zhu*, Bioinformatics, 2018, In Press.
[2]. Z. Zhou, J. Tu, X. Xiong, X. Shen, and Z.-J. Zhu*, Anal. Chem., 2017, 89, 9559–9566.
[3]. J. Tu, Y. Yin, M. Xu, R. Wang, and Z.-J. Zhu*, Metabolomics, 2018,14: 5.
 LipidIMMS Analyzer使用的简明教程
LipidIMMS Analyzer使用的简明教程
(建议在PC端使用)
LipidIMMS Analyzer

0数据准备
Peak table:csv文件,前三列分别为 “mz”, “rt”, “ccs”,后面为每个样本的峰面积。
MS/MS文件:可以支持MGF,CEF,MSP等,具体的导出方法请参考教程。
RT calibration table: csv文件,用于保留时间的校正。
注意:具体的数据格式和准备,还请大家参考LipidIMMS网站上所提供的教程。
1步骤一:上传数据
填写项目的名称,选择离子化的正负模式,仪器平台(比如Agilent),上传peak table(csv文件)和MSMS文件(MGF/CEF/MSP)即可,选择“Load data”。右边的结果展示栏中会展示上传的数据信息,以及绘制的peak profile图片。点击“Next”可以进入到下一步。在这里小编直接使用网站上所提供的demo数据。注意,如果是非离子淌度的数据,只需要把CCS这一列空着就好。
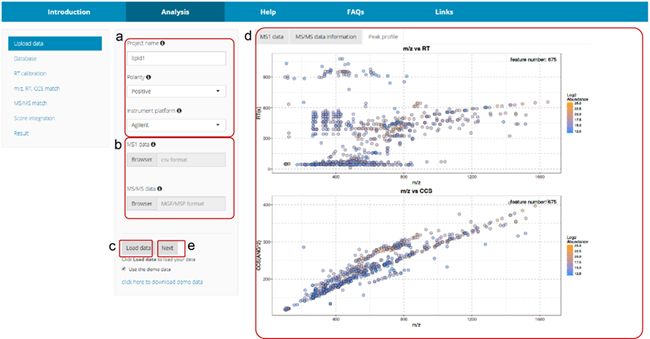
2步骤二:数据库加载
选择我们感兴趣的脂质类别,点击“Submit”,进行数据库的加载。这里,小编直接加载了所有的类别。


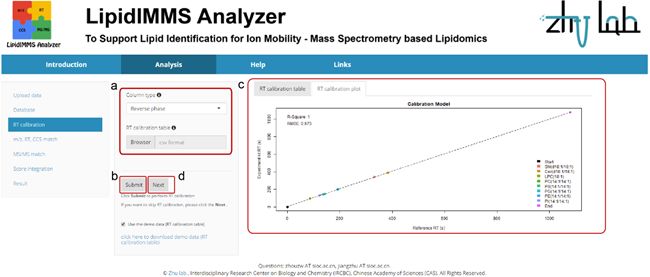
3步骤三:保留时间校正
选择色谱柱类型,比如小编选择的反相柱(reverse phase)。上传之前准备好的RT calibration table即可,点击“submit”。右边就会显示RT校正的效果啦。注意:这一步是后面进行RT match的前提,如果不需要进行RT match,可以直接选择“Next”。
4步骤四:m/z, RT, CCS匹配
大家可以根据自己的实验数据和需求,首先选择和调整匹配的方式和参数。比如我不希望进行保留时间匹配,那么直接勾掉“RT match”就可以,这样匹配的时候就不会去匹配RT,设置的RT match的参数也不会被使用。同理,比如我的数据是利用LC-MS采的,没有CCS值,那么就可以直接去掉“CCS match”,然后进行匹配。选择好匹配条件后,点击“Submit”即可。对于匹配过程中,每个参数的具体意义,大家还可以点击右边的感叹号小图标进行查看哦。

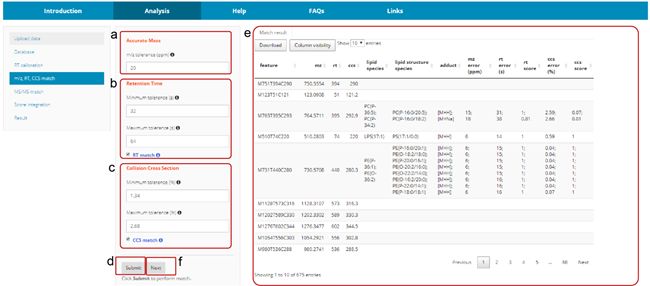
5步骤五:MS/MS匹配
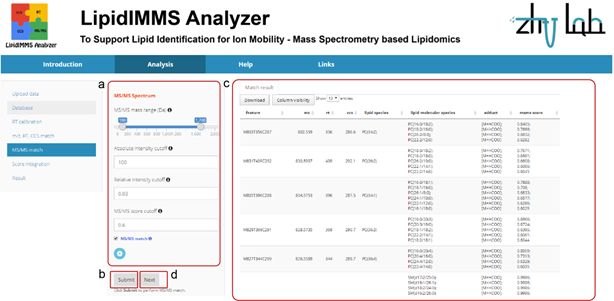
第五步是MS/MS的匹配。与上面相同,如果你不希望进行MS/MS匹配,可以直接去除“MS/MS Match”前面的勾,这样就不会进行MS/MS的匹配啦。设置好匹配的参数后,点击“Submit”就会进行匹配啦。匹配完成后,会在右边的结果展示表格中展示出MS/MS匹配的具体结果。
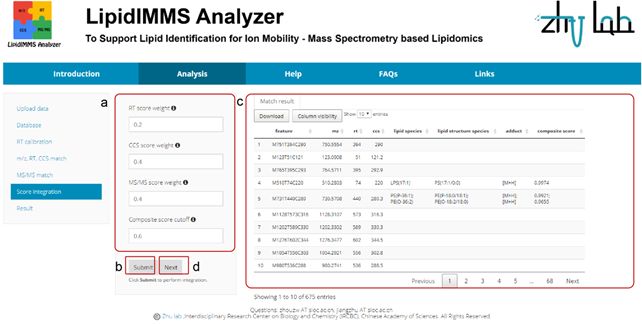
6步骤六:多维度得分整合
在完成前面的步骤之后,我们就可以进入得分整合的阶段。我们需要根据前面选择的匹配条件调整每个维度得分的权重,比如我们前面没有进行CCS 匹配,那么就需要将“CCS score weight”设为0。注意,三个维度得分权重的加和需要等于1。完成参数设置后,点击“Submit”即可,右边就会展示最终的鉴定结果。
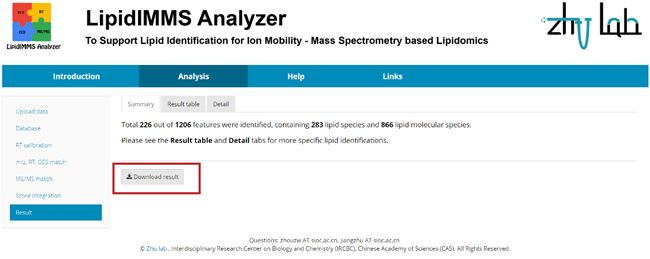
7步骤七:结果展示
在结果展示界面中,包含了三个选项卡,“Summary”,“Result table”和“Detail”。
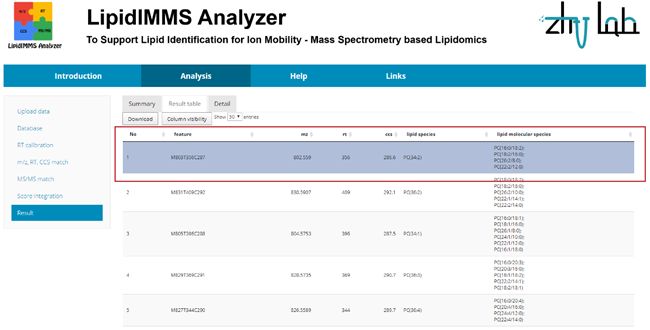
在Summary当中,会对数据处理的情况进行汇总。比如小编点点鼠标,就轻轻松松就鉴定到了226个脂质。同时,点击“Download result”就可以下载所有的鉴定结果,这些都会添加到“result table.csv”文件当中哦。
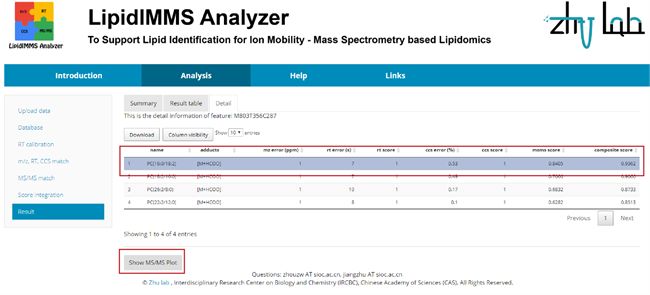
同时我们还可以在网站上“Result table”选项卡中,对鉴定结果进行浏览。此外,我们还可以选中每一个鉴定结果,并在“Detail”选项卡里去看具体的鉴定结果。在这里,小编选中第一个feature的鉴定结果,我们可以进一步点击“Detail”选项卡,这样我们可以看到更进一步的match结果。
而在“Detail”选项卡,我们还可以在选择任何一个candidate,点击屏幕下方的“Show MS/MS plot”选项卡来查看他们的MS/MS匹配结果。比如,小编选中了第一个candidate, PC(16:0/18:2),点击“Show MS/MS plot”,我们就可以更进一步地手动去核对我们的鉴定结果啦。
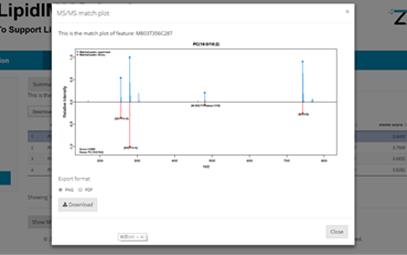
8结语:
关于LipidIMMS Analyzer使用的新技能,小编就介绍到这里啦。LipidIMMS有着强大的功能,等着大家去细细探索。衷心地希望今天介绍的这个工具能够对大家的科研有所帮助,也祝大家的科研越来越好!

以上是关于circRNA鉴定工具之CIRIquant的主要内容,如果未能解决你的问题,请参考以下文章