python 读取dicom tag 结果为空值
Posted
tags:
篇首语:本文由小常识网(cha138.com)小编为大家整理,主要介绍了python 读取dicom tag 结果为空值相关的知识,希望对你有一定的参考价值。
参考技术A 可能出现了中文乱码的问题。DICOM全称为Digital Imaging and Communications in Medicine,即医学数字成像和通信标准。本文中读取的CT图像就是使用DICOM标准存储的。其实不只CT图像,大部分临床影像都被存储为DICOM格式,如MR与PET图像。DICOM文件中除了包含有影像数据外,还囊括了大量机器,患者信息。这些信息被存储在一个个tags or attributes之下。Pydicom包为我们提供了非常简单易用的方法来读取这些文件。
Python 由 Guido van Rossum 于 1989 年年底出于某种娱乐目的而开发, Python 语言是基于 ABC 教学语言的,而 ABC 这种语言非常强大,是专门为非专业程序员设计的。但 ABC 语言并没有获得广泛的应用, Guido 认为是非开放造成的。
Python 的“出身”部分影响了它的流行,Python 上手非常简单,它的语法非常像自然语言,对非软件专业人士而言,选择 Python 的成本最低,因此某些医学甚至艺术专业背景的人,往往会选择 Python 作为编程语言。
Python对DICOM图像进行阈值分割
Python对Dicom图像进行处理,离不开pydicom,opencv-python,matplotlib,numpy四个代码库,安装完成这四个代码库后,
可以读取Dicom图像,并对图像进行处理,显式处理后的结果,下面就举例说明:
import cv2 import numpy import dicom from matplotlib import pyplot as plt # 读取单张Dicom图像 dcm = dicom.read_file("../Data/vhm.420.dcm") dcm.image = dcm.pixel_array * dcm.RescaleSlope + dcm.RescaleIntercept # 获取图像中的像素数据 slices = [] slices.append(dcm) # 复制Dicom图像中的像素数据 img = slices[ int(len(slices)/2) ].image.copy() # 对图像进行阈值分割 ret,img = cv2.threshold(img, 90,3071, cv2.THRESH_BINARY) img = numpy.uint8(img) # 提取分割结果中的轮廓,并填充孔洞 im2, contours, _ = cv2.findContours(img,cv2.RETR_LIST,cv2.CHAIN_APPROX_SIMPLE) mask = numpy.zeros(img.shape, numpy.uint8) for contour in contours: cv2.fillPoly(mask, [contour], 255) img[(mask > 0)] = 255 # 对分割结果进行形态学的开操作 kernel = cv2.getStructuringElement(cv2.MORPH_ELLIPSE,(2,2)) img = cv2.morphologyEx(img, cv2.MORPH_OPEN, kernel) # 根据分割mask获取分割结果的像素数据 img2 = slices[ int(len(slices)/2) ].image.copy() img2[(img == 0)] = -2000 # 显式原始数据,mask和分割结果 plt.figure(figsize=(12, 12)) plt.subplot(131) plt.imshow(slices[int(len(slices) / 2)].image, ‘gray‘) plt.title(‘Original‘) plt.subplot(132) plt.imshow(img, ‘gray‘) plt.title(‘Mask‘) plt.subplot(133) plt.imshow(img2, ‘gray‘) plt.title(‘Result‘) plt.show()
运行后的结果:
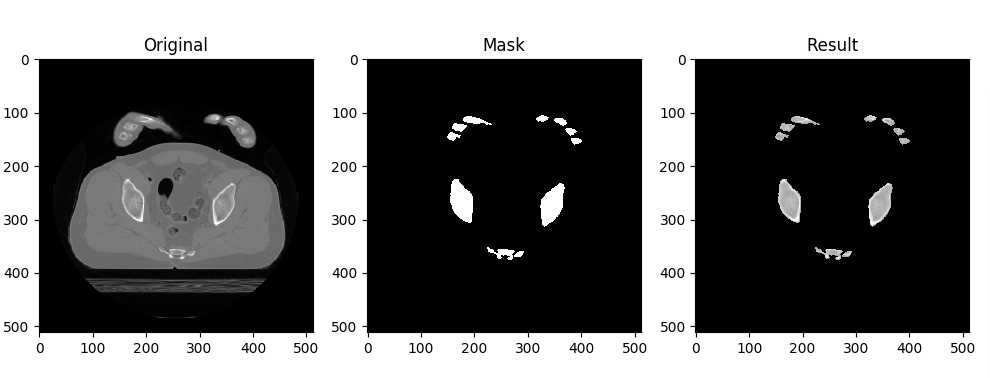
本例子以单张Dicom图像为例,分割图像中的骨骼信息,也可以同时读取多张Dicom图像,分割其他组织器官。
以上是关于python 读取dicom tag 结果为空值的主要内容,如果未能解决你的问题,请参考以下文章