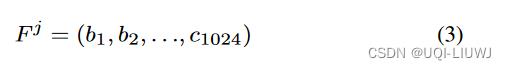论文笔记:BridgeDPI: a novel Graph Neural Network for predicting drug–protein interactions
Posted UQI-LIUWJ
tags:
篇首语:本文由小常识网(cha138.com)小编为大家整理,主要介绍了论文笔记:BridgeDPI: a novel Graph Neural Network for predicting drug–protein interactions相关的知识,希望对你有一定的参考价值。
2022 Bioinformatics
1 introduction
- 使用图神经网络预测药物和蛋白质的相互作用(DPI,drug-protein interaction)
- 目前预测药物和蛋白质相互作用的方式主要有以下几种:
- 基于对接的方法(docking-based)
- 使用分子动力学模拟重建蛋白质与药物在空间中的接触关系
- 目标是寻找药物分子在蛋白质上结合的最佳位点
- 缺点是需要精确的蛋白质结构,实际有时无法得到蛋白质的结构。
- 基于机器学习的方法
- 需要手动的特征
- ——>需要一定的专业知识和经验
- 需要手动的特征
- 基于深度学习的方法
- 早期的模型一般来说都是以下步骤:
- 编码蛋白质和药物
- 设计一个特征提取器,来提取蛋白质和药物的特征
- 融合蛋白质和药物的特征,通过全连接层进行预测
- 缺点是,这种类型的模型并没有考虑蛋白质之间(protein-protein association PPA)/药物(drug-drug association DDA)之间的关系
- 蛋白质通常和相似的药物有关联;药物也通常对相似的蛋白质起作用
- 故而,后来的模型融入DDA和PPA,来提升DPI的预测效果
- 早期的模型一般来说都是以下步骤:
- 基于对接的方法(docking-based)
- DDA和PPA的生成也有三种方法
- 基于结构的(structure-based)
- 可以很准确地得到PPA,但是受限于蛋白质结构数据比较稀少
- 基于序列的
- 如果两个蛋白关联的结构相似,其中一个可以与药物相互作用,另一个也可以与药物作用。
- 通常需要构建一个包含现有药物和蛋白质的网络,并计算药物对和蛋白质对的相似性得分。
- 缺点是比较依赖相似性得分的质量,且不能应用到低频或没有的蛋白质,有时候这种方法计算出来的结果并不一定是对的,有些类似的蛋白质不能与类似的药物相互作用。
- 基于方法的
- 以蛋白质序列和药物分子作为药物蛋白相互作用预测的输入。
- 缺点是模型没有网络层次的信息。
- 基于结构的(structure-based)
2 模型部分
2.1 整体模型

2.2 蛋白质和药物的embedding
- 使用经典的K-mer 嵌入表示
- K-mer表示k长度的氨基酸序列
- 论文中k取3
- 由于氨基酸有20种,所以1-mer是20个,2-mer是20*20个,3-mer是20*20*20个
- 所以用一个20+20*20+20*20*20=8420维的向量表示蛋白质嵌入
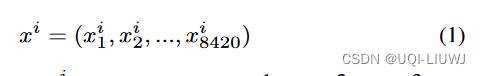
- 其中
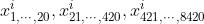 分别表示1-mer,2-mer和3-mer
分别表示1-mer,2-mer和3-mer - 对每个k-mer,进行正则化:
- K-mer表示k长度的氨基酸序列
- 使用Morgan FP来表示一个药物分子,这是一个1024维的向量
- 获得了蛋白质和药物的embedding之后,使用全连接层分别提取特征;同时使用CNN来提取药物和蛋白质的序列信息(我们记CNN的结果为
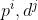 )
) - 所以我们得到处理后的蛋白质和药物的特征为:
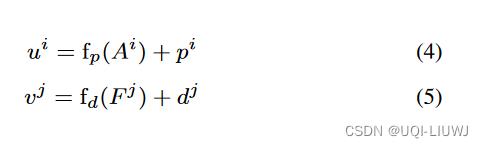
- f(A),f(F)就是经过全连接层之后的结果,p,d就是CNN的输出
2.3 桥节点
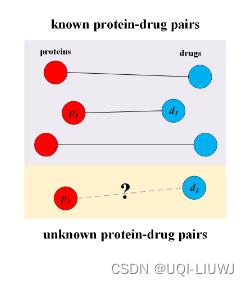
- 得到了蛋白质和药物的embedding之后,接下来的问题是,我们怎么预测蛋白质和药物之间的关系呢?(尤其是没有连边的蛋白质和药物对) 【比如P2和d2)
传统的方法是找和p2相似的蛋白质p1,和d2相似的药物d1。由于p1和d1有边,所以p2和d2有边
这里作者引入了桥节点这一概念,桥节点和每一个蛋白质节点、以及每一个药物节点都有连边
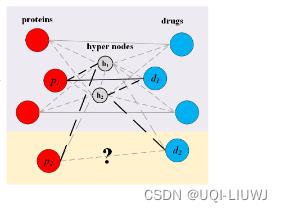
此时我们可以通过p2-h1-p1得到p2和p1之间的关系;d2-h1-d1得到d2和d1之间的关系
所以最终p2和d2之间的关系可以通过p2-h1-p1-d1-h2-d2来传递过去
2.4 GNN
- 假设桥节点集为
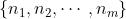 ,m是桥节点的数量
,m是桥节点的数量 - 对于任何一对蛋白质-药物对
 ,我们可以得到一个m+2个点的图,其中边权重用cos相似度表示
,我们可以得到一个m+2个点的图,其中边权重用cos相似度表示

- 希望图中边权重都是正的,所以使用ReLU(A)表示最终的图邻接矩阵
- GNN的计算图如下(这里使用了残差连接):
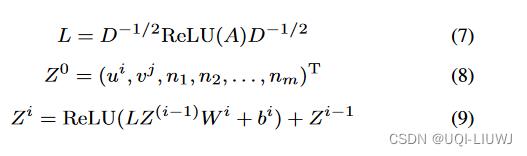
 是邻接矩阵(相似矩阵)
是邻接矩阵(相似矩阵)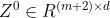 是点的embedding(d是每个点的embedding维度)
是点的embedding(d是每个点的embedding维度)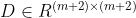 是度矩阵
是度矩阵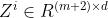 是输出
是输出
2.5 输出预测结果
-
GNN的输出是
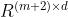 维,其中第一第二行是蛋白质和药物的embedding
维,其中第一第二行是蛋白质和药物的embedding -
这两个embedding之后做哈达玛积(逐元素乘积),然后送入全连接层
-

3 实验
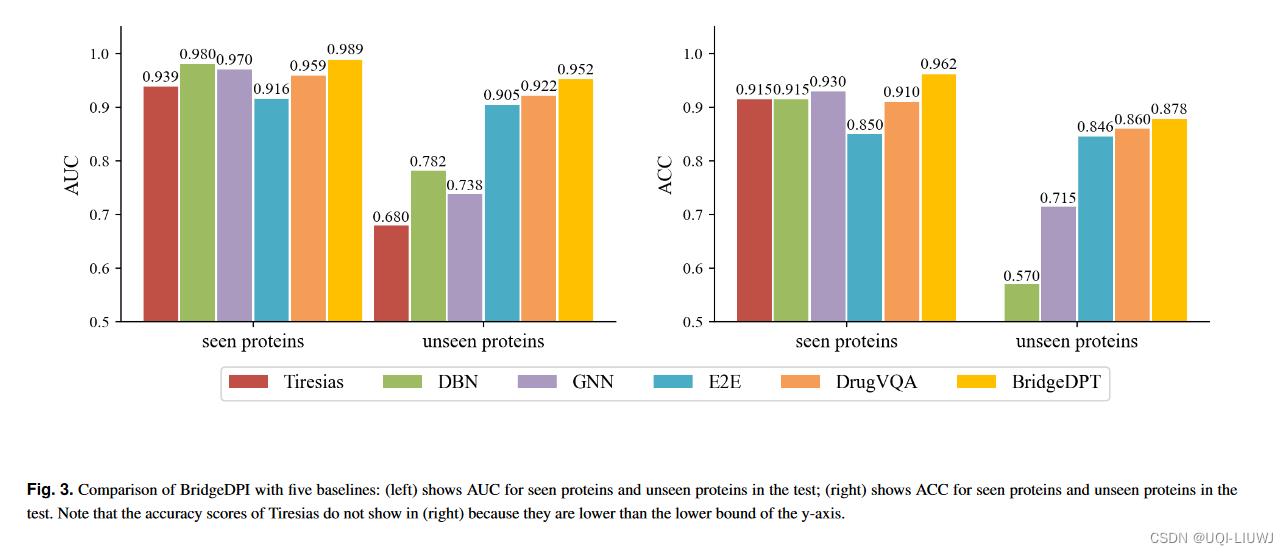
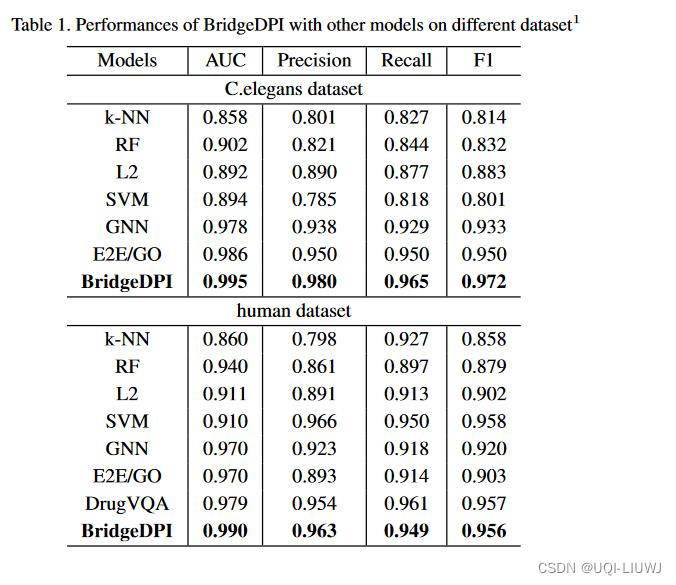
桥节点数量对结果的影响

以上是关于论文笔记:BridgeDPI: a novel Graph Neural Network for predicting drug–protein interactions的主要内容,如果未能解决你的问题,请参考以下文章