MONAI Label 安装流程及使用攻略
Posted Tina姐
tags:
篇首语:本文由小常识网(cha138.com)小编为大家整理,主要介绍了MONAI Label 安装流程及使用攻略相关的知识,希望对你有一定的参考价值。
这部分为monailabel的安装实操,分为服务端安装和客户端安装。预祝大家顺利安装。如果遇到问题,可以在交流群里探讨。
在开始前,可以把以下链接打开,
官方安装教程
monailabel github
服务器(Server)端安装
服务器电脑条件: 可以是 Ubuntu 和 Windows 操作系统, 但必须要有 GPU/CUDA。并能支持深度学习训练。因为会在服务器端调用训练和测试代码,没有GPU,带不动。
- step1: 新建一个 conda 环境
$: conda create -n monailabel python=3.9
官方推荐 python 3.7/3.8/3.9 版本。未验证其他 python 版本上的 MONAI LABEL 是否能正常工作。
- step2: 安装 torch 等相关库
$: proxychains pip install torch torchvision torchaudio
可以直接 pip, 但可能因为网络原因失败。失败多装几次。
$: pip install torch torchvision torchaudio --extra-index-url https://download.pytorch.org/whl/cu113
也可以使用镜像源安装
$: pip install torch torchvision torchaudio -i https://pypi.tuna.tsinghua.edu.cn/simple
- step3: 安装 monialabel
$: pip install monailabel
此步也可以选择从 GitHub 和 Docker 安装。需要看官方教程

如图,会安装很多依赖项
终端输入检查是否成功安装
$: monailabel

- step4: monailabel 功能介绍
从上图可以看到,monailabel 有4个大功能
可以使用 monailabel -h查看简介

- start_server: 当需要用monailabel进行分割标注时,需要在服务器端启动,就用这个命令
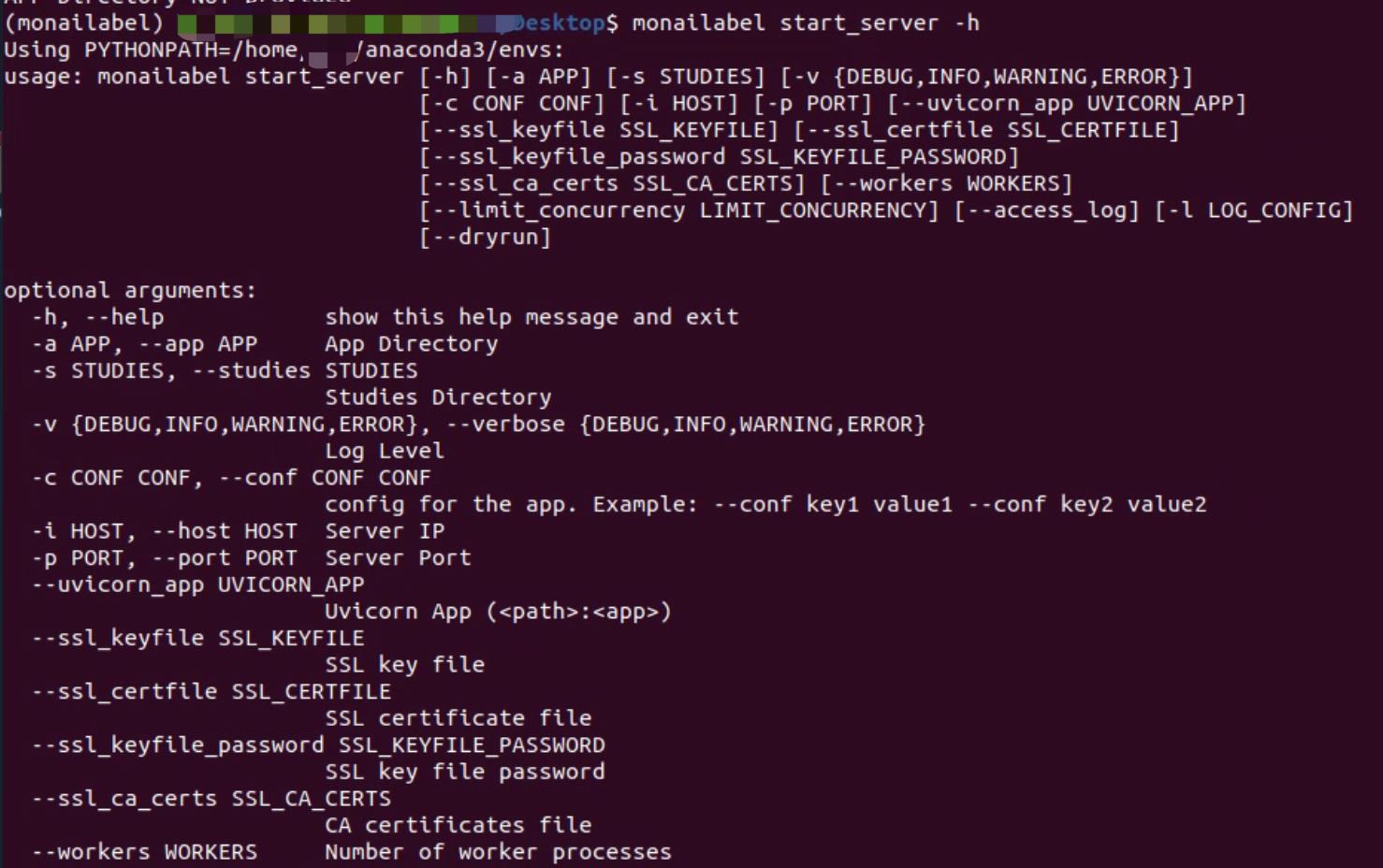
参数很多,用的时候再介绍
eg: 启动一个分割程序
$: monailabel start_server --app apps/radiology --studies datasets/Task09_Spleen/imagesTr --conf models deepedit
这里的 apps/radiology实际用的时候改为真实地址, datasets/Task09_Spleent 同理

运行的时候会下载预训练的网络,这里可以看到官方使用的模型是 dynunet
- apps: 内置一些应用,主要包括影像学应用和病理学应用,要用内置模型进行分割,需要先下载对应的模型及训练和测试程序,在这里统称为 app。

一般安装monailabel后,这些应用都是自动安装的。如果没有,使用下面命令安装
$: monailabel apps --download --name radiology --output apps
这个app很关键,如果用于医学影像数据(MRI,CT等)的分割,安装 radiology app, 病理数据安装 pathology app.
- datasets: 用于下载官方提供的一些数据示例。

eg: 我们使用它下载官方脾脏数据:
$: monailabel datasets -d -n Task09_Spleen -o datasets
或
$: monailabel datasets --download --name Task09_Spleen --output datasets
该数据集一共有1.5G,下载不下来的,可以私信我发给您。
- plugins: 列举或可以通过它下载一些客户端交互插件。如 cvat,dsa, ohif, qupath, slicer。

到此,服务端的安装结束。

客户端(Client)安装
何为客户端?简单讲,就是我们要在哪台电脑上安装标注软件进行分割。比如可以在你的笔记本上,也可以在你的服务器上标注,此时你的笔记本或者服务器就是客户端。
你也可以将服务端和客户端安装在同一台电脑上,也可以安装在不同的电脑上。同一台电脑之间通信肯定比其他电脑快。当然如果想随时随地都可以进行标注,安装在笔记本上也是一个不错的选择,但是速度怎么样还待考证。
目前 monailabel 支持/合作的标注软件有:
- 3D Slicer: 用于影像数据标注。⭐️⭐️⭐️⭐️⭐️
- OHIF: 用于影像数据标注。
- QuPath: 用于病理数据标注。
- Digital Slide Archive (DSA):用于病理数据标注。
选择适合你的标注软件进行安装,并且将 monailabel 配置到标注软件里。详细教程参考官方插件安装说明
因为我是做影像数据的,所以下面以 3D slicer为例,进行安装演示。
- step1: 3D slicer 下载&安装
3D slicer下载地址

官方推荐下载 Preview version 版本进行下载安装
- step2: 打开 3D slicer,配置 MONAI Label
打开软件,然后通过下面路径找到 MONAI Label 安装它
go to View -> Extension Manager -> Active Learning -> MONAI Label

安装后重启3D slicer
重启之后在工具栏可以看到 monai 图标。
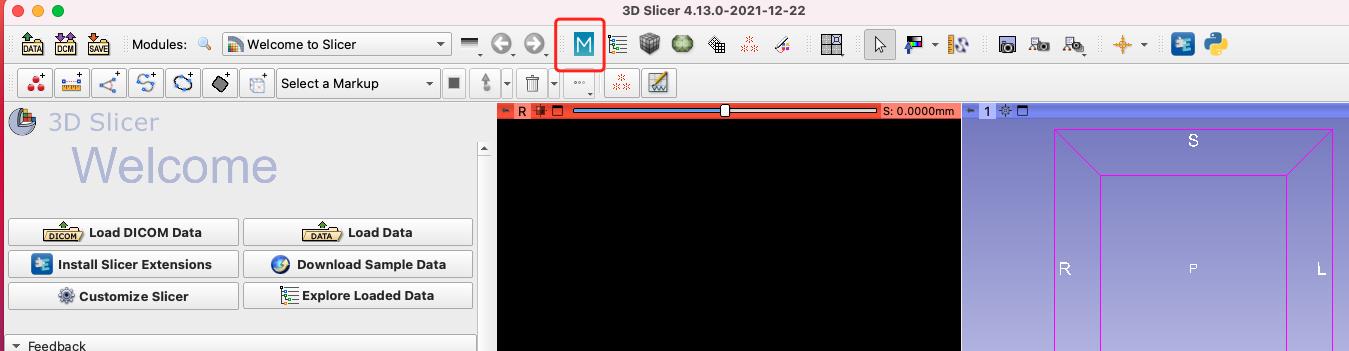
如果没有,可以通过点击 modules 旁边的放大镜,搜索 MONAILabel, 然后点击 switch to mudule就可以使用了

要将插件更新到最新版本,您必须卸载现有的 3D Slicer 版本并再次下载并安装新的 3D Slicer Preview version版本。(其实稳定版也可以,随便)
到这一步,服务器和客户端都已经安装完毕。

标注测试
此过程主要学习客户端和服务端的交互,确保二者可以进行通信。
Case 1:服务器端和客户端在同一台电脑
- step1:服务器端启动server
$: monailabel start_server --app apps/radiology --studies datasets/Task09_Spleen/imagesTr --conf models deepedit
这里的 apps/radiology实际用的时候改为真实地址,datasets/Task09_Spleent同理.
运行时,会看到它会去github下载预训练好的模型,这一过程如果没有科学&上网可能会断掉。

解决办法:
1.采用代理
2.去它给的地址下载下来,放在文件夹下(上图中,from后面的是模型地址,from前面是下载后放的地址),它就不会重复下载了。
服务端运行好后,会在最后给一个网址及端口号,把它复制到3D slicer里面。测试能否连接成功。

Case 2:服务器端和客户端不在同一台电脑
这和case1唯一不同的是,不能直接复制服务端给的网址(http://0.0.0.0:8000), 而要改为服务端真实的ip地址(eg: http://10.23.4.1:8000)
ip的获取在终端可用 ifconfig 命令查到。
当然测试的时候也是遇到了很多bug:
- train时提示内存不足
(out of memory)
出现这个原因是我发现他用的GPU刚好是一块最小的GPU,把它切换成正常可用的GPU就行了。
只需要在服务端启动server时指定显卡即可。
CUDA_VISIBLE_DEVICES=x monailabel start_server --app apps/radiology --studies datasets/Task09_Spleen/imagesTr --conf models deepedit
x=你的GPU卡号,如 x=0或1或2,也可设置多块,x=0,1, 也可以等于全部 x=all
文章持续更新,可以关注微信公众号【医学图像人工智能实战营】获取最新动态,一个关注于医学图像处理领域前沿科技的公众号。坚持已实践为主,手把手带你做项目,打比赛,写论文。凡原创文章皆提供理论讲解,实验代码,实验数据。只有实践才能成长的更快,关注我们,一起学习进步~
我是Tina, 我们下篇博客见~
白天工作晚上写文,呕心沥血
觉得写的不错的话最后,求点赞,评论,收藏。或者一键三连

以上是关于MONAI Label 安装流程及使用攻略的主要内容,如果未能解决你的问题,请参考以下文章