使用流行的遗传算法python库是哪个
Posted
tags:
篇首语:本文由小常识网(cha138.com)小编为大家整理,主要介绍了使用流行的遗传算法python库是哪个相关的知识,希望对你有一定的参考价值。
参考技术A建议使用由华南农业大学、暨南大学、华南理工大学高校硕博学生联合团队推出的Python高性能遗传和进化算法工具箱:Geatpy。它是目前进化计算领域与platemo、matlab遗传算法工具箱等有相当的权威和影响力的高性能实用型进化算法工具箱,而其效率和易用性居于领先地位。
目前已得到多所高校研究生实验室以及企业采用,为相关领域的研究和应用注入了全新的活力。
它支持GA、DE、ES等进化算法,支持单目标、多目标进化优化、复杂约束优化等问题的求解,提供丰富的遗传算法和多目标进化优化算法模板,采用高性能的C内核和mkl矩阵运算,提供功能强大的开源进化算法框架,尤其适合数学建模和研究进化算法的研究生们。
官网:Geatpy
多目标优化求解案例:
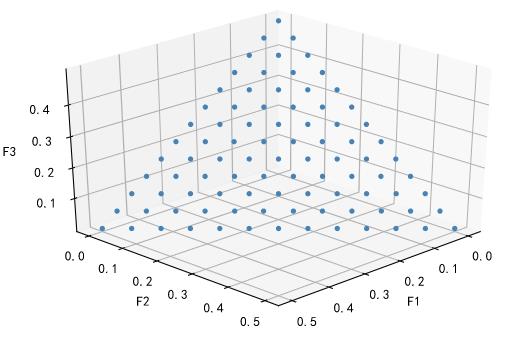
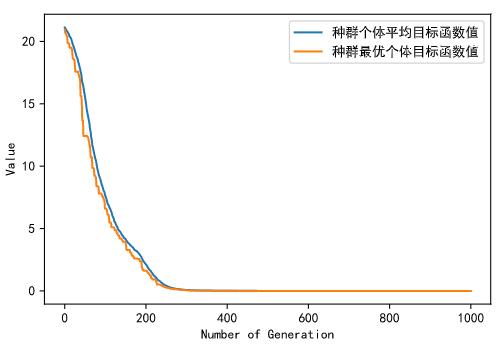
复杂约束单目标优化求解样例:
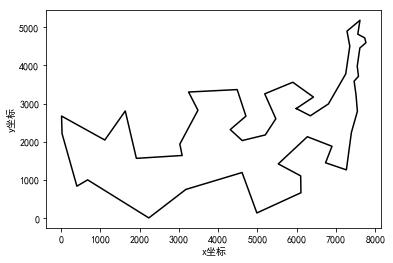
旅行商问题求解样例:
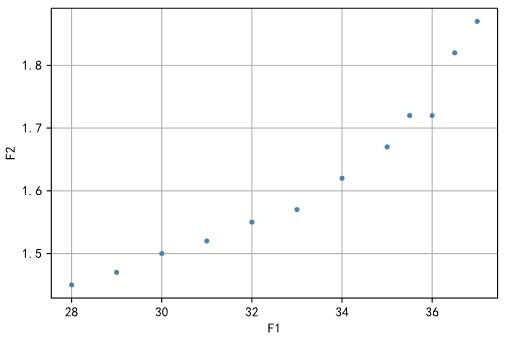
多目标背包问题求解样例:
使用方法:
第一步:实例化一个问题类把待优化的问题写在里面。
第二步:编写执行脚本调用遗传或其他进化算法模板,完成问题的求解。
官网教程:Geatpy教程
python有没有简单的遗传算法库
首先遗传算法是一种优化算法,通过模拟基因的优胜劣汰,进行计算(具体的算法思路什么的就不赘述了)。大致过程分为初始化编码、个体评价、选择,交叉,变异。
以目标式子 y = 10 * sin(5x) + 7 * cos(4x)为例,计算其最大值
首先是初始化,包括具体要计算的式子、种群数量、染色体长度、交配概率、变异概率等。并且要对基因序列进行初始化
[python] view plain copy
pop_size = 500 # 种群数量
max_value = 10 # 基因中允许出现的最大值
chrom_length = 10 # 染色体长度
pc = 0.6 # 交配概率
pm = 0.01 # 变异概率
results = [[]] # 存储每一代的最优解,N个二元组
fit_value = [] # 个体适应度
fit_mean = [] # 平均适应度
pop = geneEncoding(pop_size, chrom_length)
其中genEncodeing是自定义的一个简单随机生成序列的函数,具体实现如下
[python] view plain copy
def geneEncoding(pop_size, chrom_length):
pop = [[]]
for i in range(pop_size):
temp = []
for j in range(chrom_length):
temp.append(random.randint(0, 1))
pop.append(temp)
return pop[1:]
编码完成之后就是要进行个体评价,个体评价主要是计算各个编码出来的list的值以及对应带入目标式子的值。其实编码出来的就是一堆2进制list。这些2进制list每个都代表了一个数。其值的计算方式为转换为10进制,然后除以2的序列长度次方减一,也就是全一list的十进制减一。根据这个规则就能计算出所有list的值和带入要计算式子中的值,代码如下
[python] view plain copy
# 0.0 coding:utf-8 0.0
# 解码并计算值
import math
def decodechrom(pop, chrom_length):
temp = []
for i in range(len(pop)):
t = 0
for j in range(chrom_length):
t += pop[i][j] * (math.pow(2, j))
temp.append(t)
return temp
def calobjValue(pop, chrom_length, max_value):
temp1 = []
obj_value = []
temp1 = decodechrom(pop, chrom_length)
for i in range(len(temp1)):
x = temp1[i] * max_value / (math.pow(2, chrom_length) - 1)
obj_value.append(10 * math.sin(5 * x) + 7 * math.cos(4 * x))
return obj_value
有了具体的值和对应的基因序列,然后进行一次淘汰,目的是淘汰掉一些不可能的坏值。这里由于是计算最大值,于是就淘汰负值就好了
[python] view plain copy
# 0.0 coding:utf-8 0.0
# 淘汰(去除负值)
def calfitValue(obj_value):
fit_value = []
c_min = 0
for i in range(len(obj_value)):
if(obj_value[i] + c_min > 0):
temp = c_min + obj_value[i]
else:
temp = 0.0
fit_value.append(temp)
return fit_value
然后就是进行选择,这是整个遗传算法最核心的部分。选择实际上模拟生物遗传进化的优胜劣汰,让优秀的个体尽可能存活,让差的个体尽可能的淘汰。个体的好坏是取决于个体适应度。个体适应度越高,越容易被留下,个体适应度越低越容易被淘汰。具体的代码如下
[python] view plain copy
# 0.0 coding:utf-8 0.0
# 选择
import random
def sum(fit_value):
total = 0
for i in range(len(fit_value)):
total += fit_value[i]
return total
def cumsum(fit_value):
for i in range(len(fit_value)-2, -1, -1):
t = 0
j = 0
while(j <= i):
t += fit_value[j]
j += 1
fit_value[i] = t
fit_value[len(fit_value)-1] = 1
def selection(pop, fit_value):
newfit_value = []
# 适应度总和
total_fit = sum(fit_value)
for i in range(len(fit_value)):
newfit_value.append(fit_value[i] / total_fit)
# 计算累计概率
cumsum(newfit_value)
ms = []
pop_len = len(pop)
for i in range(pop_len):
ms.append(random.random())
ms.sort()
fitin = 0
newin = 0
newpop = pop
# 转轮盘选择法
while newin < pop_len:
if(ms[newin] < newfit_value[fitin]):
newpop[newin] = pop[fitin]
newin = newin + 1
else:
fitin = fitin + 1
pop = newpop
以上代码主要进行了3个操作,首先是计算个体适应度总和,然后在计算各自的累积适应度。这两步都好理解,主要是第三步,转轮盘选择法。这一步首先是生成基因总数个0-1的小数,然后分别和各个基因的累积个体适应度进行比较。如果累积个体适应度大于随机数则进行保留,否则就淘汰。这一块的核心思想在于:一个基因的个体适应度越高,他所占据的累计适应度空隙就越大,也就是说他越容易被保留下来。选择完后就是进行交配和变异,这个两个步骤很好理解。就是对基因序列进行改变,只不过改变的方式不一样
交配:
[python] view plain copy
# 0.0 coding:utf-8 0.0
# 交配
import random
def crossover(pop, pc):
pop_len = len(pop)
for i in range(pop_len - 1):
if(random.random() < pc):
cpoint = random.randint(0,len(pop[0]))
temp1 = []
temp2 = []
temp1.extend(pop[i][0:cpoint])
temp1.extend(pop[i+1][cpoint:len(pop[i])])
temp2.extend(pop[i+1][0:cpoint])
temp2.extend(pop[i][cpoint:len(pop[i])])
pop[i] = temp1
pop[i+1] = temp2
变异:
[python] view plain copy
# 0.0 coding:utf-8 0.0
# 基因突变
import random
def mutation(pop, pm):
px = len(pop)
py = len(pop[0])
for i in range(px):
if(random.random() < pm):
mpoint = random.randint(0, py-1)
if(pop[i][mpoint] == 1):
pop[i][mpoint] = 0
else:
pop[i][mpoint] = 1
整个遗传算法的实现完成了,总的调用入口代码如下
[python] view plain copy
# 0.0 coding:utf-8 0.0
import matplotlib.pyplot as plt
import math
from calobjValue import calobjValue
from calfitValue import calfitValue
from selection import selection
from crossover import crossover
from mutation import mutation
from best import best
from geneEncoding import geneEncoding
print 'y = 10 * math.sin(5 * x) + 7 * math.cos(4 * x)'
# 计算2进制序列代表的数值
def b2d(b, max_value, chrom_length):
t = 0
for j in range(len(b)):
t += b[j] * (math.pow(2, j))
t = t * max_value / (math.pow(2, chrom_length) - 1)
return t
pop_size = 500 # 种群数量
max_value = 10 # 基因中允许出现的最大值
chrom_length = 10 # 染色体长度
pc = 0.6 # 交配概率
pm = 0.01 # 变异概率
results = [[]] # 存储每一代的最优解,N个二元组
fit_value = [] # 个体适应度
fit_mean = [] # 平均适应度
# pop = [[0, 1, 0, 1, 0, 1, 0, 1, 0, 1] for i in range(pop_size)]
pop = geneEncoding(pop_size, chrom_length)
for i in range(pop_size):
obj_value = calobjValue(pop, chrom_length, max_value) # 个体评价
fit_value = calfitValue(obj_value) # 淘汰
best_individual, best_fit = best(pop, fit_value) # 第一个存储最优的解, 第二个存储最优基因
results.append([best_fit, b2d(best_individual, max_value, chrom_length)])
selection(pop, fit_value) # 新种群复制
crossover(pop, pc) # 交配
mutation(pop, pm) # 变异
results = results[1:]
results.sort()
X = []
Y = []
for i in range(500):
X.append(i)
t = results[i][0]
Y.append(t)
plt.plot(X, Y)
plt.show()
最后调用了一下matplotlib包,把500代最优解的变化趋势表现出来。
完整代码可以在github 查看
欢迎访问我的个人博客
阅读全文
https://blog.csdn.net/qq_33353186/article/details/82014986
以上是关于使用流行的遗传算法python库是哪个的主要内容,如果未能解决你的问题,请参考以下文章